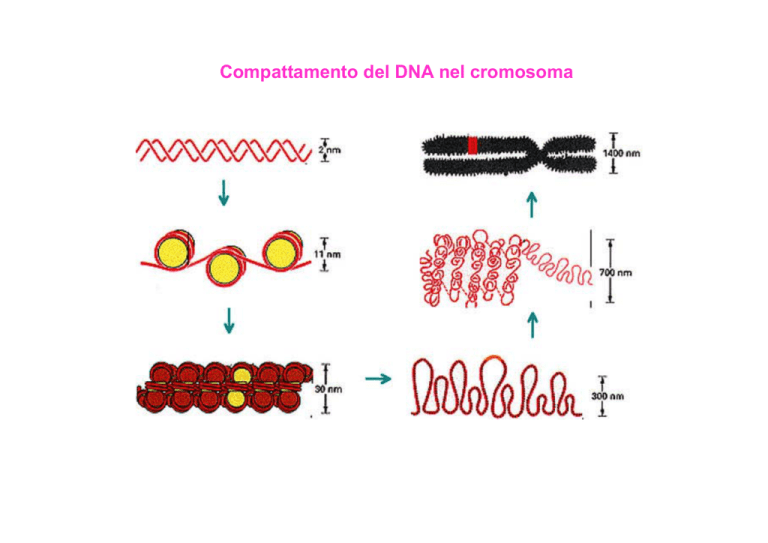
Compattamento del DNA nel cromosoma
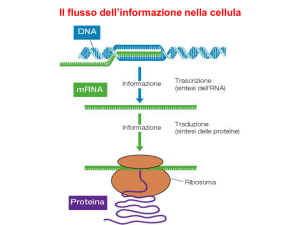
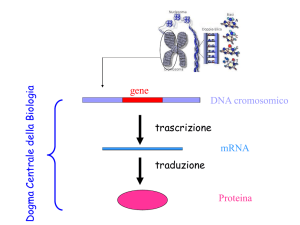
DOGMA CENTRALE DELLA BIOLOGIA
l'informazione genetica, contenuta nel nucleo nella
molecola di DNA, si trasferisce al citoplasma. I geni
del DNA vengono, nel nucleo, trascritti in una
molecola di RNA messaggero, che, passando
attraverso i pori della membrana nucleare, va nel
citoplasma dove, a livello dei ribosomi, viene
operata, tramite il codice genetico, la traduzione dal
linguaggio dei nucleotidi a quello degli aminoacidi.
Dove avviene
Il processo comincia nel nucleo (negli
eucarioti) e termina nel citoplasma o nel
reticolo endoplasmatico ruvido
ADENINA
CITOSINA
GUANINA
TIMINA
A-T 2 legami H
C-G 3 legami H
Legame
fosfodiesterico
Il DNA è il depositario dell’informazione genetica.
•
•
•
•
L’informazione genetica controlla la sequenza degli aminoacidi nelle
proteine ma il DNA non è lo stampo diretto per questa sintesi
Esistono altre molecole attraverso le quali l’informazione genetica
presente nel DNA è copiata e trasferita nel citoplasma (mRNA).
Una sequenza di DNA è definita senso se la sua sequenza è la stessa
del relativo mRNA. La sequenza posta sul filamento opposto è invece
detta antisenso. Dal momento che le RNA polimerasi lavorano
producendo una copia complementare, il filamento necessario per la
trascrizione è l'antisenso.
Altre molecole di RNA accessorie sono poi indispensabili per il
processo di sintesi proteica (tRNA e rRNA).
TRASCRIZIONE
La trascrizione è il processo mediante il quale le informazioni contenute nel DNA vengono trascritte
enzimaticamente in una molecola complementare di RNA.
La trascrizione avviene grazie a particolari enzimi detti genericamente RNA polimerasi. Tali enzimi
utilizzano nucleotidi trifosfati (ovvero nucleotidi con tre gruppi fosfato) per formare l'RNA. Durante il
processo, dai nucleosidi trifosfati vengono rimossi due gruppi fosfato per formare un legame covalente tra
un nucleotide e quello successivo.
La RNA polimerasi si lega al DNA solo presso particolari sequenze, dette promotori, che non sono
trascritte. Dal promotore iniziano a inserirsi i nucleosidi trifosfato per formare una sequenza di nucleotidi.
La trascrizione genera tre classi principali trascritti:
RNA messaggero (mRNA)
RNA transfer (tRNA)
RNA ribosomiale (rRNA)
Soltanto le molecole di mRNA vengono tradotte in proteine.
La trascrizione è un processo che presenta caratteristiche simili in procarioti ed eucarioti:
il DNA si denatura e la RNA polimerasi catalizza la sintesi di una molecola di RNA in direzione 5’ - 3’.
Soltanto una delle due eliche di DNA viene trascritta nella molecola di RNA a singola elica. Sequenze
promotrici e terminatrici stabiliscono rispettivamente dove debba cominciare e finire la trascrizione.
In Escherichia coli la sola RNA polimerasi trascrive tutte le diverse classi di RNA della cellula e cioè
l’mRNA, il tRNA e l’rRNA.
Nei nuclei delle cellule eucariotiche esistono tre RNA polimerasi con funzioni diverse:
la RNA polimerasi I trascrive tre dei quattro rRNA
la RNA polimerasi II trascrive gli mRNA e alcuni snRNA
la RNA polimerasi III trascrive il quarto rRNA, i tRNA e i rimanenti snRNA.
L’inizio della trascrizione da parte di questi enzimi è mediata da fattori trascrizionali che sono specifici
per ciascuna polimerasi e che riconoscono sequenze promotrici appropriate.
La RNA polimerasi non ha soltanto il compito di allungare la catena di RNA ma assolve differenti
funzioni. Le principali sono:
•Localizzare il promotore.
•Svolgere la doppia elica “a valle”.
•Riconoscere una base dalla catena stampo ed aggiungere una base alla catena di RNA.
•Richiudere il doppio filamento “a monte”
•Riconoscere il terminatore per distaccarsi dalla catena di DNA.
Esistono delle differenze sia strutturali che funzionali tra le RNA polimerasi batteriche ed eucariotiche.
La polimerasi batterica è un enzima costituito da cinque subunità che vengono indicate con le lettere
greche alpha (α), beta (β) e sigma (σ).
l fattore sigma è “riciclabile” nel senso che una volta che ha riconosciuto e si è legato al promotore avvia
la polimerizzazione di circa 6-9 basi e poi si distacca dal complesso enzimatico che procede con la sintesi
di RNA. Molto probabilmente il fattore sigma distaccatosi può essere utilizzato da altri complessi α2ββ'.
Le RNA polimerasi eucariotiche sono strutturalmente più complesse rispetto alle RNA polimerasi
procariotiche.
Il tipo di RNA polimerasi eucariotica più studiato è la II che, per certi versi, serve da modello per
comprendere il funzionamento delle altre. La RNA polimerasi II è formata da 12 subunità.
TRASCRIZIONE PROCARIOTI
l'RNA polimerasi si aggancia in un punto qualsiasi del DNA stampo, formando un complesso chiuso, e inizia
a dirigersi verso 5' . Durante il tragitto la polimerasi può incontrare una particolare sequenza nucleotidica che
prende il nome di promotore che “segnala” all'enzima di iniziare la trascrizione; se ciò non avviene è molto
probabile che la polimerasi si stacchi dal DNA e si agganci in un altro punto sempre casuale. A livello del
promotore si forma il cosiddetto complesso aperto.
Solitamente il promotore si trova subito a monte del sito di inizio
Una volta che la proteina ha riconosciuto il sito di attacco mette in atto una serie di reazioni enzimatiche che
portano alla denaturazione della doppia elica per un segmento di circa 12-16 basi formando il già menzionato
complesso aperto. L'apertura della doppia elica può essere vista al microscopio elettronico e, per via della
caratteristica forma che assume viene, chiamata bolla di trascrizione.
Subito dopo l'aggancio della polimerasi i primi nucleotidi vengono reclutati per essere inseriti seguendo
l'ordine 5' → 3'.
A livello della bolla di trascrizione l'enzima crea un ibrido DNA-RNA.
La catena viene allungata e il fattore sigma viene allontanato poiché può essere “riciclato”.
Ci sono numerose proteine che legandosi ai promotori ne favoriscono o ne reprimono l’associazione con la
RNA polimerasi.
PROTEINA CAP (proteina attivatrice dei geni da catabolita) che viene sintetizzata quando i batteri crescono
in un ambiente povero di glucosio. Tale proteina si lega ai promotori di geni che codificano per enzimi implicati
nel catabolismo di substrati alternativi, favorendone la trascrizione
Il PROMOTORE possiede alcuni particolari caratteristiche:
Presenta solitamente una sequenza di inizio che, in genere, è CAT.
A -10 basi a monte del punto di inizio possiede un esamero, o sequenza consensus TATAAT
A -35 basi rispetto al punto di inizio possiede una ulteriore sequenza consensus TTGACA
ALLUNGAMENTO
Il fattore sigma si dissocia o, comunque, non partecipa più agli eventi di
polimerizzazione, non appena l'RNA polimerasi sintetizza pochi nucleotidi, di solito
meno di una dozzina.
La bolla di DNA, ovvero la struttura derivante dalla temporanea denaturazione
operata dall'RNA polimerasi, avanza in direzione 3' → 5' e in questo modo, grazie al
complesso della RNA polimerasi, vengono inseriti nucleotidi tramite legami
fosfodiesterici tra gli zuccheri adiacenti allungando la catena di RNA nascente.
Il DNA è fortemente spiralizzato per cui la RNA polimerasi deve trovare il modo per
poter rilassare la molecola al fine di leggere dal filamento stampo e continuare la
trascrizione. Il modello più accreditato che spiega l'avanzamento del complesso
DNA/Polimerasi/RNA è quello secondo il quale la molecola di DNA si svolge di
fronte alla polimerasi per poi riappaiarsi immediatamente dopo che è stato letto il
nucleotide e, di conseguenza, trascritto.
Interviene un enzima appartenente alla classe delle topoisomerasi che spezza uno
o due filamenti del DNA rendendo possibile di conseguenza un rilassamento della
lunga molecola.
TERMINAZIONE
Terminazioni rho dipendenti.
Terminazioni rho indipendenti od intrinseche.
Le Terminazioni rho-dipendenti
La proteina rho è strutturalmente un esamero di circa 275KD.
In realtà la proteina rho riconosce una specifica sequenza nell'RNA trascritto e grazie alla presenza di
questo gruppo di basi si lega ad essa e prosegue in direzione opposta rispetto alla direzione di sintesi della
molecola di mRNA. In altre parole, la proteina rho, una volta riconosciuta la sequenza tipica, si lega
all'RNA e scorrre fino a giungere alla RNA-polimerasi.
Le terminazioni rho-indipendenti o terminazioni intrinseche non hanno bisogno di alcuna struttura per
interrompere la trascrizione. Il DNA contiene al proprio interno delle sequenze che istruiscono l'RNA
polimerasi a fermarsi; questi segmenti di informazione genica hanno la particolarità di essere
specularmente uguali. Prendiamo ad esempio questa sequenza di DNA ed il suo trascritto:
AGTGTTAGTAACACT (DNA)
(trascrizione)
UCACAAUCAUUGUGA (RNA)
Rispetto alla G del DNA ed alla C dell'RNA (ambedue evidenziate nella sequenza di sopra), le due catene,
se lette dalla base marcata verso sinistra o dalla base marcata verso destra sono complementari. Questo
vuol dire che, mentre la RNA polimerasi sintetizza l'RNA, questa molecola si può “auto-appaiare” formando
una forcina caratteristica. La forcina pare bloccare la terminazione e, di conseguenza, sembra poter
coadiuvare il rilascio dell'RNA già trascritto.
MATURAZIONE rRNA e tRNA PROCARIOTI
Nei procarioti l’mRNA subisce poche modificazioni dopo la sintesi, mentre i tRNA gli rRNA sono
sottoposti a tagli e modificazioni
rRNA e tRNA (RNA ribosomiale e
transfer) nei procarioti provengono
da tratti di DNA ripetuti una decina
di volte. Nel trascritto finale
troviamo sia le sequenze degli
rRNA maturi che quelle dei tRNA,
che verranno staccati tra loro da
una RNAasi III.
Maturazione dei pre-rRNA negli eucarioti
MATURAZIONE mRNA DEGLI EUCARIOTI
La maturazione degli mRNA (RNA messaggero) è un processo esclusivo degli eucarioti:
Nei procarioti invece l'estremità 5' dell'mRNA viene legata subito dai ribosomi, per cui la
traduzione avviene assieme alla trascrizione
Capping
Poliadenilazione
Metilazione
Splicing
Negli eucarioti l'mRNA appena sintetizzato nel nucleo risulta più grande di quello che
servirà alla fine nel citoplasma per la traduzione (sintesi delle proteine).
.
Il capping consiste nell'aggiunta di un 'cappuccio' all'estremità 5' di una 7-metilguanosina legata a tre
gruppi fosfato. Tale cap dovrebbe avere la funzione di proteggere l'estremità 5' e di favorire il legame del
ribosoma per l'inizio della traduzione. Rende inoltre più stabile l’mRNA
Negli eucarioti troviamo tratti di mRNA che saranno mantenuti e tratti che saranno eliminati,
rispettivamente introni ed esoni. Lo splicing consiste appunto nella rimozione degli esoni e ricucitura tra
loro degli introni. Questo è possibile grazie agli snRNP (small nuclear ribonucleoprotein particles)
costituite da snRNA e proteine, che costituiscono un complesso (detto spliceosoma) localizzati in punti
ben definiti del nucleo. Esistono casi di splicing alternativo, in cui dallo stesso mRNA iniziale possono
originare differenti splicing per rimozione di sequenze introniche ottenendo RNA maturi finali differenti.
La poliadenilazione è il legame covalente di un filamento di adenina ad una molecola di RNA messaggero.
La coda di poli-A all'estremità 3' terminale aiuta la stabilità dell‘mRNA, proteggendolo dalle esonucleasi. La
poliadenilazione è importante anche per la terminazione della trascrizione, il trasporto dell‘mRNA fuori dal
nucleo cellulare e la traduzione (o sintesi proteica). Anche alcuni mRNA procariotici sono poliadenilati,
nonostante la funzione di taglio della poli-A sia differente da quella degli organismi eucarioti.
Dopo che le trascrizione è terminata, vengono aggiunti al sito di taglio dagli 80 ai 250 residui di adenosina
Questa reazione è catalizzata dalla poliadenilato polimerasi (o poli-A polimerasi).
Splicing di un trascritto primario. L’RNA messaggero viene prodotto nel nucleo cellulare, sul modello di
un filamento di DNA, per mezzo di un enzima denominato RNA polimerasi DNA dipendente. Sempre nel
nucleo, l’mRNA primitivo così costituitosi viene sottoposto ad un rimaneggiamento, detto splicing, che
consiste essenzialmente nell’eliminazione degli introni, privi di funzione codificante. L’mRNA maturo viene
trasferito nel citoplasma e viene letto dai ribosomi.
CODICE GENETICO
UNIVERSALE
DEGENERATO (nucleotide in terza
posizione)
1 O PIU’ CODONI
64 CODONI
AMMINOACIDO
20 AMMINOACIDI
SINTESI PROTEICA
mRNA
AMMINOACIDI
ribosoma, composto da RNA ribosomiale (rRNA) e proteine ribosomiali.
RNA messaggero (mRNA).
RNA transfer (tRNA), una corta catena di RNA che è legata
covalentemente ad un dato amminoacido, costituendo un amminoacil-tRna.
Le Amminoacil tRNA sintetasi, sono enzimi che catalizzano la
formazione del legame ad alta energia tra un dato amminoacido e l‘ RNA
transfer corrispondente.
I ribosomi sono organelli immersi nel citoplasma - o ancorati al reticolo endoplasmatico - e sono le
particelle responsabili della sintesi proteica. La loro funzione è quindi quella di sintetizzare le
proteine leggendo le informazioni contenute in una catena di RNA messaggero (m-RNA).
I ribosomi sono formati da tre molecole di RNA ribosomiale e da proteine che si associano a formare
due subunità di dimensioni differenti (una più grande dell'altra).
Un ribosoma batterico ha una massa di circa 2700 kDa, un diametro di circa 20 nm ed un coefficiente di
sedimentazione di 70 S.
Esso si può suddividere in due parti o subunità, una più grande ed una più piccola:
una subunità grande di 50 S avente almeno 34 proteine (L1-L34) e due molecole di RNA (23 S e 5 S),
una subunità piccola di 30 S contenente almeno 21 proteine (S1-S21) ed un RNA di 16 S.
Il ribosoma della cellula eucariota (fatta eccezione per quelli contenuti nei mitocondri e nei cloroplasti),
invece, è più grande ed ha una massa molecolare di 4000 kDa, un diametro di 23 nm ed un coefficiente
di sedimentazione di 80 S.
Anch'esso è composto da due subunità, maggiore a 60 S e minore a 40 S:
la subunità maggiore è costituita da tre molecole di rRNA, una a 28 S, una a 5,8 S e un'ultima a 5 S.
la minore consta di una sola catena di RNA 18 S. Nel complesso le due subunità presentano inoltre più
di 80 proteine.
La loro disposizione all'interno della cellula varia a seconda del tipo di cellula ed è collegata alla
funzione di quest'ultima:
se la cellula secerne le proteine prodotte, possiede solo ribosomi attaccati al reticolo endoplasmatico
(che occupa gran parte del citosol) e alla membrana nucleare;
se la cellula immagazzina queste proteine, possiede ribosomi liberi nel citoplasma.
Le singole molecole di rRNA (tranne la 5 S) vengono sintetizzate nei nucleoli come
RNA 45 S. Il DNA contenuto nel nucleolo viene trascritto dalla RNA polimerasi I a
partire da più punti della catena di DNA, in strutture che vengono dette ad "albero
di Natale"; il tronco verrebbe rappresentato dal DNA, i rami dalle molte catene di
rRNA che vengono trascritte nello stesso momento.
L’ rRNA 45 S appena trascritto è detto pre-rRNA; in seguito a tagli, esso darà
origine a rRNA 18 S (per la subunità minore del ribosoma) e 32 S, il quale verrà
tagliato ulteriormente in 28 S e 5,8 S. Nel pre-rRNA sono presenti pseudouridine e
basi azotate metilate. La funzione di queste modifiche nelle basi si pensa sia di
evitare il taglio enzimatico, o favorire le interazioni dell'RNA interne alla catena o
con altre molecole.
Maturazione dei pre-rRNA negli eucarioti
I RIBOSOMI
Le due subunità del ribosoma si uniscono tra loro ed operano insieme per tradurre un RNA
messaggero in una catena polipeptidica durante la sintesi proteica.
Sembra che la parte più importante del ribosoma, sotto quest'aspetto, siano le molecole di RNA
ribosomiale le quali da sole sono in grado, seppur lentamente, d'operare il processo di traduzione.
Questo lascia ipotizzare che la componente proteica agisca in modo tale da potenziare e velocizzare
l'azione dell'RNA ribosomiale.
Prima che le subunità si uniscano insieme per incominciare a tradurre la molecola di m-RNA, c’è
bisogno che il complesso di pre-inizio si leghi alla subunità inferiore; a questo punto il ribosoma si può
collegare alla molecola dell’RNA messaggero, ed una volta che la subunità minore riconosce la
tripletta di inizio, si staccano i fattori di pre-inizio, le due subunità si collegano ed il ribosoma può
incominciare a sintetizzare la proteina.
La subunità maggiore è composta da tre siti, in ordine E (exit), P (peptidilico) ed A (amminoacilico).
La subunità minore possiede solo un sito d’attacco predisposto per l’mRNA.
Il primo tRNA si va ad inserire nel sito P, a questo punto arriva un ulteriore tRNA il quale si andrà a
posizionare nel sito A, ora il ribosoma scorre di una tripletta, portando gli amminoacidi tradotti
presente dal sito P, a legarsi nel sito A. La tripletta codificata da P a questo punto passa al sito E,
dove verrà rilasciato
Il sito A è il sito che attende l’arrivo di
nuove triplette da codificare;
Il sito P tiene salda la sequenza
amminoacidica, rilasciandola solamente
quando il sito A ha tradotto un’ulteriore
amminoacido;
Il sito E è il sito di rilascio
La struttura dei ribosomi nei
procarioti e negli eucarioti
L'RNA transfer (tRNA), è una piccola catena di RNA (di 7493 nucleotidi) che trasferisce un amminoacido specifico ad
una catena polipeptidica in crescita nel sito ribosomiale della
sintesi proteica.
Il tRNA ha un sito di attacco per l'amminoacido ed una
regione con tre basi, chiamata anticodone, che riconosce il
corrispondente codone a tre basi dell‘mRNA attraverso
l'appaiamento di basi complementari.
Ogni tipo di molecola di tRNA può legarsi ad un solo tipo di
amminoacido, ma essendo presenti nel DNA tipi diversi di
codoni che codificano per uno stesso amminoacido, più tipi
di tRNA con anticodoni differenti possono portare lo stesso
amminoacido.
ADATTATORE TRA AMMINOACIDI TRIPLETTE
ATTIVAZIONE DEGLI AMMINOACIDI
Gli amminoacidi devono essere attivati, per essere trasformati in composti a più alta
energia, per rendere energeticamente favorevole la formazione del legame peptidico
amminoacido + ATP → amminoacil-AMP + PPi
Ogni tRNA è amminocilato (o caricato) con uno specifico amminoacido dall‘
amminoacil-tRNA sintetasi. Esiste soltanto una amminoacil-tRNA sintetasi per ogni
amminoacido..
Le amminoacil-tRNA sintetasi hanno anche la funzione di correzione di errori
nell’accoppiamento tra amminoacido e tRNA
amminoacil-AMP + tRNA → amminoacil-tRNA + AMP
FASI DELLA BIOSINTESI
INIZIO: i ribosomi si legano al codone di avvio dell'mRNA, che indica il punto in cui l'mRNA comincia a codificare la
proteina. Questo codone è generalmente AUG (adenina-uracile-guanina) che codifica per la metionina.
Nei batteri invece la proteina inizia con la N-formil-metionina
Nei procarioti, il corretto legame tra il ribosoma e l'mRNA è facilitato dall'accoppiamento di una serie di basi nota come
sequenza di Shine-Dalgarno, che si trova tra 8 e 13 nucleotidi prima del codone di avvio.
Il tRNA, sia che rechi metionina o N-formil-metionina, accoppia le sue basi con quelle del codone di avvio coadiuvato da dei
fattori di inizio (IF) e si lega al sito P (peptide) del ribosoma formando un ponte tra la subunità minore e la subunità
maggiore.
La subunità maggiore forma quindi un complesso con quella minore e i fattori di inizio vengono liberati.
ALLUNGAMENTO: Un nuovo aminoacil-tRNA
complessato con il fattore di allungamento (una proteina
GTP dipendente, EF-Tu nei procarioti, αEF1 negli
eucarioti, più semplicemente Tu) entra sul sito A del
ribosoma ed accoppia le sue basi con quelle dell'mRNA.
La subunità ribosomiale maggiore possiede azione
peptidil-transferasica, grazie alla quale crea un legame
peptidico tra gli amminoacidi vicini. Appena questo
accade, l'amminoacido sul sito P si stacca dal suo tRNA e
la catena peptidica in crescita si lega al tRNA sul sito A. Il
ribosoma quindi si muove lungo l'mRNA spostando il
peptidil-tRNA dal sito A al sito P liberando nel contempo il
tRNA vuoto. Questo processo è noto come
traslocazione.
TERMINAZIONE: Il processo continua finché il ribosoma non
incontra uno dei tre possibili codoni di arresto (stop), che sono
UAG, UAA, UGA. La crescita della proteina si interrompe ed i
fattori di rilascio (RF1, RF2, RF3 nei batteri, RF1 ed RF3 negli
eucarioti), proteine che simulano l'azione del tRNA, si legano al
sito A e liberano la proteina nel citoplasma
Fasi Della Traduzione: inizio
• Attacco delle subunità ribosomali e del primo a.a.
portato dal tRNA iniziatore all’mRNA
• Posizionamento sul primo codon da tradurre
Subunità
Fattori d’inizio
tRNA iniziatore
Fonte di Energia
PROCARIOTI
30S/ 50S
Sì
formilmetionina
GTP
EUCARIOTI
40S/60S
Sì
metionina
ATP/GTP
Fasi Della Traduzione: allungamento
Allungamento della catena polipeptidica mediante attacco di
aminoacil-tRNA e sintesi del legame peptidico.
PROCARIOTI
EUCARIOTI
30S/50S
40S/60S
Subunità
Fattori di Allungamento
tRNA
Fonte di energia
Sì
Sì
tutti
tutti
GTP
GTP
Fasi Della Traduzione: terminazione
• Avviene quando il ribosoma arriva ad un
codone di stop (UAA, UAG, UGA) che si
colloca nel sito A
• Rilascio della catena polipeptidica e rilascio
delle due subunità ribosomali
Subunità
Fattori di
terminazione
Fonte di energia
PROCARIOTI
EUCARIOTI
30S/50S
40S/60S
3
1
GTP
GTP
La fase di traduzione ha inizio quando l’RNA messaggero si
attacca al ribosoma.
Gln
G
U
Ala
U
C
G
Val
A
C
A
anticodone
codone
G
C
U
C
A
A
C
C
U
G
U
A
Gly
U
G
G
A
Successivamente su ogni codone si attaccherà il tRNA con
l’anticodone complementare, portandosi dietro un amminoacido.
Gln
G
U
Ala
U
C
G
C
U
C
A
G
A
Val
A
C
C
C
U
G
U
A
A
Gly
U
G
G
A
Successivamente
su ogni
codone
si attaccherà
il tRNA con si
La fase di traduzione
ha inizio
quando
l’RNA messaggero
l’anticodone
complementare, portandosi dietro un amminoacido.
attacca
al ribosoma.
Gln
Val
Gly
Ala
G
U
U
C
C
G
C
U
G
C
A
A
A
A
C
C
U
G
U
A
U
G
G
A
Gln
Val
Gly
Ala
G
U
U
C
C
G
A
G
C
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
Gln
Val
Gly
Ala
G
U
U
C
C
G
A
G
C
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
Gln
Val
Gly
Ala
G
U
U
C
C
G
A
G
C
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
Val
Gly
Gln
Ala
C
G
U
U
C
G
A
G
C
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
Val
Ala
Gly
Gln
C
C
G
A
G
U
U
G
C
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
L’amminoacido del primo tRNA si lega poi a quello del secondo…
Val
Ala
Gly
Gln
C
C
G
A
G
U
U
G
C
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
L’amminoacido del primo tRNA si lega poi a quello del secondo…
Val
Gly
Ala
Gln
C
C
G
A
G
U
U
G
C
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
L’amminoacido del primo tRNA si lega poi a quello del secondo…
Val
Gly
Ala
Gln
C
C
G
A
G
U
U
G
C
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
… il primo tRNA si allontana…
Val
Gly
Ala
Gln
C
C
G
A
A
G
C
U
G
U
U
C
A
A
C
C
U
G
U
A
U
G
G
A
… il primo tRNA si allontana…
Val
Gly
Ala
Gln
C
G
A
C
G
C
U
G
U
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
… il primo tRNA si allontana…
Val
Gly
Ala
Gln
C
G
A
C
G
C
U
G
U
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
… e un nuovo tRNA si attacca all’RNA messaggero
Val
Gly
Ala
Gln
C
G
A
C
G
C
U
G
U
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
… e un nuovo tRNA si attacca all’RNA messaggero
Val
Gly
Ala
Gln
C
G
C
U
G
U
U
C
A
A
C
C
U
G
U
A
A
U
G
G
A
… e un nuovo tRNA si attacca all’RNA messaggero
Val
Gly
Ala
Gln
C
A
G
G
C
U
G
U
U
C
A
A
C
C
U
G
U
A
G
U
A
… e così via.
Val
Ala
Gln
Gly
C
G
C
U
G
U
U
G
G
A
C
A
A
C
C
U
G
U
A
A
U
… e così via.
Val
Ala
Gln
Gly
C
G
C
U
G
U
U
G
G
A
C
A
A
C
C
U
G
U
A
A
U
… e così via.
Val
Ala
Gln
Gly
C
G
G
C
U
U
A
U
C
A
A
G
G
A
C
C
U
G
U
A
U
… e così via.
Val
Ala
Gln
Gly
G
G
U
C
U
U
C
C
A
A
G
G
A
C
C
U
G
U
A
A
U
… e così via.
Val
G
U
Ala
Gln
Gly
U
C
G
C
U
C
A
A
G
G
A
C
C
U
G
U
A
A
U
… e così via.
Val
Ala
Gln
Gly
C
G
C
U
C
A
A
G
G
A
C
C
U
G
U
A
A
U
… e così via.
Ala
Gln
Gly
Val
C
G
C
U
C
A
A
G
G
A
C
C
U
G
U
A
A
U
… e così via.
Ala
Gln
Gly
G
C
U
C
A
A
Val
G
G
A
C
A
U
C
C
U
G
U
A
… e così via.
Ala
Gln
Gly
G
C
U
C
A
A
Val
G
G
A
C
A
U
C
C
U
G
U
A
… e così via.
Ala
Gln
Gly
Val
G
C
U
C
A
A
G
G
A
C
A
U
C
C
U
G
U
A
Eventi post-traduzionali
Specifici segnali, contenuti nella sequenza amminoacidica delle
proteine, le dirigono alle loro destinazioni cellulari finali
La sintesi proteica inizia sui ribosomi liberi nel citoplasma.
Le proteine destinate a nucleo e mitocondri sono completate
qui e hanno segnali che permettono il legame e l’entrata
negli organelli a cui sono destinate.
Le proteine destinate a: RE, Golgi, lisosomi e all’esterno
della cellula completano la loro sintesi su ribosomi attaccati
alla superficie del RE.
Entrano, quindi nel RE grazie all’interazione di una sequenza
segnale idrofobica con un canale della membrana.
Il reticolo endoplasmatico (RE) è un complesso organulo, con una
struttura cellulare costituita da regioni citoplasmatiche delimitate da
membrane che possono avere la forma di cisterne o tubuli. Lo spazio
interno che si identifica tra le pieghe del reticolo è detto lume ricco di
enzimi che catalizzano diverse reazioni chimiche.
Vi sono due tipologie di RE differenti per morfologia e funzione: il reticolo
endoplasmatico rugoso (RER), detto così per la presenza di numerosi ribosomi
che gli danno, nella visione al microscopio, un aspetto ruvido, e quello liscio (REL),
che evidentemente non presenta i ribosomi sulla superficie.
Dentro ai compartimenti del reticolo endoplasmatico si svolge un'intensa attività di
reazioni chimiche che elaborano continuamente materiali, alcuni dei quali vengono
ivi depositati, altri trasportati in altre parti della cellula, altri ancora avviati verso
l'esterno.
L'apparato del Golgi è un organulo di composizione lipo-proteica
Esso è formato da cisterne membranose appiattite, impilate le une sulle altre,
deputate alla sintesi di lipidi, glicolipidi e glicoproteine (mediante l'aggiunta di residui
glucidici).
L'apparato di Golgi ha una funzione molto importante ovvero di rielaborare,
selezionare ed esportare i prodotti cellulari.
Questo organulo può interagire con altri (come il reticolo endoplasmatico rugoso)
per indirizzare ed etichettare certe vescicole contenenti prodotti cellulari verso la
loro destinazione, che può essere quello di confluire in altri organi o fondersi con la
membrana plasmatica e farne uscire il contenuto.
Le vescicole che gemmano dal Reticolo Endoplasmatico Rugoso (RER) sono
trasportate alla faccia cis dell'apparato del Golgi, dove si fondono con la sua
membrana rilasciando il loro contenuto all'interno del lume. Una volta nel lume, le
molecole vengono modificate, raggruppate e spedite verso la loro destinazione
finale
MODIFICAZIONI POST-TRADUZIONALI
Una modificazione post traduzionale è la modificazione chimica di una proteina in seguito alla sua
traduzione. Per molte proteine, si tratta dell'ultima tappa di biosintesi.
Ogni proteina è un polipeptide composto da una catena in cui possono essere incorporati (durante la
sintesi proteica vera e propria) 20 diversi amminoacidi. Molti di questi amminoacidi possono essere
modificati attraverso l'aggiunta di alcuni gruppi funzionali in grado di allargare la funzionalità
complessiva della proteina. Modificazioni tipiche, ad esempio, sono l'aggiunta di gruppi acetile, fosfato,
lipidici o glucidici.
Un ulteriore tipo di modificazione post traduzionale è la rimozione di amminoacidi dalla regione Nterminale della proteina attraverso l'azione di una proteasi.
Modificazioni come la fosforilazione, sono comuni meccanismi di attivazione ed inattivazione di proteine
ed enzimi.
Spesso nelle cellule eucariotiche, la catena peptidica subisce il taglio di alcuni frammenti che non sono
direttamente implicati nella funzione biologica. Tali frammenti sono i peptidi segnale e sono necessari
per l’ingresso della proteina nel reticolo endoplasmatico.
SINTESI INSULINA IN CELLULA PANCREATICA
CATENA A
CATENA B
30 aa
21 aa
Unite da ponti S-S
ESONE 2
ESONE 1
PREPROINSULINA
PEPTIDE SEGNALE
PROINSULINA
FORMA S-S
IN APPARATO DEL GOLGI
UN ENZIMA RIMUOVE 33aa
C
A
B
INSULINA
l‘ ormone insulina, in seguito alla formazione dei
ponti disolfuro, è tagliato due volte, con la rimozione
del cosiddetto propeptide presente nel centro della
catena. L'insulina matura consiste dunque di due
polipeptidi legati tra loro attraverso ponti disolfuro.
GLI ANTIBIOTICI E ALCUNE TOSSINE INTERFERISCONO CON LA
SINTESI PROTEICA
Il cloramfenicolo inibisce la sintesi proteica legandosi alla subunità 50S
ribosomiale bloccando il meccanismo di traduzione. Il cloranfenicolo si
interpone in uno spazio della subunità 50s tra il sito A e il sito P, bloccando l'
enzima peptidil-transpeptidasi, che è adibito al trasporto della catena
proteica dal sito A al sito P.
Le tetracicline inibiscono la sintesi proteica dei batteri legandosi alla
subunità 30S del ribosoma batterico in modo tale da impedire l’accesso
dell’aminoacil tRNA al sito accettare (A).
La streptomicina inibisce la sintesi proteica legandosi alla subunità 16S del
rRNA batterico interferendo con la subunità 30S e impedendole di interagire
col formil-metionil-tRNA
Ciò quindi previene l'inizio della sintesi proteica, impedendo al batterio di
espletare le proprie attività e causandone la morte
La cicloesimide blocca la peptidil transferasi dei ribosomi eucariotici
Tossina ricina e difterica (natura proteica) inattivano rispettivamente la
subunità 60s dei ribosomi eucariotici e il fattore di allungamento EF-2.
La regolazione dell’attività dei geni
La trascrizione di un gene e la sintesi di una proteina specifica è
soggetta a controllo; essa avviene:
• secondo le necessità della cellula e
• in base alle disponibilità dell’ambiente
- In assenza del substrato, è inutile sintetizzare gli enzimi che
“lavorano” quel substrato.
- Quando un metabolita sia presente nella cellula in grande
quantità, è dannoso continuare a produrlo senza necessità
operone
Come la cellula regola
l’espressione dei geni secondo
le necessità dell’organismo e in
risposta alla situazione
dell’ambiente
In biologia si definisce operone un insieme di geni che vengono regolati in modo strettamente
coordinato.
L'organizzazione dei geni in operoni è un elemento fondamentale nella regolazione genica dei
procarioti
gli operoni contengono infatti, oltre ai geni che devono essere trascritti, sequenze particolari,
denominate siti di controllo, che con vari meccanismi regolano l'espressione dei geni dell'intero
operone
Un operone contiene sempre i seguenti elementi:
uno o, di solito, più geni strutturali, ovvero geni che codificano per determinati enzimi o proteine
necessari alla cellula.
un promotore, situato a monte dei geni, ovvero una sequenza di DNA che, legandosi all‘ RNA
polimerasi, permette l'inizio della trascrizione. L'RNA polimerasi ha infatti bisogno di riconoscere la
sequenza del promotore per iniziare il processo.
un operatore, un frammento di DNA, che può essere situato a monte, a valle o anche lontano dal
promotore, che regola l'espressione dei geni strutturali. L'operatore svolge questa funzione
interagendo con una specifica proteina chiamata proteina repressore o proteina attivatore, a
seconda che, appunto, impedisca o stimoli l'espressione.
un gene regolatore, che codifica per la proteina regolatrice. Questo gene, tuttavia, non viene
normalmente considerato parte integrante dell'operone, in quanto in alcuni casi può essere
dislocato in un punto del genoma anche molto lontano dall'operone stesso.
CONTROLLO ESPRESSIONE GENICA
-REGOLAZIONE LENTA (CONTROLLO TRASCRIZIONALE): regola il
livello endocellulare di una proteina e quindi l’efficienza dei
meccanismi ad essa correlati.
-REGOLAZIONE RAPIDA (ENZIMATICA): controllo mediato da
modificazioni chimiche dell’enzima (fosforilazione) o dall’interazione
con una o più molecole prodotte direttamente o indirettamente dalla
proteina stessa
REGOLAZIONE DELLA TRASCRIZIONE (procarioti)
-GENE STRUTTURALE: codifica per le proteine enzimatiche
Ogni gene strutturale è preceduta da DNA con funzione di regolazione:
-PROMOTORE: regione per il legame dell’RNA polimerasi
-OPERATORE: regione per il legame di molecole di repressore
-GENE REGOLATORE: codificano per proteine che “gestiscono” il
programma (repressori o induttori della sintesi di proteine enzimatiche)
presente anche a notevole distanza dai geni controllati
OPERONE
gruppo di geni che sintetizzano enzimi coinvolti in una catena di
reazioni controllati dagli stessi geni regolatori
Il controllo genico degli operoni è un tipo di regolazione che permette agli organismi di regolare l'espressione a
seconda delle condizioni dell'ambiente in cui vivono. La regolazione può essere positiva o negativa.
La regolazione negativa coinvolge il legame di una proteina repressore all'operatore per impedire la trascrizione.
Gli operoni sottoposti a regolazione negativa si distinguono in operoni inducibili o repressibili.
Negli operoni negativi inducibili, una proteina repressore si trova legata, in condizioni normali, all'operatore,
impedendo così la trascrizione dei geni dell'operone. Se però nella cellula è presente una particolare molecola,
detta induttrice, essa si lega alla proteina repressore, cambiandone la conformazione e rendendola incapace di
legare l'operatore e permettendo così la trascrizione.
Negli operoni negativi repressibili, la trascrizione dei geni dell'operoni avviene regolarmente in condizioni
normali. La proteina repressore, infatti, pur essendo attivamente prodotta dal gene regolatore, è incapace di
legarsi all'operatore nella sua conformazione normale. Tuttavia, alcune molecole chiamate corepressori possono
legarsi alla proteina repressore, e cambiarne la conformazione in modo da renderla capace di legarsi
all'operatore, e di impedire così la trascrizione.
Gli operoni possono anche essere sottoposti a regolazione positiva. In questo caso una proteina attivatore si
lega all'operatore stimolando la trascrizione. Anche gli operoni sottoposti a controllo positivo si suddividono in
operoni inducibili o repressibili.
Negli operoni positivi inducibili, la proteina attivatore è normalmente incapace di legarsi all'operatore. Certe
molecole, tuttavia, possono legarsi alla proteina attivatore e cambiare la sua conformazione in modo da renderla
capace di legarsi al DNA e incentivare, così, la trascrizione.
Negli operoni positivi repressibili, la proteina attivatore si trova legata all'operatore in condizioni normali, e la
trascrizione avviene perciò regolarmente. Determinate molecole però possono legarsi all'attivatore e imperdirgli,
cambiandone la conformazione, di legarsi all'operatore. In questo modo, la trascrizione viene inibita
lattosio
Il lattosio è un disaccaride formato da
galattosio e glucosio
Rappresenta per la cellula una fonte di
glucosio da cui ricavare energia
L’utilizzo del lattosio da parte del batterio
richiede più enzimi (βgalattossidasi,
permeasi) codificati da geni strutturali
controllati dagli stessi geni regolatori =
operone lac
Operone lac: la struttura
GENI
regolatore
promotore operatore
Geni strutturali
DNA
RNApolimerasi
trascrizione
RNA
Proteina repressore
attiva
RNA
repressore inattiva
lattosio
RNA
RNA
traduzione
Successione eventi: la repressione
1- in assenza del substrato (lattosio) la proteina repressore si lega a un tratto di
DNA (operatore) e blocca l’accesso alla RNApolimerasi al promotore dal quale
inizia la trascrizione dei geni strutturali
RNA polimerasi non può legarsi:
trascrizione bloccata
Promotore lac
Il repressore viene codificato
da un gene regolatore
repressore attivato
repressore legato
all’operatore
Induttore
(lattosio)
L’induzione
2- In presenza del substrato (induttore), questo si lega alla proteina
repressore e libera l’operatore.
3- RNApolimerasi si lega all’operatore e inizia la trascrizione
RNApolimerasi si lega
al
promotore
Inizia la trascrizione
dei geni strutturali
Induttore (substrato)
legato al repressore
mRNA trascritto
Operone
spento
Operone
acceso
La logica del vivente
In presenza di glucosio
non è necessario
ricorrere al lattosio per
ricavare energia.
La sintesi degli enzimi
che attaccano il
lattosio viene
repressa.
In carenza di glucosio,
occorre ricorrere al
lattosio per ricavare
energia.
Il lattosio si lega al
repressore e libera il
gene operatore.
Geni e loro funzioni
• Gene regolatore
• Gene promotore
• Gene operatore
• Geni strutturali
• Proteina repressore
• Sito di avvio della trascrizione del
DNA in mRNA
• Sito di inserimento per repressore:
blocco della sintesi dell’ mRNA
• enzimi
promotore
RNA polimerasi
operatore
STRUTTURA DELL’OPERONE
- IN ASSENZA DEL SUBSTARTO, LA PROTEINA REPRESSORE SI LEGA A UN
TRATTO DI DNA (gene operatore) E BLOCCA L’ACCESSO ALLA RNA
POLIMERASI AL GENE PROMOTORE DAL QUALE INIZIA LA TRASCRIZIONE
DEI GENI STRUTTURALI:
IN PRESENZA DEL SUBSTRATO (INDUTTORE), QUESTO SI LEGA ALLA PROTEINA REPRESSORE
E LIBERA L’OPERATORE. IN QUESTO MODO, L’RNA POLIMERASI SI LEGA ALL’OPERATORE E
INIZIA LA TRASCRIZIONE:
NEGLI EUCARIOTI ANCHE IL LIVELLO DI CONDENSAZIONE DELLA
CROMATINA E’ UN ULTERIORE SISTEMA DI CONTROLLO
CORPO DI BARR
L'inattivazione del cromosoma X, è una peculiarità dei caratteri legati al sesso nei mammiferi,
legata alla regolazione dell'attività dei geni.
Nei nuclei delle cellule somatiche femminili è visibile il corpo di barr che corrisponde al cromosoma X
altamente condensato e per la maggior parte inattivato e che questo viene scelto a caso tra i
cromosomi X di derivazione materna o paterna, secondo un processo indipendente da cellula a
cellula. Una volta che il cromosoma X è stato inattivato in una cellula, tutti i discendenti da quella
cellula erediteranno il tipo di inattivazione
Gli organismi con i cromosomi sessuali hanno un diverso dosaggio genico, cioè un numero di coppie
alleliche diverso tra i due sessi, (una femmina normale ha infatti due coppie per ogni locus del
cromosoma X, mentre un maschio ne possiede una sola).
L’inattivazione di un cromosoma X è responsabile della compensazione del dosaggio
L'inattivazione non avviene nelle cellule germinali, in quanto questo causerebbe la mancata
espressione dei geni legati al sesso nello zigote.






![Lezione 13 Codice genetico e sintesi proteica [modalità compatibilità]](http://s1.studylibit.com/store/data/000920742_1-3ab75c8c6018e2c240427969189fe9cd-300x300.png)


