VARIABILITÀ GENETICA ED EFFETTI TERAPEUTICI DELLE STATINE Come è ben noto ai medici e ai pazienti , esistono profonde differenze nella risposta del singolo individuo al trattamento con i farmaci, sia per quanto riguarda gli effetti terapeutici che le reazioni avverse e gli effetti collaterali. Questa variabilità può dipendere da fattori di tipo metabolico, di genere e di età o a interazioni con altre sostanze o farmaci, ma negli ultimi due decenni si sono sviluppati molti studi volti ad indagare l’influenza dei fattori di tipo genetico. Basandosi sull’ipotesi che sequenze differenti di nucleotidi dei geni codificanti per proteine regolanti l’assorbimento, il metabolismo e l’eliminazione di un farmaco o altri codificanti recettoriali, secondi messaggeri, potessero giustificare le variazioni inter-­‐individuali nella risposta ai farmaci, sono stati progettati e condotti numerosi studi di farmacogenetica. L'obiettivo di questi studi è sostanzialmente quello di predire , o almeno prevedere la risposta individuale a un certo principio attivo e a un certo dosaggio tramite un test genetico, ottenendo così una strategia terapeutica ottimale e personalizzata, con minore incidenza di reazioni avverse e migliori risultati terapeutici . La farmacocinetica e la farmacodinamica di ogni principio attivo sono regolati dall’intervento di numerosi e complessi percorsi metabolici e recettoriali in cui sono coinvolte proteine codificate da geni diversi. È, inoltre, risaputo che la maggioranza dei geni contiene variazioni individuali della sequenza nucleotidica, che possono determinare l’introduzione di uno o più aminoacidi diversi in una specifica proteina , con possibili ripercussioni sulla sua funzione. Un altro aspetto delle mutazioni è quello di modificare i meccanismi di trascrizione e traduzione dei prodotti di espressione genetica (mRNA e proteine appunto) determinando sovra o sotto espressione dei medesimi. Questi polimorfismi sono in genere puntiformi (ossia interessanti un singolo nucleotide), ma possono anche manifestarsi su ampi tratti di DNA in forma di inserzioni, delezioni o traslocazioni. L’assetto genetico di ognuno, quindi, determina, in modo più o meno ampio, in base all’ entità delle mutazioni, le caratteristiche dei recettori e delle proteine coinvolte nei processi di assorbimento e metabolismo, con ovvie ricadute in termini di efficacia del farmaco e di effetti collaterali, come dimostrato dall’interazione con alcuni citocromi molto importanti, in quanto sono responsabili del metabolismo di Fase I per molti farmaci di uso corrente e per alcune sostanze endogene (es. ormoni). Questa classe di enzimi è responsabile della maggior parte delle reazioni di ossidazione a livello epatico: la loro funzione è quella di trasformare diverse sostanze di origine endogena o esogena e ogni forma di citocromo ha una sua specificità per quanto riguarda la sostanza da ossidare. Polimorfismi attualmente noti di enzimi metabolizzanti i farmaci Fortunatamente, il numero degli enzimi metabolizzanti i farmaci è relativamente contenuto; in particolare sono noti il sistema enzimatico del citocromo P450 e i geni che codificano questi enzimi, detti CYP: nell’uomo ne sono stati identificati 57, suddivisibili in 18 famiglie e 42 sub-­‐
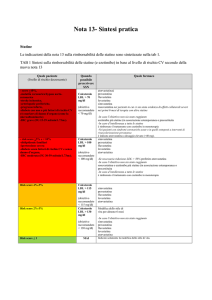
famiglie in base alla percentuale di sequenze aminoacidiche identiche. Anche la quantità di polimorfismi attualmente noti è piuttosto esigua, ciò nondimeno vengono attualmente riconosciute quattro categorie di soggetti in base alle caratteristiche di metabolizzazione: 1) metabolizzatori lenti con capacità di inattivazione ridotta o assente ( in genere mutazione in entrambi gli alleli del gene). 2) metabolizzatori intermedi con un allele normale ed uno inattivo, in cui potrebbero essere sufficienti dosaggi più bassi. 3) metabolizzatori rapidi con due alleli normali e normale metabolismo farmacologico. 4) metabolizzatori ultrarapidi con sovra espressione dei geni coinvolti nel metabolismo dei farmaci, per cui possono essere necessari dosaggi superiori alla norma. Gli inibitori della HMGCoA reduttasi (statine) sembrano essere tra le classi di farmaci in cui tali fenomeni si manifestano in modo più evidente. Esistono come è noto delle sensibili differenze di efficacia fra le diverse molecole , (fig.1) Variazioni di risposta alla stessa molecola da un individuo all’altro Ma oltre alle diversità legate all’azione della singola molecola esistono importati variazioni di risposta alla stessa molecola da un individuo all’altro. Una metanalisi farmacogenetica pubblicata su Nature Communications condotta su oltre 40 mila soggetti in trattamento con statine ( 18 studi fra RCT e osservazionali), ha infatti permesso d riconoscere due nuove varianti genetiche,in grado di interferire con la risposta al trattamento con statine in termini di riduzione del colesterolo LDL ; le varianti genetiche individuate finora davano conto di circa il 5% della variabilità interindividuale, dati ricavati da studi genetici (meta-­‐analysis of genome-­‐wide association studies :GWAS nei trial JUPITER, CARDS e ASCOT). Il lavoro in oggetto realizzato dal consorzio GIST (Genomic Investigation of Statin Therapy), ha invece individuato due loci completamente diversi, anch’essi implicati nella variabilità individuale di risposta alle statine. Anche per quanto attiene agli effetti collaterali delle statine è stata riconosciuta l’esistenza di un polimorfismo a singolo nucleotide rs4363657 del gene SLCO1B1 sul cromosoma 12 fortemente correlato con la miopatia da statine. L’ipotesi degli Autori è che, per quanto non sia ancora possibile (verrebbe da dire “pensabile”) sottoporre ad un test genetico i pazienti candidati al trattamento con statine, questa evidenza potrebbe rivelarsi preziosa , in futuro per scegliere la terapia ipocolesterolemizzante più appropriata e personalizzata per ogni singolo paziente. L’intenzione è buona, tuttavia restano aperte alcune questioni. Innanzitutto, come detto all’ inizio, il problema della variabilità geneticamente indotta alla risposta ai farmaci non riguarda solo le statine; basti pensare alle notevoli variazioni di effetto degli anticoagulanti orali (vecchi e nuovi …), agli antiaggreganti piastrinici (soggetti resistenti al clopidogrel). In secondo luogo le metanalisi hanno il pregio di raggiungere grandi numeri di pazienti, ma talvolta e questo ci sembra il caso, rischiano di mescolare eccessivamente le carte. Il grande numero di studi inseriti, infatti rende probabile che molte , se non tutte le statine siano state coinvolte, ma quanti pazienti assumevano l’ una o l’altra e quali di questi manifestava effetti diversi? E li avrebbe manifestati in egual misura con un'altra molecola. In altri termini la farmaco varianza geneticamente indotta è di classe o di molecola? Forse sarà un quesito fuori luogo se posto nei luccicanti saloni delle feste della ricerca avanzata, ma nelle fangose trincee della medicina quotidiana ha la sua importanza. Così come sarebbe interessante conoscere la ricaduta di queste scoperte sulla potenziale induzione di diabete da parte delle statine. Indagine genetica e costi E non possiamo chiudere queste righe senza rivolgere un pensiero all’ aspetto economico: il costo di un’ indagine genetica. per valutare la risposta dei Citocromi coinvolti nel metabolismo delle statine, oscilla fra i 150 e i 200 euro, a questi costi vanno aggiunti i costi imputabili al cosiddetto ‘effetto a cascata’. I pazienti infatti, ottenuto il risultato, necessitano di un consulto qualificato al fine di poter tradurre in provvedimenti pratici le indicazioni che il test mette in evidenza, questo comporta logicamente ulteriori prestazioni sanitarie e costi aggiuntivi per il sistema sanitario. Se si pensa che nell’attuale contesto di risorse limitate e di politica di risparmio estremo, la classe farmacologica in oggetto, peraltro inserita in una nota ministeriale che ne limita l’ erogazione a carico del SSN, all’ interno delle ASL è oggetto di costanti indagini, dibattiti e discussioni tra Specialisti, MMG e amministratori , è quantomeno difficile immaginare di sottoporre i pazienti ad indagini genetiche al fine di “scegliere la statina più efficace”. Antonio Ferrero SC Cardiologia ASLTO5, CdL Magistrale Professioni Sanitarie TD Università di Torino Tecla Marchese Geriatria e Malattie Metaboliche, Clinical Investigator Diabetologia ASLTO5 BIBLIOGRAFIA: •
www.laboratoriogenoma.eu/prestazioni_categoria.asp?IdCat=31 •
www.chimeragenomics.it/test-­‐diagnostici/farmacogenetica •
http://www.quotidianosanita.it/scienza-­‐e-­‐farmaci/articolo.php?articolo_id=24063&fr=n • NATURE COMMUNICATIONS | 5:5068 | DOI: 10.1038/ncomms6068 |www.nature.com/naturecommunications •
www.giornaledicardiologia.it/articoli.php?archivio=yes&vol_id • Emilsson, V. et al. Genetics of gene expression and its effect on disease. Nature 452, 423–428 (2008) • Fehrmann, R. S. et al. Trans-­‐eQTLs reveal that independent genetic variants associated with a complex phenotype converge on intermediate genes, with a major role for the HLA. PLoS Genet. 7, e1002197 (2011) • Stranger, B. E. et al. Population genomics of human gene expression. Nat. Genet. 39, 1217–1224 (2007). • Kwan T. et al. Genome-­‐wide analysis of transcript isoform variation in humans. Nat. Genet. 40, 225–231 (2008). • Mehta, D. et al. Impact of common regulatory single-­‐nucleotide variants on gene expression profiles in whole blood. Eur. J. Hum. Genet. 21, 48–54 (2013) • Huang, R. S. et al. Population differences in microRNA expression and biological implications. RNA Biol. 8, 692–701 (2011). Circulation 2004:110:227-­‐239