La maggior parte dei trascritti del genoma sono
RNA non codificanti per proteine
RNA non codificanti
large - rRNA 18+28S
Xist
……
……
…?…
small - 5S rRNA
tRNA
snRNAs
snoRNAs
scRNAs
gRNAs
piRNAs
miRNAs
siRNAs
rasiRNAs
…?….
traduzione
traduzione
splicing
modificaz./process. rRNA
controllo traduzionale
editing
stabilità del genoma
controllo traduzionale
stabilità dell’RNA
silenziamento trascriz.
Modificazioni dell’rRNA
• Metilazione (55 lievito, 110 vertebrati)
• Pseudouridilazione (45 in lievito, 100 vertebrati)
snoRNA e snoRNP
• Gli small nucleolar RNA (piccoli RNA nucleolari o snoRNA) sono
piccoli RNA coinvolti in alcune reazioni di modificazione chimica
a carico di rRNA e tRNA
• Queste modificazioni (metilazione e pseudouridilazione) sono in
grado di aumentare l'attività dell'RNA maturo.
• Gli snoRNA sono RNA di 60-300 nucleotidi
• Sono spesso codificati nelle sequenze introniche di proteine
ribosomiali oppure sono trascritti indipendentemente come
unità policistriniche
snoRNA e snoRNP
• Assieme a proteine costituiscono le small nucleolar
ribonucleoprotein (piccole ribonucleoproteine nucleolari o
snoRNP)
• La funzione dei snoRNA è quella di guidare il complesso proteico
snoRNP sul sito di modificazione dell'RNA bersaglio attraverso un
appaiamento specifico mentre la componente proteica esegue la
reazione chimica
• Box C/D
metilazione
• Box H/ACA
pseudouridilazione
La proteina Snu13p interagisce con entrambe le box C e D degli snoRNA e riconosce il
motivo di struttura K-turn. Nop1p (la fibrillarina) interagisce con entrambe le box D e D’
(non mostrato) mentre le proteine Nop58p e Nop56p riconoscono la box D e la box D’,
rispettivamente. A monte delle box D e D’ sono mostrate le sequenze guida antisenso e
complementari alla molecola di RNA target.
Nei loop interni delle strutture a forcina sono
presenti le sequenze guida
bipartite complementari all’RNA target che
fiancheggiano i due nucleotidi non
appaiati, di cui uno è l’uridina che verrà
pseudouridilata.
Il sito di pseudouridilazione è posizionato a
13-16 nt a monte delle box H/ACA.
Lewin, IL GENE VIII, Zanichelli
editore S.p.A. Copyright ©
2006
Maturazione di pre-rRNA nei batteri
Maturazione di pre-rRNA negli eucarioti
Tipi di splicing
• Splicing tRNA
nucleasi e ligasi
• Splicing di introni di tipo I
autosplicing
• Splicing di introni di tipo II
• Splicing nucleare
spliceosoma
transesterificazioni
meccanismo
simile
Splicing di tRNA
Autosplicing
Introni di gruppo I e II
Confronto dei meccanismi di splicing
Tutti i tipi di spicing comportano due reazioni di trans-esterificazione irreversibili
per degradazione dell’introne
Tutti gli introni di gruppo I e II sono caratterizzati da una complessa struttura
tridimensionale, essenziale per il loro meccanismo di azione (autocatalisi)
Introni di gruppo I
Lo splicing degli introni del gruppo I riguarda principalmente i geni che
codificano rRNA, tRNA e mRNA degli organelli di funghi e piante
Struttura dell’introne
dell’rRNA di Tetrahymena
In vivo sono necessari fattori proteici per il corretto ripiegamento dell’RNA
Proteine codificate dagli introni stessi
Lo splicing degli introni del gruppo I richiede l’intervento di una guanosina
esterna (GTP) che si lega in una tasca dell’introne che deve essere rimosso
(sito di legame G)
Il GTP attacca con il gruppo 3’OH il fosfato del sito di splicing al 5’ (di solito U)
Reazione di trans-esterificazione
Liberazione dell’esone al 5’ e legame del GTP all’estremità 5’ dell’introne
All’estremità 5’ dell’introne c’è un residuo di guanina che si lega al sito di legame G
Reazione di esterificazione
L’estremità 3’-OH del primo esone attacca il legame estere al sito 3’ di splicing
unendosi così al secondo esone e liberando l’introne
Introni di gruppo II
Introni di gruppo II
• sono meno comuni degli introni di gruppo I
• si trovano nei genomi degli organelli di funghi e piante (la maggior parte
negli introni dei cloroplasti)
• la reazione di self-splicing è molto più lenta di quella degli introni di
gruppo I
Meccanismo di azione
Il meccanismo è diverso da quello degli introni di gruppo I e somiglia a
quello utilizzato dallo spliceosoma negli mRNA eucarioti
• le giunzioni di splicing al 5’ (GUGYG) e al 3’ (AY) sono conservate
•
•
•
L’RNA si ripiega in una struttura conservata con la formazione di un sito
attivo che lega ioni Mg+2
Sei domini a doppia elica attorno a un core centrale
Non utilizzano una guanosina esterna ma un residuo interno di
adenosina (presente nel VI dominio)
Struttura generale degli introni di gruppo II
Un residuo di Adenina nell’introne
guida l’attacco nucleofilo
(step 1).
Le frecce indicano le giunzioni esone-introne
L’esone è tratteggiato
splicing degli introni del gruppo II
in mRNA mitocondriali, di cloroplasti, funghi, piante
In vivo sono necessarie proteine per il corretto ripiegamento dell’RNA
Il 2’OH del residuo di adenosina attacca il fosfato della giunzione al 5’ formando un legame
2’-5’ con la prima base dell’introne (G)
reazione di trans-esterificazione
L’esone a monte viene liberato mentre l’introne assume una struttura a cappio (lariat)
L’esone a monte attacca con la sua estremità 3’OH la giunzione di splicing al 3’
reazione di esterificazione
La reazione porta alla giunzione dei due esoni e la liberazione dell’introne a cappio
Processo a due step
Splicing nucleare
Il DNA non codificante è quindi quella
parte di genoma che maggiormente
varia al variare della complessità
INTRONI
UTRs
REGIONI
INTERGENICHE
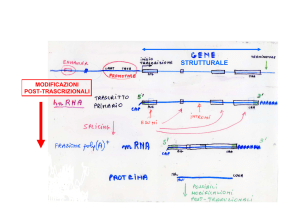
• DNA -> RNA eterogeneo (hnRNA) -> mRNA
• Il passaggio hnRNA -> mRNA consiste in:
– Incappucciamento: alterazioni chimiche all’estremità 5’
– Splicing : rimozione degli “introni”
– Poliadenilazione: sostituzione dell’estremità 3’ con
un’estensione di circa 250 basi A non presenti nella
sequenza del gene
• Introni/Esoni
• Esistono almeno 8 tipi diversi di introni
• Quello associato in modo predominante ai geni che codificano
proteine segue la “regola GU-AG” (cioè: introne = GU*…*AG)
• Esistono delle regole ben precise che determinano la rimozione
precisa degli introni
• Splicing alternativo
mRNA – Modifiche al 5’ e al 3’
mRNA – Modifiche durante la trascrizione
SCAF: splicing complex associated factors
Lewin, IL GENE VIII, Zanichelli
editore S.p.A. Copyright ©
2006
Esoni e domini funzionali delle proteine
Lewin, IL GENE VIII, Zanichelli
editore S.p.A. Copyright ©
2006
Le giunzioni di splicing sono conservate
Sequenze minime di consenso di spicing, numero minimo di complementarietà
Mutazioni in questa regione alterano lo splicing
Lo splicing degli mRNA richiede lo “spliceosome”
un complesso RNA-proteine
spliceosome
(~100 proteins + 5 small RNAs)
pre-mRNA
spliced mRNA
snRNA (smal nuclear RNA)
U1 snRNA
U5 snRNA
U2 snRNA
U4 snRNA
U6 snRNA
I 5 snRNA sono
associati a proteine
formando complessi
ribonucleoproteici
chiamati snRNP
Lo spliceosoma
5 RNA e più di 200 proteine (7 comuni) (snurp snRNP) formano lo spiceosoma
The spliceosome is a ribonucleoprotein complex
composed of multiple snRNPs
Lo spliceosomea
Il complesso snRNP U1
Interazioni tra
sequenze introniche
e snRNA
U1 = sito di splicing 5’
U2 = sito di ramificazione
U6 = sito di splicing 5’
Lewin, IL GENE VIII, Zanichelli
editore S.p.A. Copyright ©
2006
Lewin, IL GENE VIII, Zanichelli
editore S.p.A. Copyright ©
2006
Formazione del complesso di pre-splicing (complesso early)
(5 appaiamenti) con U1
Py:polipirimidina SC35, ASF/SF2 auxiliary splicing factor
fattori di splicing ricchi di ser/arg
Due vie per la definizione delle giunzioni
Riconoscimento del sito donatore più stringente
Tappe iniziali dello splicing del pre-mRNA
Complesso A: assembling U2 sul sito di ramificazione richiede ATP
Complesso B: binding
Tappa finale dello splicing
Complesso C: catting reazioni di transesterificazione
U6 si lega a U2 con una struttura a forcina
The spliceosomal
splicing cycle
La composizione dello
spliceosoma cambia
nel corso dello splicing
Regolazione dello splicing
Difetti nello splicing implicati in numerose malattie
Splicing alternativo fonte di variazione genetica e
meccanismo di regolazione di attività cellulari
Conseguenze di mutazioni delle sequenze consenso di splicing
Fattori che modulano lo splicing
Proteine con motivi DEXH/D box: RNA-elicasi ATP-dipendenti
Svolgimento di brevi tratti a doppia elica presenti nel pre-mRNA
Proteine SR (ricche in Ser e Arg fosforilabili): proteine modulari
con dominio di legame per l’RNA (RRM) e dominio RS
Legame a elementi di enhancer di splicing per stimolare il legame di U2AF
Legame ai siti di ramificazione per stimolare il legame di U1 al sito 5’
Sequenze enhancer e silenziatori di splicing esoniche (ESE e ESS)
Sequenze enhancer e silenziatori di splicing introniche (ISE e ISS)
Richiamano rispettivamente le proteine SR (attivatori che richiamano U2AF)
o le proteine hnRNP (inibitori che impediscono il legame dello spliceosoma)
regolazione positiva dello splicing
controllo negativo del sito di splicing
Splicing alternativo
Splicing alternativo
• Scoperto inizialmente nel 1980
• Meccanismo sottoposto a regolazione cellularte
anche se può essere casuale
• Il gene Dscam di Drosophila può avere sino a
38000 differenti variani di splicing
Gene DSCAM (codifica per una proteiba della
superficie cellulare coinvolta nella connettività tra
neuroni)
Il gene DSCAM è lungo 61.2 kb e dopo la trascrizione e
lo splicing produce un mRNA di circa 7.8 kb composto
da 24 esoni. Gli esoni 4, 6, 9, e 17 sono mutualmente
esclusivi e alternativi. Ogni mRNA finale prodotto
contiene una delle12 possibile alternative dell’esone
4, una delle 48 possibili per l’esone 6, una delle 333
possibili per l’esone 9 e una delle 2 possibili per
l’esone 17. Tutte le possibili combinazioni dei singoli
esoni 4, 6, 9, e 17 permettono di ottenere 38016
diverse varienti dell’mRNA per il gene DSCAM e
quindi altrettante proteine diverse.
Implicazioni nell’evoluzione
• Lo splicing alternativo modifica l’espressione del genoma
aumentandone la complessità
• Quante proteina sono prodotte dai genomi eucariotici?
• Quanti geni sono necessari per un organismo pluricellulare
complesso?
• Come le varianti di splice contribuiscono all’evoluzione?
• Quanto sono stabili i siti di splicing durante l’evoluzione?
Possibili effetti dello splicing alternativo
• Inclusione/esclusione di domini funzionali che possono
modificare la funzione o la localizzazione
• Variazioni nella struttura proteica
• Variazioni nella regione 5’ non tradotta che modificano
l’efficienza di traduzione
• Variazioni nella sequenza di poliadenilazione che
alterano la stabilità dell’mRNA
Diverse tipologie di splicing alternativo
proteoma più vario e complesso del trascrittoma
Many cellular factors may affect
which splice variant is produced
Pre-mRNA
RNA binding proteins
Pre-RNA secondary structure
Small ncRNAs?
Protein:protein complexes
Other mRNAs?
mRNA
1. The phenotype is determined by the proteome & transcriptome.
2. Selection acts on the phenotype, and is blind to the genotype.
Therefore: two species/individuals that have different forms of a protein will be selected
differently - even if the genes DNA sequence is identical.
CAMKII (chinasi II
calmodulina dipendente
Caspasi 2
-13-15-16-17-
-13-14-
-13-17-
Segnali pro e anti apoptotici
Attività simile ma diversa localizzazione
sarcolemma
nucleo
S = esone a splicing specifico
Fattore di trascrizione Zn-finger Gene fru
Espresso in alcuni neuroni sensori
Controllo del comportamento
P = promotori alternativi
citosol
multiple introns may be spliced differently in
different circumstances, for example in different
tissues.
Heart muscle
1
1
2
2
Uterine muscle
5
3
3
1
3
4
4
5
5
Thus one gene can encode more than one protein. The proteins are
similar but not identical and may have distinct properties. This is
important in complex organisms
Splicing alternativo della tropomiosina
scelta di un diverso promotore
Trans-splicing
Trans-splicing
Trans-splicing di gruppo II discontinuo
mitocondri vegetali
Scambio di tratti di DNA tra regioni geniche
differenti
Trans-splicing di leader attaccato
tripanosomi
fornisce una presequenza al 5’
Cappuccio a RNA trascritti dalla RNApol I o III
Utile per trascritti policistronici
Efficienza di traduzione (5’ non tradotto
dell’RNA coinvolto nel legame con i ribosomi)
Trans-splicing del t-RNA
Alcuni organismi
Ricongiungere le due metà del tRNA
presenti in due regioni geniche differenti
RNA-editing
Inserzione di U (Tripanosomi)
Conversione A>I e C>U (mammiferi)
RNA guida
Terminal-UridilTrasferasi
Ribonucleasi 3’->5’
U-specifica
Editing nei
mammiferi
ADAR
Editing differente nei due tessuti
ApoB-100 -> trasporto lipidi (LDL)
ApoB48 -> assorbimento lipidi
AA
AA
L’editing richiede
APOBEC1 = attività catalitica
ACF = fornisce la specificità del sito
AA
Stop
Stop
Stop