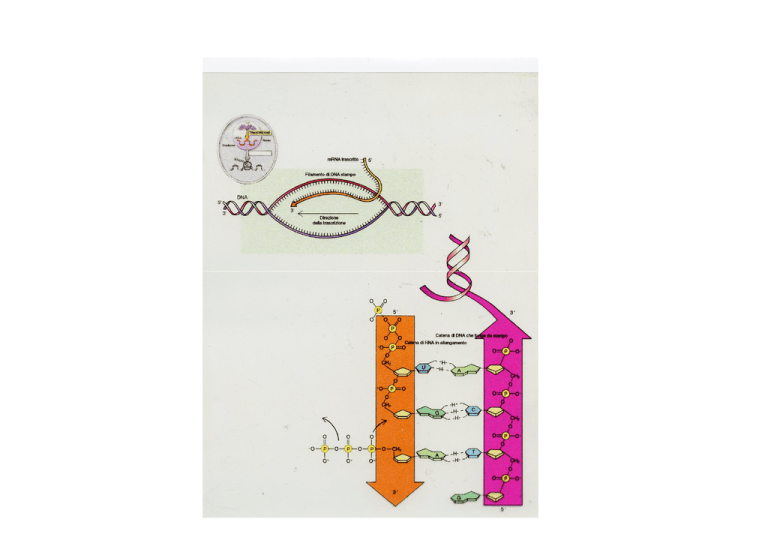
I meccanismi di catalisi della sintesi
di DNA e RNA sono identici
U
U
OH
Ribo-nucleotide trifosfato
TRASCRIZIONE
La RNA polimerasi è totalmente processiva
Non ha bisogno di innesco
Inizia a livello di un promotore
Assenza di efficace proof-reading
L’RNA prodotto si stacca subito dal DNA
Accurata regolazione a livello di ogni singolo gene
Trascrittoma = insieme dei trascritti presenti in una cellula
Sito di inizio
posizione +1
Posizioni
-
Posizioni +
DNA
ALLUNGAMENTO
INIZIO
(regolazione)
RNA
Comlesso chiuso
Complesso aperto
TERMINAZIONE
Trascrizione
iniziale
TRASCRIZIONE
PROMOTORE FORTE:
ha struttura più vicina
alla sequenza consenso
RNA POL PROCARIOTICA
•
•
•
•
•
beta
beta ‘
alfa 1
alfa 2
omega
+
• Sigma
(oloenzima)
Core enzimatico inizia la trascrizione “a caso”
La trascrizione
inizia solo in corrispondenza di un promotore
(sequenze -10 e -35)
Presente in promotori
particolarmente forti
Media l’apertura
del DNA
(AA aromatici)
Sito di inizio
della trascrizione
Proteina rho
simile a TRCF
TERMINAZIONE rho-indipendente
terminatore
Mutazioni
“negative”
Sito di legame
dell’attivatore
Sito di legame del
repressore
OPERONE LATTOSIO
Lattosio assente
Lattosio presente
OPERONE TRIPTOFANO
Poco
Trp
attivazione
Molto Trp
repressione
NEGLI EUCARIOTI
VARI LIVELLI DI CONTROLLO DELLA ESPRESSIONE GENICA
NEI PROCARIOTI:
PROCARIOTI:
soprattutto a livello
TRASCRIZIONALE
IN BATTERI: UNA SOLA POLIMERASI
che trascrive tutti gli RNA
rRNA
IN EUCARIOTI:
tRNA, rRNA 5S
3 TIPI DIVERSI ( I, II e III )
POL II MEDIA LA TRASCRIZIONE DEGLI mRNA
Intervento di numerosi fattori che regolano l’inizio della trascrizione
cromosoma
INIZIO DELLA
TRASCRIZIONE
ALLA TATATATA-BOX
TBP
- riconosce la TATA box
- piega il DNA
COMPLESSO DI
PRE-INIZIO
TFIIE e TFIIH
Mediano la transizione
da complesso chiuso - aperto
ULTIMO EVENTO
fosforilazione della coda
della pol II
Varie ripetizioni del peptide
Tyr-Ser-Pro-Thr-Ser-Pro-Ser
I FATTORI TRASCRIZIONALI (ATTIVATORI)
RECLUTANO:
• proteine che richiamano la RNA pol II
• proteine attive nello smantellamento dei
nucleosomi e sul rimodellamento della
cromatina
• proteine che stimolano l’inizio o
l’allungamento
FATTORI TRASCRIZIONALI
in “TRANS”
(proteine )
ELEMENTI
“IN CIS”
( Sequenze
del promotore)
ENHANCER
DISAGGREGAZIONE
DEI
NUCLEOSOMI
ENZIMI
MODIFICATORI DEI NUCLEOSOSMI
HAT
RIMODELLAMENTO
SWI / SNF
ATPasi
CONTROLLO COMBINATORIO
ELICA
DI
RICONOSCIMENTO
INTERAZIONI PROTEINE - DNA
ZINC FINGERS
LEUCINE ZIPPER
AMINOACIDI IDROFOBICI
A DISTANZA DI 7 aa
(1 GIRO DI ELICA = 3.6 aa)
LEUCINE ZIPPER
DOMINIO
ELIXELIX-LOOPLOOP-ELIX
b-HLH
dimerizzazione
Legame al
DNA
INIBITORI DELLA TRASCRIZIONE
E’ attiva solo in batteri
(ANTIBIOTICO)
E’ attiva in tutti gli organismi
E’ UN VELENO !!
E’ attiva solo in eucarioti
Inibisce pol II
e (parzialmente) pol III
E’ UN VELENO !!
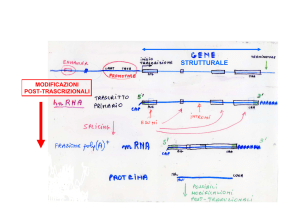
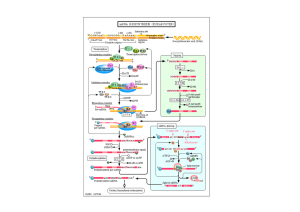
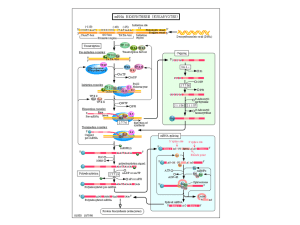
hnRNA (heterogeneus nuclear RNA)
oppure pre-mRNA
oppure trascritto primario
che diventa
mRNA
(insieme di molecole eterogenee citoplasmatiche)
+
rRNA 28S
rRNA 18S
tRNA
-
Elettroforesi in
gel di agarosio
con etidio bromuro
FORMAZIONE DEL Cap
al 5’ dell’ mRNA
GTP
GMP
Legame 5’ – 5’
Legame al
ribosoma
metilazione
5’
mRNA
Poli-adenilazione
dell’mRNA
poliA-polimerasi
Terminazione
RNAsi 5’ - 3’
“SILURO”
RNAsi 5’ - 3’
“rho – simile”
5’ NT “non tradotto”
3’ NT
Numero introni per gene
Sequenze consenso presenti sul
pre-mRNA
e che guidano lo splicing
Sito di splicing 5’
(donatore)
Y=CoU
R=GoA
N=AoGoCoU
Sito di splicing 3’
(accettore)
Prima
Trans-eserificazione
Avvicina
gli esoni
Seconda
Trans-eserificazione
degradazione
- Formazione del cappio
- Giunzione degli esoni adiacenti
- Rilascio dell’introne
Esone n
Prima transtrans-esterificazione
Esone n+1
Seconda transtrans-esterificazione
Esone n
Esone n+1
Introne
a cappio
SPLICING ALTERNATIVO
• Nei trascritti del 75% dei geni umani
• In genere 2 prodotti diversi, in alcuni casi molti di più
Il meccanismo di splicing “ignora” l’esone 3 oppure il 4
SPLICING ALTERNATIVO
Modalità diverse di splicing alternativo
Le diverse forme di mRNA possono essere
prodotte contemporaneamente o essere mutualmente esclusive
mRNA complessati con proteine specifiche
che ne mediano l’esportazione
•
•
•
Proteine che regolano dello splicing
che si legano alle giunzioni esone - esone
che si legano alla coda di poli-A
Complesso del
poro nucleare
Legame
al ribosoma
Cap
GLI INTRONI
sono stati persi dai procarioti (introni precoci)
o acquisiti dagli eucarioti (introni tardivi)
?
A COSA SERVONO I GENI INTERROTTI
?
Rimescolamento degli esoni nell’evoluzione.
Domini proteici spesso codificati da un esone
Domini presenti in proteine diverse
Duplicazione genica e trasposizione come possibile meccanismo