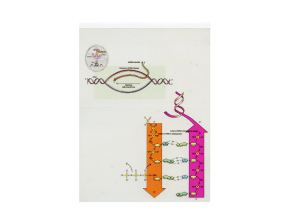
Trascrizione negli eucarioti
RNA pol II trascrive anche i microRNA
Sensibilità alla alfa-amanitina una tossina prodotta dal fungo: Amanita phalloides
Gli elementi di regolazione della trascrizione in cis
ALCUNE PROTEINE CHE LEGANO IL PROMOTORE
SONO REPRESSORI (RARE)
LA REPRESSIONE AGISCE PIU’
FREQUENTEMENTE A LIVELLO DELLA STRUTTURA
DELLA CROMATINA
Trascrizione geni eucariotici
Fattori generali della trascrizione: Costituiscono insieme alla RNA polimerasi II
L’APPARATO BASALE DELLA TRASCRIZIONE
TFIIX
TFIID= TBP (TATA binding protein)+ varietà di TAF (9)
( TBP associated factors)
TFII- H è una chinasi, una
ATPasi, una elicasi
La regolazione dello stato di
fosforilazione del CTD controlla
anche passaggi successivi
all’inizio di trascrizione
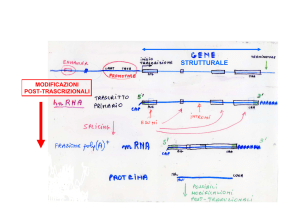
Complesso di taglio e
poliadenilazione
CPSF cleavage and polyadenylation
specificity factor
CstF cleavage stimulation factor
CF1 e CF2 endonucleasi
TERMINAZIONE TRASCRIZIONE
Due modelli per spiegare la terminazione della trascrizione negli
Eucarioti
A) Il trasferimento dei fattori di poli-adelinazione dalla RNA
POLIMERASI all’RNA innesca un cambio conformazionale della
proteina che ne rallenta l’attività e ne provoca l’arresto spontaneo
B) La mancanza del cap sulla 2° molecola di RNA viene avvertita
dalla polimerasi che si arresta
GENI TRASCRITTI DA POLIMERASI II
Prodotto della trascrizione è il pre-mRNA
SPLICING
Tutti geni per mRNA dei vertebrati contengono introni
(eccezione di alcuni istoni) 60% - 98% della lunghezza di un gene
FINO A 300 NEL GENE PER LA PROTEINA TITINA
Negli altri eucarioti la situazione è variabile (lievito POCHISSIMI
GENI HANNO INTRONI)
Alcuni geni di archeobatteri contengono introni
Alcuni geni per tRNA eucariotici contengono introni
-Variano in lunghezza da 50 a 10000 nt
-Non c’è conservazione di sequenza tra
introni dello stesso gene in organismi
diversi, anche vicini nella scala evolutiva
20-50 nt
Forma a cappio dell’introne
RNA splicing si realizza in due tappe
SPLICEOSOMA
snRNP (piccole particelle ribonucleoproteiche nucleari) e proteine
nucleari chiamate fattori di splicing
Lo “splicing” si può dividere in due tappe
a) Riconoscimento di sequenze consensus sull’RNA substrato e
assemblaggio dello spliceosoma
b) Reazione di taglio e “ligation” con modifica della struttura
dell’RNA substrato
Entrambe le catene di DNA possono fungere da filamento stampo
DNA GENOMICO DI ADENOVIRUS
FATTORI BASALI
APPARATO BASALE DELLA TRASCRI ZIONE
ATTIVATORI
AUMENTANO L’EFFICIENZA DI
INIZIO DELLA TRASCRIZIONE IN MODO
COSTITUTIVO O REGOLATO, INTERAGISCONO
CON I CO-ATTIVATORI O DIRETTAMENTE
CON I FATTORI BASALI
CO-ATTIVATORI
FORNISCONO UNA CONNESSIONE
TRA GLI ATTIVATORI E L’APPARATO BASALE
Enhancer: elemento che lega fattori di
trascrizione regolando la trascrizione
Differisce dai promotori in quanto:
1. Necessita di un promotore per
funzionare
2. Può essere molto distante dal sito di
inizio trascrizione (>10kbp)
3. Lavora in qualsiasi orientamento
TRASCRIZIONE EUCARIOTI
Fattori di trascrizione
fattori basali, attivatori (costitutivi, non costitutivi), co-attivatori, repressori
Enhancer
Promotore
200bp
100bp
Il promotore del gene umano dell’insulina
Nero: fattori ubiquitari
Rossi:fattori specifici delle cellule beta pancreatiche PDX è un fattore trascrizionale
fondamentale per lo sviluppo delle cellule B del pancreas
PROMOTORE EUCARIOTICO POLIMERASI II
-75
-25
Introni autocatalitici
Thomas Cech 1982
Proprietà catalitiche dell’RNA
Autosplicing
Introni di Gruppo II
Introni di gruppo I
Possiamo considerare gli snRNA dello
spliceosoma come agenti che
compensano la mancanza di
informazioni di sequenza degli introni
dei geni nucleari
Essi forniscono le informazioni
necessarie per formare particolari
strutture secondarie dell’RNA
Splicing alternativo:
Gli esoni possono essere allungati, accorciati, eliminati o inclusi
Gli introni possono essere rimossi o trattenuti, in parte o completamente da un
mRNA
Lo splicing alternativo può essere mediato da uso :
a)
b)
c)
sito di splicing alternativo al 5’
sito alternativo di splicing al 3’
exon skipping o salto dell’esone, la modalità più comune di splicing
alternativo nei mammiferi
d) introne trattenuto
e) esoni mutualmente escusivi
C) un esempio di un meccanismo complesso di splicing
alternativo in cui è presente contemporaneamente un sito alternativo
al 5’ e un salto dell’esone
Splicing alternativo
(a) Inclusione alternativa di un esone
(b) Esoni mutualmente esclusivi
(c) Sito di splicing 5’ alternativo
(d) Sito di splicing 3’ alternativo
(e) Conservazione di un introne
Gene regulation through alternative splicing is more versatile than is
regulation through promoter activity.
Changes in promoter activity alter predominantly the expression levels of
the mRNA. In contrast, changes in AS can modulate transcript expression
levels and alter the structure of the gene product by inserting, or deleting,
novel protein parts.
I cambi strutturali nell’mRNA possono avvenire:
a) nella regione codificante con:
- Introduzione, o delezione di tratti di sequenza
- Introduzione di un codone di stop prematuro della traduzione (PTC)
b) Nella regione al 5’ o 3’ UTR dell’mRNA
Gli effetti:
mRNA :
a) degradazione dell’mRNA con conseguente regolazione negativa
dell’espressione genica
b) Alterazioni della stabilità
b) Alterazioni nel processo di localizzazione intracellulare
Proteina: perdita di funzione o cambiamenti funzionali minimi di
difficile individuazione
Tra i due casi estremi si osservano alterazioni in:
a) proprietà di binding
b) localizzazione intracellulare
c) attività enzimatica
d) stabilità
e) modifiche post-traduzionali
In media un gene umano genera duo o tre trascritti, casi estremi: gene per la
proteina neurexin 3 può formare fino a 1728 trascritti dovuti a AS a
4 differenti siti
Gene della tropomiosina
Recettore IL-4
Recettore ormone della crescita
• The mechanism of splicing has been determined in great detail, in
contrast, it is not yet fully understood how splice sites are selected
• The major problem is the degeneracy of splicing regulatory
sequences, such as the 5 ’ , 3 ’ splice sites, branch points and
exonic/intronic sequence elements. These can only be described as
consensus sequences that are loosely followed
• As splice sites are degenerate, additional sequence elements
located in the exon or in adjacent intronic elements aid their
recognition by binding to regulatory proteins.
These proteins can be subdivided into serine-arginine-rich
SR proteins and hnRNPs
Architecture of a pre-mRNA and the important cis-acting sequence elements that direct the
splicing reaction
Exonic and intronic sequences modulate the splice site selection
SR: serine-arginine-rich proteins A/B: hnRNPs
In general these proteins bind weakly to RNA.
Increased specificity is achieved by the binding of multiple
proteins to RNA that is aided by protein-protein interactions
The formation of a specific protein-RNA complex from several
intrinsically weak interactions has several advantages:
a) the protein interaction can be influenced by small changes in
the concentration of regulatory proteins which allows the
alternative usage of exons depending on a tissue and/or
development-specific concentration of regulatory factors;
through these combination, a small number of splicing factors
regulate a large number of pre-mRNA that encode protein
isoforms with different biological properties
b) phosphorylation of regulatory factors that alter protein-protein
interactions can influence splice-site selection
Schematic diagram of the sequences and proteins involved in regulating
alternative splicing
Most of the splicing regulatory mechanisms that have been described act
by enhancing or preventing the binding of spliceosome to the pre-mRNA
Caratteristiche di un esone non costitutivo
mRNA 1
+
-
-
-
Sito di splicing 3’
+
-
- +
+
-
Sito di splicing 5’
mRNA 2