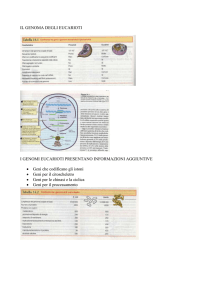
• Unavoltasequenziatoungenoma,occorre
iden5ficareigeniecercarediassegnareloro
unafunzione.
Ilmodellobiologico
Struttura di un gene eucariota
promotore
5’
Esone
Introne Esone
AUG gt
FaDoridi
trascrizione
AUG gt
ag
ag
Introne
gt
gt
ag
ag
3’
Esone
DNA
ag
TrascriDo
primario
TER
Sitoditaglio
SegnaleperilpolyA
CAP
AUG
5’UTR
AAA-AAA mRNA
TER
3’UTR
proteina
Leregionineiriquadricontengonosegnaliinterminidellelorosequenze
Ricercadigeni
• Ricercadisegnalidisequenza(mo5vi)
• Modellista5s5cidelgene(peresempioHMM)
• Similarità’congenino5nellostessooinaltri
organismi
Ricercadigeni
• Ricercadisegnalidisequenza(mo5vi)
• Modellista5s5cidelgene(peresempioHMM)
• Similarità’congenino5nellostessooinaltri
organismi
Matriciposizionespecifiche
Supponiamodiconoscerelesequenzedivarieistanzediunaregione
possiamoallinearleeiden5ficarelelorocaraDeris5chedisequenza
CTTGGTGACGTG!
TAGGATGAGTCG!
A
TACGTAGAGTCG!
TAGGATTTATCG!
T
TAGCGCGAGTCG! Conteggi
C
TAATCGCTACAG!
G
...!
1
2
3
4
5
6
7
8
9
10 11 12
0
6
0
0
2
1
0
4
2
0
1
0
6
1
1
1
1
3
1
2
0
4
1
0
1
0
1
1
1
1
1
0
1
1
4
0
0
0
3
4
2
1
4
0
3
2
0
7
Frequenze
1
2
3
4
5
A
0
.86 0
0
.29 .14 0
T
.85 .14 .14 .14 .14 .43 .14 .29 0
C
.14 0
.14 .14 .14 .14 .14 0
.14 .14 .57 0
G
0
.43 .57 .29 .14 .57 0
.43 .29 0
0
6
7
8
9
10 11 12
.57 .29 0
.14 0
.57 .14 0
1
Matriciposizionespecifiche
1
2
3
4
5
A
0
.86 0
0
.29 .14 0
T
.85 .14 .14 .14 .14 .43 .14 .29 0
C
.14 0
.14 .14 .14 .14 .14 0
.14 .14 .57 0
G
0
.43 .57 .29 .14 .57 0
.43 .29 0
0
6
7
8
9
10 11 12
.57 .29 0
.14 0
.57 .14 0
1
Seilnumerodicasièsufficientementealto.lefrequenzepossonoapprossimarele
probabilità.peròdobbiamotenercontoinqualchemododelcampionamentoincompleto
Nonpossiamoassumerechenontroveremomaiunasequenza
cheappar5eneallaclasseches5amoanalizzandoconunaCin
dodicesimaposizione
Siu5lizzailmetododeglipseudocount.Unopseduocountèunvalore(non
necessariamenteintero)chesiaggiungealnumerodicasiosserva5per
modificarelaprobabilitàaDesainunmodello.Peresempiopossiamo
aggiungere1oppure1/NdoveNèilnumerodicasi.
Matriciposizionespecifiche
1
2
3
4
5
6
7
8
9
10
11
12
A
0
6
0
0
2
1
0
4
2
0
1
0
T
6
1
1
1
1
3
1
2
0
4
1
0
C
1
0
1
1
1
1
1
0
1
1
4
0
G
0
0
3
4
2
1
4
0
3
2
0
7
1
2
3
4
5
6
7
8
9
10
11
12
A
1
7
1
1
3
2
1
5
3
1
2
1
T
7
2
2
2
2
4
2
3
1
5
2
1
C
2
1
2
2
3
2
2
1
2
2
5
1
G
1
1
4
5
3
4
3
1
8
1
2
3
4
5
6
7
8
9
10
11
Conteggiconpseudocounts
Frequenze
2
5
1
12
A
.09 .64 .09 .09 .27 .18 .09 .45 .27 .09 .18 .18
T
.64 .18 .18 .18 .18 .36 .18 .27 .09 .45 .18 .18
C
.18 .09 .18 .18 .18 .18 .18 .09 .18 .18 .45 .18
G
.09 .09 .36 .45 .27 .18 .45 .09 .36 .27 0.9 .73
Frequenze≅Probabilità
Perrendereivaloriaddi5viusiamoi
logaritmi
1
2
3
4
5
6
7
8
9
10
11
12
A
.09 .64 .09 .09 .27 .18 .09 .45 .27 .09 .18 .18
T
.64 .18 .18 .18 .18 .36 .18 .27 .09 .45 .18 .18
C
.18 .09 .18 .18 .18 .18 .18 .09 .18 .18 .45 .18
G
.09 .09 .36 .45 .27 .18 .45 .09 .36 .27 0.9 .73
1
2
3
4
5
6
7
8
9
10
11
12
A
-2.5
-0.7
-3.5
-3.5
-1.9
-2.5
-3.5
-1.1
-1.9
-3.5
-2.5
-2.5
T
-0.7
-2.5
-2.5
-2.5
-2.5
-1.5
-2.5
-1.9
-3.5
-1.1
-2.5
-2.5
C
-2.5
-3.5
-2.5
-2.5
-2.5
-2.5
-2.5
-3.5
-2.5
-2.5
-1.1
-2.5
G
-2.5
-2.5
-1.5
-1.1
-1.9
-2.5
-1.1
-3.5
-1.5
-1.9
-3.5
-0.5
Finestrascorrevolelungak
1
N
k
p[j=(k+1/2)]
p(j+1)
p(j+2)
p(j+3)
p(j+4)
p
posizione
Sogliaderivatadaunadistribuzionecasuale.peresempio
costruendovariesequenzecasualiconlastessacomposizionedi
quellareale.
Sequencelogo
1
2
3
4
5
6
7
8
9
10
11
12
A
0.15 0.95 0.18 0.15 0.54 0.38 0.15 0.84 0.49 0.15 0.58 0.09
T
1.06 0.27 0.36 0.30 0.36 0.77 0.30 0.51 0.16 0.77 0.58 0.09
C
0.30 0.14 0.36 0.30 0.54 0.38 0.30 0.17 0.33 0.31 1.46 0.09
G
0.15 0.14 0.72 0.75 0.54 0.38 0.75 0.17 0.66 0.46 0.29 0.73
Comeiden5fichiamolesoDosequenze(omo5vi)?
CTTGGTGACGTG!
TAGGATGAGTCG!
TACGTAGAGTCG!
TAGGATTTATCG!
TAGCGCGAGTCG!
TAATCGCTACAG!
...!
• Sperimentalmente(peresempio
ChipSeq)
• Confrontandoregionispecifichedi
genichesicomportanoallostesso
modo
– Gibbssampling
– MEME
– ...
Sequenzedigenicontrollatedallo
stessopromotore
CATCTACATGGACGGTAGACGACGTTGATGGACGTACAGTT...!
GTAGACAACGTTGGCTGCAGATGATGACGTAAATGCACACA...!
CTACACCGTGACACCAGGTACACGCTGAGGCGCAGGACATT...!
HannosoDosequenzecomunichequindisonoputa5vamenteilsitodilegame?
Sequenzedigenicontrollatedallo
stessopromotore
CATCTACATGGACGGTAGACGACGTTGATGGACGTACAGTT...!
GTAGACAACGTTGGCTGCAGATGATGACGTAAATGCACACA...!
CTACACCGTGACACCAGGTACACGCTGAGGCGCAGGACATT...!
HannosoDosequenzecomunichequindisonoputa5vamenteilsitodilegame?
Seconoscessimol’allineamento:
CATCTACATGGACGGTAGACGACGTTGATGGACGTACAGTT...!
GTAGACAACGTTGGCTGCAGATGATGC...!
CTACACCGTGACACCAGGTACACGCTGAGGCGCAGGACATT...!
1
2
3
4
5
6
A
0.15 0.95 0.18 0.15 0.54 0.38
T
1.06 0.27 0.36 0.30 0.36 0.77
C
0.30 0.14 0.36 0.30 0.54 0.38
G
0.15 0.14 0.72 0.75 0.54 0.38
Seconoscessimoilmo5vo:
1
2
3
4
5
6
A
0.15 0.95 0.18 0.15 0.54 0.38
T
1.06 0.27 0.36 0.30 0.36 0.77
C
0.30 0.14 0.36 0.30 0.54 0.38
G
0.15 0.14 0.72 0.75 0.54 0.38
p
posizione
...ACGGTAGACGACGTTGATGGACGT...!
Unpossibilemetodo:MEME
CATCTACATGGACGGTAGACGACGTTGATGGACGTACAGTT...!
GTAGACAACGTTGGCTGCAGATGATGACGTAAATGCACACA...!
CTACACCGTGACACCAGGTACACGCTGAGGCGCAGGACATT...!
Datepsequenzes1...sp,trovalesoDo-sequenzepiùsimili,unaper
ognisequenza.Inaltreparole:
argmin dist(si , s j )
s1,...,s p i< j
Dovedist(si,sj)èladistanzadiHamming(numerodiposizioniincuii
simbolisonodiversitraleduesequenze)
∑
Ricercadigeni
• Ricercadisegnalidisequenza(mo5vi)
• Modellista5s5cidelgeneodelleregioniche
contengonoigeni(peresempioHMM)
• Similarità’congenino5nellostessooinaltri
organismi
Esempio:isoleCpG
• Notazione:
– C-G–e’lacoppiadibasiC-GsulledueelichecomplementaridelDNA
– CpG–e’ildinucleo5deCG
• Processodime5lazionenelgenomaumano
– CmutaspessoinT,maquestosuccederaramentequandolaCe’partedi
CpG
– Quindinelleregioniintornoaipromotoridimol5geniIdinucleo5diCpG
sonomoltopiu’frequen5
– QuesteregionisonochiamateisoleCpG(disolitolunghedaqualche
cen5naioaqualchemigliaiadibasi)
• Problemi:
– Dataunasequenza,provienedaun’isolaCpG?
– ComeindividuoleisoleCpGinunasequenza?
ModellodiMarkovperdiscriminare
CpG
A
aAT
T
aAC
aGT
C
aGC
G
Lefrecceindicanolaprobabilita’a
cheunabaseprecedaosegua
l’altraoDenutedallefrequenze
osservateinuntrainingset.
• TrainingSet:
– SequenzediDNAchesisasonoisoleCpGe
sequenzechenonlosono
• Daida5siderivanoduemodellidiMarkov:
– ‘+’model:perleisoleCpG
– ‘-’model:pertuDoilresto
ModellodiMarkovperdiscriminare
isoleCpG
+
c st
• Con5amoilnumerodivolteincuiilsimbolotsegueil
simbolosnelleisoleCpG
+
cst
• Con5amoilnumerodivolteincuiilsimbolotsegueil
simbolosnelrestodellesequenze
cst−
• Lefrequenzesonoquindi: +
cst+
−
a =
ast =
∑
t'
+
cst'
st
∑c
−
t' st'
PossiamoassegnareunpunteggioSadunasequenzax:
S(x) = log
P(x|model + )
=
P(x|model − )
∑
a +x i −1 x i
L
log −
i =1
a x i −1 x i
A
C
G
T
A
.180
.274
.426
.120
C
.171
.368
.274
.188
G
.161
.339
.375
.125
T
.079
.355
.384
.182
Dataunasequenza,vienedaun’isola
CpG?
P(x|model + )
S(x) = log
=
P(x|model − )
∑
+
a x i −1 x i
L
log −
i =1
a x i −1 x i
ValoridiS(x)posi5viindicanocheèpiùprobabileche
provengadaun’isolaCpGchedalrestodelle
sequenzeeviceversa
ComeindividuoleisoleCpG?
posizione
Frequenzainregioninote
Score
Possousaredellefinestrescorrevoli.Peresempio,sceltauna
finestralunga101basi,calcoloilpunteggioeloassegnoalla
basecentrale
10
5
0
Non-CpG
CpG
islands
-0.4-0.3-0.2-0.100.10.20.30.4
Score
Possou5lizzaregliHMM(Hidden
MarkovModel)
SitraDainpra5cadicostruireunmodello
singolochetengacontodelleproprietàdi
entrambeleregioni(isoleeno)
Possou5lizzaregliHMM(Hidden
MarkovModel)
• Posso osservare solo i simboli emessi dal sistema ma
non so quale percorso è stato seguito (per esempio se la
base proviene da un’isola CpG o no)
• Le emissioni e le transizioni dipendono solo dallo stato
presente non su quello che e’ successo prima.
DuemodellidiMarkov
aAT
A
T
A
aGT
aAC
C
ast+
G
aGC
=
cst+
∑
t'
+
cst'
aAT
T
aGT
aAC
C
G
aGC
−
st
a =
−
st
c
∑c
−
t' st'
DuemodellidiMarkov
aAT
A+
T +
A-
aGT
aAC
C +
ast+
G+
aGC
=
cst+
∑
t'
+
cst'
aAT
T -
aGT
aAC
C -
G-
aGC
−
st
a =
−
st
c
∑c
−
t' st'
ModellonascostodiMarkov
A+
T +
C +
G+
A-
T -
C -
G-
Ulteriore(cioèle
connessioniall’interno
deiduesetnonsono
mostratemacisono)
setdiprobabilita’di
transizioneda+a-.
OgnistatoemeDeunsimbolo(chedipendedalleprobabilità
cheabbiamoassegnatoallevarietransizioni).
Dataunasequenza(peresempioCGCG)vorreisaperequalèla
probabilitàchesiastataemessadalmodelloedaqualestato
provieneciascunsimboloconmaggioreprobabilità.
PeresempiopotrebbeessereC+G+C-G-oppureC+G+C+G-
GenScanStates
•
•
•
N-intergenicregion
P-promoter
F-5’untranslatedregion
•
Esngl–singleexon(intronless)(transla5on
start->stopcodon)
•
Einit–ini5alexon(transla5onstart->donor
splicesite)
•
Ek–phasekinternalexon(acceptorsplice
site->donorsplicesite)
•
Eterm–terminalexon(acceptorsplicesite->
stopcodon)
•
Ik–phasekintron:0–betweencodons;1–
aqerthefirstbaseofacodon;2–aqerthe
secondbaseofacodon
Misurediaccuratezza
Matricediconfusione
Sensibilita’
TP/(TP+FN)
Specificità
TN/(FP+TN)
Caso
posi8vo
Caso
nega8vo
Predizione
posi5va
TP
FP
Posi5vepredic5ve
value(precisione)
TP/(TP+FP)
Predizione
nega5va
FN
TN
Nega5vepredic5ve
value
TN/(FN+TN)
Falsediscoveryrate
FP/(TP+FP)
Accuratezza
(TP+TN)/(TP+FN)
MCC =
TP × TN − FP × FN
(TP + FP)(TP + FN )(TN + FP)(TN + FN )
Esempio
Specificità
Considerandoinucleo5di
Considerandogliesoni
Coefficientedicorrelazione
Sensibilità
Metodidiversi
Numerodisequenzeanalizzate
perlapredizione (inparentesinumerodi
digeni
sequenzepercuièstata
predeDal’assenzadigeni
CurvaROC