Esercitazione corso di Biodiversità Animale e Genetica Molecolare
Archiviazione dei dati genotipici:
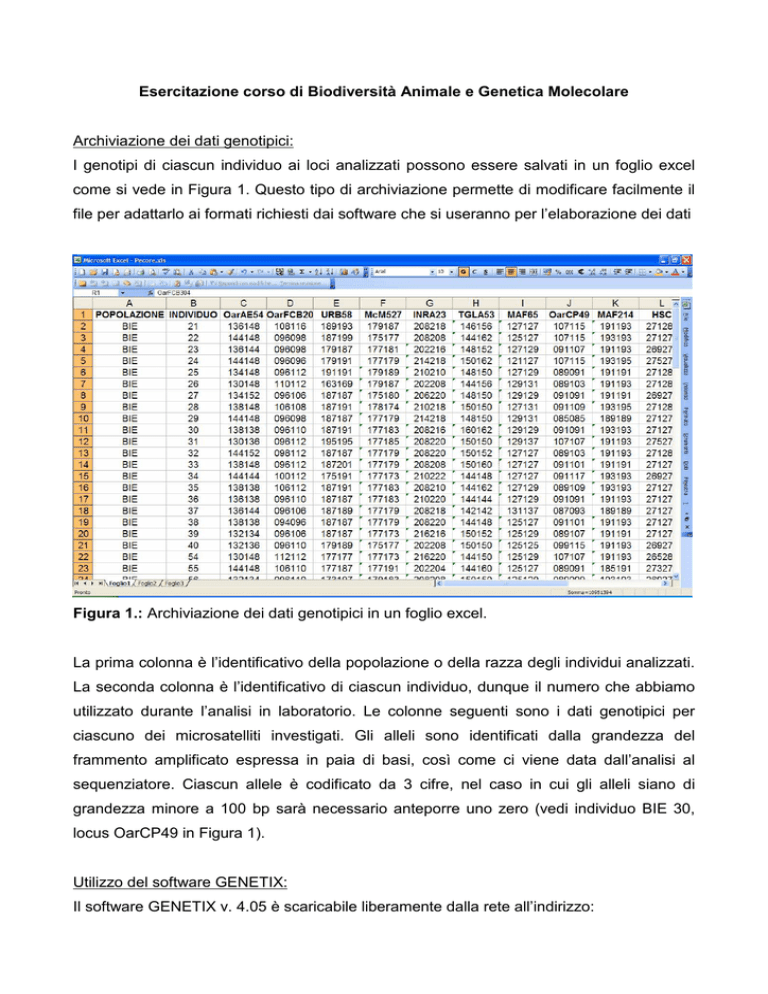
I genotipi di ciascun individuo ai loci analizzati possono essere salvati in un foglio excel
come si vede in Figura 1. Questo tipo di archiviazione permette di modificare facilmente il
file per adattarlo ai formati richiesti dai software che si useranno per l’elaborazione dei dati
Figura 1.: Archiviazione dei dati genotipici in un foglio excel.
La prima colonna è l’identificativo della popolazione o della razza degli individui analizzati.
La seconda colonna è l’identificativo di ciascun individuo, dunque il numero che abbiamo
utilizzato durante l’analisi in laboratorio. Le colonne seguenti sono i dati genotipici per
ciascuno dei microsatelliti investigati. Gli alleli sono identificati dalla grandezza del
frammento amplificato espressa in paia di basi, così come ci viene data dall’analisi al
sequenziatore. Ciascun allele è codificato da 3 cifre, nel caso in cui gli alleli siano di
grandezza minore a 100 bp sarà necessario anteporre uno zero (vedi individuo BIE 30,
locus OarCP49 in Figura 1).
Utilizzo del software GENETIX:
Il software GENETIX v. 4.05 è scaricabile liberamente dalla rete all’indirizzo:
http://www.genetix.univ-montp2.fr/genetix/genetix.htm
Questo programma permette di calcolare diversi parametri per lo studio della genetica di
popolazione, ha inoltre il vantaggio di creare input file per altri programmi che utilizzeremo
nel corso dell’esercitazione. Il suo più grande svantaggio è quello di essere scritto
completamente in francese.
Per creare l’input file è necessario copiare in blocco note il foglio excel visto in precedenza
dato che il programma richiede un file “.txt”. Poi si apre il programma cliccando sull’icona e
si seleziona dal menu “Fichier” (che corrisponde a File), la voce “Importer”; a questo punto
comparirà la finestra rappresentata in Figura 2.
Figura 2.: Finestra di importazione dati del software GENETIX.
Una volta spuntate le opzioni che si addicono al file che si vuole analizzare basterà
cliccare OK perché finisca il processo di importazione. Ora, nella schermata (Figura 3) si
vedrà il file così come lo abbiamo importato, inoltre nella prima riga sono indicate le
funzioni che si possono svolgere. Cliccando con il mouse sopra ciascuna delle voci si avrà
una descrizione più precisa di quella funzione. In questa esercitazione solo alcune funzioni
verranno utilizzate.
Figura 3.: Schermata principale di GENETIX una volta importato il file di input
Funzione Variabilité. Utilizzando questa funzione otteniamo:
•
Le frequenze alleliche a ciascun locus e per ogni allele in ciascuna delle
popolazioni studiate. Questa funzione ci permette di identificare i “private alleles”
•
L’eterozigosi attesa e osservata a ciascun locus e in ciascuna popolazione
permettendoci di capire quali siano le popolazioni più variabili
•
I valori dell’indice di fissazione FIS a ciascun locus e per ogni allele in ciascuna delle
popolazioni studiate
Inoltre, prima di ottenere il file con i risultati il programma ci chiede se desideriamo anche
creare il file di input per PHYLIP (un programma che useremo in seguito).
Funzione Distances. Questa funzione ci permette di ottenere una stima delle distanze
genetiche. La stima si ottiene a partire dalle frequenze alleliche osservate in ciascuna
popolazione. Le distanze genetiche calcolate sono:
•
Nei 1972
•
Nei 1978
•
Nei minimum distance
•
Cavalli-Sforza
Purtroppo non è facile scegliere quale sia la distanza genetica più adatta. Secondo molti
autori, nel caso si vogliano studiare razze europee (che si sono separate da centinaia di
anni e quindi da un periodo relativamente breve), quella più appropriata, tra quelle sopra
elencate, è la “Nei minimum distance”. In Figura 4 è rappresentato l’output relativo alla
stima di questa distanza. I risultati sono rappresentati sottoforma di una matrice e il
numero tra parentesi corrisponde al numero di individui presenti in ciascuna popolazione.
Figura 4.: Matrice delle distanza Nei minimum distance
Funzione Outils. Questa funzione permette di convertire (Conversion) l’input preparato
per GENETIX negli input utilizzati da altri software. In particolare, è possibile creare l’input
per GENEPOP e FSTAT che useremo nel corso dell’esercitazione. Inoltre, cliccando su
“Diagnostic” possiamo ottenere alcune statistiche relative ai nostri dati (numero di
alleli/locus nella campione totale e in ciascuna popolazione, popolazioni analizzate e loro
numerosità, grafici).
Utilizzo del software GENEPOP:
Anche questo software può essere scaricato liberamente dalla rete all’indirizzo:
http://kimura.univ-montp2.fr/%7Erousset/Genepop.htm
GENEPOP è in grado di svolgere diverse analisi che sono riassunte nella schermata
principale del programma (Figura 5). Quelle che utilizzeremo si riferiscono al paragrafo
“Testing” e servono a testare l’esistenza dell’equilibrio di Hardy-Weinberg e la presenza di
una significativa differenziazione tra le popolazioni oggetto di studio (opzioni 1 e 3).
Funzione Hardy-Weinberg exact test (1). Questa funzione presenta a sua volta diverse
scelte dovute al tipo di test che si desidera utilizzare. Nel caso i dati molecolari derivino da
marcatori microsatellite, che, per definizione, presentano molti alleli, l’analisi più
appropriata è quella del test di Guo e Thompson (1992, probability test).
Figura 5.: schermata principale del software GENEPOP
Il risultato del test viene dato a ciascun locus in ciascuna popolazione, se invece si utilizza
l’opzione Global Test si ottiene il risultato per ciascun locus nel totale degli individui
analizzati. Gli altri 2 test che testano l’eccesso o il deficit di eterozigoti sono test più potenti
del precedente (Rousset e Raymond, 1995).
Funzione exact test for population differentiation (3). Questa analisi serve a testare
l’ipotesi che all’interno della popolazione analizzata siano presenti più sotto-popolazioni (in
questo caso razze) che si differenziano una dell’altra sulla base delle frequenze alleliche. Il
programma paragona, a ciascun locus, ogni possibile coppia di razze ed infine dà un
risultato complessivo riassumendo le informazioni derivate da ciascun locus.
Utilizzo del software PHYLIP
Il PHYLIP è un pacchetto di software scaricabile liberamente all’indirizzo:
http://evolution.genetics.washington.edu/phylip.html
Questo pacchetto può essere utilizzato per molti tipi di analisi molecolari e può analizzare
sia sequenze molecolari che frequenze alleliche. Durante l’esercitazione utilizzeremo solo
i programmi utili per l’analisi di frequenze alleliche. In particolare useremo i programmi
necessari per stimare diverse distanze genetiche e per rappresentare graficamente tali
distanze tramite dei dendogrammi (alberi). I programmi vanno utilizzati in un ordine ben
preciso perché l’output di uno diventa l’input di quello successivo.
Seqboot. Questo è il primo programma da utilizzare. L’input file è quello che abbiamo
ottenuto dal GENETIX ed è rappresentato dalle frequenze alleliche osservate in ciascuna
delle razze analizzate. Questo programma permette di effettuare un ricampionamento dei
dati iniziali generando, a partire dall’input file, un numero variabile (100 – 1000) di nuovi
datasets tramite una tecnica chiamata “bootstrapping”. Questi dataset vengono generati a
caso e saranno poi analizzati nello stesso modo di quello originale
Figura 6.: Schermata del software Seqboot
In Figura 6 è rappresentata la schermata iniziale del software Seqbboot. Per cambiare i
parametri è sufficiente digitare la lettera che si trova a sinistra del parametro che si
desidera cambiare, quelli usati in figura sono i parametri che verranno utilizzati durante
l’esercitazione. Il file di output conterrà i 100 nuovi datests; a questo punto è necessario
nominarlo infile per poterlo utilizzare con il programma seguente, Genedist.
Genedist. Questo programma stima diversi tipi di distanze genetiche:
•
Nei 1972
•
Cavalli – Sforza
•
Reynold
Lo utilizzeremo per la stima delle distanze genetiche di Reynold. Infatti, questo tipo di
distanza è la più appropriata quando si confrontano razze europee, in quanto utilizza come
fonte di variazione tra le razze solamente la deriva genetica senza considerare la
mutazione. Infatti, con periodi di differenziazione relativamente brevi (centinaia di anni), la
mutazione non può aver fatto sentire i suoi effetti.
Genedist stimerà le distanze genetiche nel dataset originale e nei 100 nuovi datasets che
sono stati creati con Seqboot.
Figura 7.: Schermata principale del software Genedist
In Figura 7 è rappresentata la schermata principale di Genedist con i parametri da
utilizzare nel caso si vogliano stimare le distanze di Reynold analizzando 100 datasets. Il
file di output sarà dato da un file contenente tutte la stima delle distanze genetiche
sottoforma di matrici, ci saranno tante matrici quanti sono i datasets analizzati. Anche in
questo caso il file di output dovrà essere nominato “infile” e sarà utilizzato nel software
Neighbour.
Neighbour. Questo software permette di rappresentare graficamente le distanze
genetiche sottoforma di dendogrammi o alberi. Esistono 2 modi di costruire gli alberi a
partire dalle matrici delle distanze genetiche: UPGMA e Neighbour-Joining (NJ). Nel
nostro caso il tipo NJ è preferibile in quanto le popolazioni che stiamo studiando hanno
una differente numerosità effettiva e di conseguenza un diverso tasso di evoluzione, questi
sono gli assunti su cui si basa la costruzione di un albero NJ. In Figura 7 è rappresentata
la schermata principale di Neighbour.
Questa volta i file di output saranno 2, uno chiamato outfile con la rappresentazione
grafica degli alberi ottenuti da ciascuna delle matrici analizzate e l’altro outree con le
indicazioni necessarie a disegnare tali alberi.
Figura 8.: Schermata principale di Neighbour
Questa volta il file chiamato outree dovrà essere rinominato intree e sarà usato come input
per il programma Consense.
Consense. Questo è l’ultimo programma che utilizziamo. Serve per ricavare un solo
albero a partire da tutti quelli che sono stati disegnati. Vicino ai rami dell’albero sarà
indicato un numero che indica quante volte è stata trovata quel tipo di diramazione tra tutti
i datasets che sono stati analizzati.
La regola utilizzata per costruire questo “consensus tree” è quella indicata di default dal
programma (extended majority rule), questa include qualsiasi gruppo di popolazioni che è
apparso più del 50% delle volte negli alberi utilizzati come input. Se nessun gruppo appare
più del 50% delle volte vengono considerati i gruppi che appaiono con % minori ma che
sono i più frequenti.
A questo punto è possibile visualizzare l’albero nella sua forma finale. E’ importante
ricordare che questi non sono alberi filogenetici quindi la rappresentazione migliore
sarebbe quella “unrooted” cioè senza radici.
Figura 8.: Schermata principale di Consense
Per visualizzare l’albero unrooted è possibile utilizzare il programma Drawtree, sempre
compreso nel pacchetto PHYLIP, purtroppo però non è possibile visualizzare il valore di
bootstrap.