Anna Zimmermann
Resistenza batterica
2012/2013
Liceo Cantonale Lugano 1
Biomedicina: parassitologia, microbiologia e genetica
Resistenza Batterica Lavoro di maturità di:
Anna Zimmermann
Docente responsabile:
Vania Della Chiesa
2012/2013 1 Anna Zimmermann
Resistenza batterica
2012/2013
Abstract In questo lavoro si approfondisce il tema della resistenza batterica agli antibiotici β-lattamici, argomento
particolarmente attuale poiché essa si è sviluppata negli ultimi decenni e causa serie difficoltà nel campo
medico. Lo sviluppo di resistenze batteriche rende gli antibiotici inefficaci, mettendo così a rischio la vita
umana. Nel lavoro sono approfonditi alcuni aspetti teorici dei batteri, degli antibiotici β-lattamici (penicilline e
cefalosporine) e della resistenza batterica. Sperimentalmente sono stati ricercati i geni della resistenza ai βlattamici che determinano la produzione di ESBL (mutazioni dei geni TEM, SHV e OXA e il gene CTX-M) in
ceppi batterici della famiglia degli enterobatteri isolati da diversi ambienti (Lago Cadagno, acque del fiume
Ticino, prima e dopo il depuratore di Giubiasco, e acque di scarico dell’ospedale San Giovanni di Bellinzona).
Nel dettaglio 38 ceppi batterici, selezionati da 250 ceppi di partenza tramite un test selettivo per individuare
potenziali batteri ESBL, sono stati analizzati con antibiogrammi alla ricerca del fenotipo ESBL, che si
manifesta con una sinergia tra i tre antibiotici Augmentin (AMC), Ceftriaxone (CRO), Cefotaxima (CTX) e con
la sensibilità alla Cefoxitina (FOX). Per la ricerca di specifici geni di resistenza sono stati estratti i plasmidi dai
38 ceppi, sono state ampliate specifiche sequenze tramite PCR ed evidenziate mediante l’elettroforesi sul gel.
Questa procedura ha permesso di confermare la capacità di produrre enzimi ESBL per alcuni ceppi, assieme
al test dell’antibiogramma e all’esame selettivo, in quanto per definire con certezza questo fenotipo è
necessario eseguire più test. I risultati ottenuti nella parte pratica mostrano che, dei 38 ceppi selezionati dai
250 prelevati dagli ambienti, solo 4 risultano produttori di ESBL. Per questi ceppi sono stati trovati i seguenti
geni di resistenza: 1 gene CTX-M ,1 gene OXA, 2 geni SHV e 1 gene TEM.
2 Anna Zimmermann
Resistenza batterica
2012/2013
Sommario
1. Introduzione.............................................................................................................................................. 5 1.1 Premessa ........................................................................................................................................... 5 1.2 Obiettivi.................................................................................................................................................. 5 1.3 Ipotesi..................................................................................................................................................... 6 1.3.1 Ipotesi 1 ............................................................................................................................................ 6 1.3.2 Ipotesi 2 ............................................................................................................................................ 6 2. Svolgimento teorico .................................................................................................................................. 7 2.1 I Batteri.................................................................................................................................................... 7 2.1.1 Informazioni generali ....................................................................................................................... 7 2.1.2 La cellula batterica ........................................................................................................................... 8 2.1.2.1 Il citoplasma .................................................................................................................................. 8 2.1.2.2 Involucro cellulare......................................................................................................................... 8 2.1.2.3 Appendici facoltative .................................................................................................................... 9 2.1.3 Modalità di scambio genetico .......................................................................................................... 9 2.1.4 I batteri e l’uomo............................................................................................................................ 10 2.1.5 I batteri coliformi (Enterobatteri) .................................................................................................. 10 2.2 Gli antibiotici ................................................................................................................................... 10 2.2.1 Cosa sono gli antibiotici [1] ............................................................................................................. 10 2.2.2 I differenti tipi di antibiotici e le loro modalità d’azione [1,2]......................................................... 11 2.2.3 I β-­‐lattamici [6]................................................................................................................................ 11 2.3 La resistenza agli antibiotici ............................................................................................................ 12 2.3.1 Che cosa è la resistenza agli antibiotici [1]...................................................................................... 12 2.3.2 Cosa determina lo sviluppo della resistenza [1].............................................................................. 12 2.3.3 I tipi di resistenza [9] ...................................................................................................................... 13 2.3.4 Le β-­‐lattamasi di tipo ESBL [6]........................................................................................................ 13 3. Svolgimento pratico ................................................................................................................................ 14 3.1 Materiale............................................................................................................................................... 14 3.2 Procedimento........................................................................................................................................ 16 3.2.1 Preparazione dei 38 ceppi all’antibiogramma e antibiogramma ................................................... 16 3.2.2 Estrazione di DNA totale dal ceppo di controllo 23/07 + ESBL....................................................... 19 3.2.3 Estrazione dei plasmidi dai 38 ceppi d’interesse............................................................................ 19 3.2.4 Preparazione dei campioni alla PCR e PCR per i geni TEM, SHV, CTX-­‐M e OXA ............................. 20 3 Anna Zimmermann
Resistenza batterica
2012/2013
3.2.4.1 Preparazione dei campioni per la PCR normale per i geni CTX-­‐M e OXA.................................... 20 3.2.4.2 Preparazione dei campioni per la PCR multiplex per i geni TEM e SHV ...................................... 21 3.2.4.3 Amplificazione del DNA............................................................................................................... 21 3.2.5 Preparazione dei campioni per elettroforesi su gel e elettroforesi su gel ..................................... 21 3.3 Risultati ................................................................................................................................................. 22 3.3.1 Risultati antibiogrammi.................................................................................................................. 22 3.3.2 Risultati della PCR per i geni TEM, SHV, CTX-­‐M e OXA................................................................... 26 3.3.2.1 Risultati ricerca gene TEM........................................................................................................... 26 3.3.2.2 Risultati ricerca gene CTX-­‐M ....................................................................................................... 27 3.3.2.3 Risultati ricerca gene OXA ........................................................................................................... 28 3.3.2.4 Risultati ricerca gene SHV ........................................................................................................... 29 3.3.3 Sintesi dei risultati.......................................................................................................................... 30 3.4 Discussione............................................................................................................................................ 30 4. Conclusione................................................................................................................................................. 32 5. Ringraziamenti ........................................................................................................................................... 32 6. Bibliografia .................................................................................................................................................. 33 4 Anna Zimmermann
Resistenza batterica
2012/2013
1. Introduzione 1.1 Premessa Quando mi sono iscritta al lavoro di maturità nell’ambito della biologia medica, non sapevo esattamente quale aspetto di
questa materia approfondire. Il tema della biomedicina mi interessa molto, perché mi affascina sapere come il nostro
corpo lavora e funziona. Esso è una macchina complessa e “meravigliosa” dove le diverse strutture interagiscono per
mantenere una condizione di stabilità interna. Di fronte a situazioni di squilibrio, ad esempio la presenza di un organismo
estraneo o l’aumento della temperatura interna, il nostro organismo reagisce subito cercando di riportarci ad una
condizione di equilibrio. Nelle prime lezioni i professori hanno fatto delle piccole presentazioni sui possibili argomenti che
si potevano affrontare e a me hanno interessato in particolar modo gli argomenti riguardanti l’immunologia, l’emicrania e
la resistenza batterica. Inizialmente ero intenzionata ad approfondire il tema dell’immunologia, poiché mi incuriosisce
molto sapere come il nostro corpo riesce a combattere e a sconfiggere i diversi agenti patogeni mediante specifici
meccanismi di difesa. Discutendo con la mia docente responsabile ho però scartato questo tema siccome è un
argomento molto vasto e complesso. La mia attenzione è quindi caduta sull’argomento della resistenza batterica, ossia il
fenomeno per cui certi antibiotici sono inefficaci nei confronti di determinati batteri. In un certo senso questo tema è
collegato alla mia prima scelta, poiché nel momento in cui il nostro sistema di difesa non funziona come dovrebbe, o se
il battere è troppo forte per essere neutralizzato dai nostri anticorpi o linfociti T, gli antibiotici possono salvarci la vita, a
meno che i microbi non siano resistenti al farmaco. Ed è proprio sulla resistenza agli antibiotici, fenomeno in costante
crescita e di cui spesso si sente parlare, che ho deciso di focalizzare il mio lavoro di maturità.
1.2 Obiettivi Mediante una prima parte teorica intendo ampliare le mie conoscenze nell’ambito della resistenza batterica. Cosa sono
gli antibiotici, come agiscono sui batteri, quali sono i fattori che determinano la resistenza batterica e cosa accade a
livello molecolare quando un batterio diventa resistente, sono tutte questioni che intendo approfondire nella mia ricerca
bibliografica.
La parte sperimentale del mio lavoro di maturità, sotto proposta e supervisione della Dottoressa Demarta (collaboratrice
dell’Istituto Cantonale di Microbiologia di Bellinzona), ha l’obiettivo di ricercare specifici geni di resistenza in ceppi
batterici isolati dall’ambiente. I ceppi in esame sono enterobatteri prelevati dal Lago Cadagno, dalle acque del fiume
Ticino, prima e dopo il depuratore di Giubiasco, e dalle acque di scarico dell’ospedale San Giovanni di Bellinzona. La
presenza di questi geni può determinare la produzione di enzimi EBSL, delle β-lattamasi a spettro esteso, che rendono
inefficaci la maggioranza degli antibiotici di tipo β-lattamico (folto gruppo di antibiotici che comprende ad esempio le
penicilline e le cefalosporine). I geni ricercati, che conferiscono questa resistenza, sono mutazioni dei geni TEM, SHV e
OXA e il gene CTX-M. I ceppi produttori di ESBL rivestono particolare importanza clinica in quanto sono diffusi negli
ambienti ospedalieri e, a causa della loro multi resistenza, sono difficili da trattare [12,13].
I risultati ottenuti potranno, eventualmente, essere confrontati con quelli di un altro lavoro di maturità che ha preso in
considerazione batteri clinici, cioè batteri provenienti da pazienti d’ospedale [14]. Questo confronto potrebbe permettere
di tracciare la distribuzione dei diversi geni di resistenza per le ESBL in differenti ambienti.
5 Anna Zimmermann
Resistenza batterica
2012/2013
1.3 Ipotesi 1.3.1 Ipotesi 1 Per quanto concerne il fenomeno della resistenza, mi aspetto che sia più frequente nei batteri provenienti da ambienti in
cui vi è maggior presenza di antibiotici. Ipotizzo questo risultato in quanto il DNA è soggetto a mutazioni spontanee (si
calcola 1 mutazione ogni 1'000'000 scissioni), che possono anche determinare lo sviluppo di resistenze agli antibiotici. I
batteri resistenti, a contatto con gli antibiotici, ne sono selezionati positivamente e hanno così la possibilità di riprodursi e
di diffondersi. Questi batteri possono trasmettere i geni della resistenza ad altri batteri, tramite plasmidi, diffondendo così
ulteriormente il fenomeno della resistenza batterica [11].
I batteri in esame provengono dal lago Cadagno, dalle acque di scarico dell’Ospedale San Giovanni di Bellinzona e dalle
acque del fiume Ticino, prima e dopo il Depuratore di Giubiasco.
Per i batteri provenienti dal Lago Cadagno e dalle acque prelevate dopo il Depuratore di Giubiasco, mi aspetto di
riscontrare un livello di resistenza minore rispetto a quello dei batteri isolati dagli altri due ambienti. Il primo ambiente è
un lago naturale di alta montagna e dunque essenzialmente puro e privo di antibiotici. Nelle acque in uscita dal
depuratore non dovrebbero essere presenti batteri, in quanto vi vengono effettuati trattamenti meccanici, quali la
decantazione e la filtrazione, per eliminare ogni contaminazione batterica [8]. A tal fine non vi è impiego di antibiotici.
Presumo invece che nelle acque di scarico dell’ospedale e nelle acque prelevate dal fiume prima della stazione di
depurazione, la resistenza batterica sia molto più frequente. Nel primo caso mi aspetto più batteri resistenti, perché
all’ospedale si adoperano molto gli antibiotici. Nel secondo caso mi aspetto lo stesso risultato, perché la stazione di
depurazione è il luogo dove tutte le acque fognarie affluiscono, tra cui le acque dello scarico dell’ospedale, di case
farmaceutiche e di case private, e questi sono tutti luoghi in cui vi è, o vi può essere, utilizzo di antibiotici [4].
1.3.2 Ipotesi 2 Dal confronto dei risultati relativi alla frequenza di specifici geni di resistenza per le ESBL in ceppi clinici rispettivamente
ambientali, mi aspetto delle differenze. Le diverse condizioni ambientali possono esercitare delle pressioni selettive sui
batteri, favorendo quelli meglio adattati. I diversi geni di resistenza possono determinare caratteristiche differenti, che
possono favorire o sfavorire la vita dei batteri nei diversi ambienti. L’organismo umano è un ambiente diverso da quello
delle acque libere (laghi e fiumi); ad esempio nel nostro organismo i batteri sono confrontati con il sistema immunitario,
mentre in natura i batteri sono confrontati con altri fattori come le variazioni di temperatura. Le diverse caratteristiche
ambientali possono quindi selezionare batteri con diversi geni di resistenza [10].
6 Anna Zimmermann
Resistenza batterica
2012/2013
2. Svolgimento teorico 2.1 I Batteri 2.1.1 Informazioni generali I batteri appartengono al dominio Prokaryota (procarioti), che comprende i due regni Eubacteria (eubatteri) e
Archaebacteria (archibatteri)1. Tutti i procarioti sono organismi unicellulari, che presentano una cellula meno complessa
di quella degli eucarioti e di dimensioni minori. Gli archibatteri sono caratterizzati dal fatto che riescono a sopravvivere
negli ambienti più estremi, ad esempio negli ambienti molto caldi, o molto acidi, dove le condizioni impediscono lo
sviluppo di altre forme di vita. Comprendono i metanobatteri, gli alobatteri e i termoacidofili [11]. Gli eubatteri, che
rappresentano la maggior parte dei procarioti, colonizzano ambienti più ospitali e comprendono i cianobatteri (capaci di
fotosintesi) e i batteri (vedi schema 1).
Schema 1 Illustra la suddivisione del dominio procarioti
1
Classificazione di Cavalier-Smith, 2004 [11]
7 Anna Zimmermann
Resistenza batterica
2012/2013
2.1.2 La cellula batterica Le cellule batteriche presentano le seguenti strutture
(vedi figura 1): •
•
•
citoplasma: contenente il cromosoma, i
ribosomi e a volte i plasmidi;
involucro cellulare: costituito dalla membrana
cellulare, dalla parete cellulare e a volte da
una capsula esterna;
appendici facoltative: pili e flagelli.
2.1.2.1 Il citoplasma Figura 1 Rappresentazione della cellula batterica [3] La caratteristica principale dei batteri è di avere il DNA libero nel citoplasma, cioè non racchiuso all’interno di una
membrana nucleare [10]. Il materiale genetico di un battere è costituito da un unico cromosoma, che possiede all’incirca
3000 geni, che possono venir letti in entrambe le direzioni [10,3]. Questi microorganismi possono presentare nel loro
citoplasma anche dei plasmidi, cioè delle piccole molecole circolari di DNA separate dal cromosoma. Sui plasmidi sono
generalmente codificate, oltre alle istruzioni genetiche per la loro replicazione autonoma, delle informazioni facoltative
per la vita dei batteri. Ne esistono di vari tipi, tra cui i plasmidi R, che possono conferire resistenza a determinati
antibiotici (vedi cap. 2.3) [3]. I plasmidi possono essere trasferiti da un batterio all’altro attraverso il processo di
coniugazione (vedi cap. 2.1.3). La cellula procariota non presenta organuli nel citoplasma ad eccezione dei ribosomi,
che hanno la funzione di sintetizzare le proteine.
2.1.2.2 Involucro cellulare La cellula batterica ha una struttura esterna complessa, che può essere costituita da tre involucri. Il più esterno è la
capsula, che facilita l’adesione ad altre cellule e che può proteggere i batteri patogeni dalle difese immunitarie
dell’organismo ospite. La capsula è una struttura facoltativa. Il secondo è la parete cellulare, che svolge delle funzioni
molto importanti quali mantenere la forma della cellula batterica, impedirne la lisi in caso di esposizione ad un ambiente
ipotonico e fornirle protezione [3]. In base alla composizione chimica della parete si possono distinguere gli eubatteri
dagli archibatteri. I primi hanno la parete cellulare composta da una sostanza particolare chiamata peptidoglicano,
mentre gli archibatteri ne sono privi. Sempre in base alla composizione della parete cellulare, i batteri vengono suddivisi
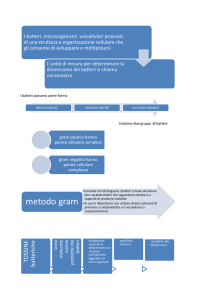
in batteri Gram positivi (Gram +) e Gram negativi (Gram -). Sostanzialmente i batteri Gram positivi hanno una parete
cellulare molto semplice con uno strato spesso di peptidoglicano. I Gram negativi hanno una parete più complessa,
8 Anna Zimmermann
Resistenza batterica
2012/2013
costituita da un sottile strato di peptidoglicano e uno strato di fosfolipidi, che costituisce un rivestimento semipermeabile.I
nomi Gram positivo e Gram negativo derivano dal
fatto che le pareti dei due tipi di batteri reagiscono
diversamente a contatto con un colorante. A
contatto con questa sostanza, i batteri Gram
positivi si colorano di blu-violetto, mentre quelli
negativi di rosso (colorazione di Gram, vedi figura
2). I batteri Gram negativi sono più pericolosi per
l’uomo, perché lo strato di fosfolipidi
semipermeabile nella parete cellulare ostacola
l’ingresso degli antibiotici nei batteri (vedi cap.
2.3.2) [3]. La terza struttura è la membrana
cellulare, presente in tutte le cellule dei viventi. Le
funzioni della membrana cellulare sono contenere
e delimitare il citoplasma, proteggere il contenuto
della cellula, regolare l’entrata e l’uscita delle
Figura 2 Fotografia di colonie batteriche sottoposte alla colorazione di Gram. Nell’immagine si possono vedere le due colorazioni: blu-­‐violetto sostanze grazie alla sua permeabilità selettiva,
per i Gram + e rosso per i Gram -­‐ (ICM)
conferire stabilità alla cellula e riconoscere, grazie
a delle proteine di superficie, sostanze presenti nell’ambiente esterno [11].
2.1.2.3 Appendici facoltative I pili sono delle appendici che permettono ai procarioti di attaccarsi gli uni agli altri, o di aderire a superfici esterne, ad
esempio a rocce o a pareti intestinali. Inoltre ci sono dei pili, detti pili sessuali, necessari per attuare la coniugazione
(vedi cap. 2.1.3). Questi ultimi presentano una lunghezza maggiore rispetto agli altri. Un altro tipo particolare di
appendici sono i flagelli, il cui compito è di consentire lo spostamento alla cellula batterica. Essi sono più lunghi dei pili
sessuali [3].
2.1.3 Modalità di scambio genetico I batteri hanno tre modi per scambiarsi materiale genetico. Il primo è la trasformazione, dove il battere acquisisce un
frammento di DNA estraneo dal liquido che lo circonda. Il secondo metodo è la trasduzione; in questo caso è necessario
l’intervento di un batteriofago, che ingloba casualmente un frammento di DNA batterico nel suo DNA durante
un’infezione. Quando il batteriofago infetta un altro batterio, il frammento di DNA assunto dall’infezione precedente,
viene introdotto nella cellula ospite. L’ultima tecnica di trasferimento di materiale genetico è la coniugazione, che
comporta lo scambio di plasmidi tra batteri. La coniugazione prevede che la cellula batterica donatrice, attraverso dei pili
sessuali, riesca ad attaccarsi alla cellula batterica ricevente. Dal momento in cui le due cellule si sono unite, gli strati
esterni si fondono creando un ponte citoplasmatico, attraverso il quale il DNA della cellula donatrice passerà, una volta
duplicato, alla ricevente. Il meccanismo che fa in modo che i frammenti di DNA estraneo si integrino nel DNA del battere
è simile al crossing-over, che avviene tra i cromosomi eucarioti: la parte del DNA che il battere ha ricevuto sostituisce
una parte del suo DNA [3].
9 Anna Zimmermann
Resistenza batterica
2012/2013
2.1.4 I batteri e l’uomo I batteri possono vivere liberi o in simbiosi con altri organismi. Tra i simbionti vi sono i batteri parassiti, che causano
malattie agli esseri viventi che li ospitano, tra cui anche l’uomo. La malattia può essere determinata da diversi fattori, ad
esempio dalla produzione di sostanze che possono facilitare l'invasione dei tessuti o la capacità di aggirare i meccanismi
di difesa dell'ospite. Tra queste ultime vi sono sostanze che compromettono l’attività delle cellule capaci di fagocitosi
(macrofagi) o che rendono i batteri non riconoscibili alle cellule deputate alla difesa. In questo modo, i batteri possono
invadere e distruggere i tessuti dell’organismo ospitante. Un altro fattore che rende i batteri patogeni è la secrezione di
tossine, cioè di sostanze tossiche per gli altri organismi. Esistono due tipi di tossine: le esotossine, che sono proteine
secrete dai batteri, e le endotossine, che sono lipopolisaccardidi del rivestimento esterno dei batteri Gram negativi [7,3].
Non tutti i batteri simbionti sono patogeni; vi sono anche delle specie innocue e altre persino utili o indispensabili
all’essere umano. I batteri intestinali della specie Escherichia coli, ad esempio, forniscono vitamine utili all’uomo [11]. Vi
sono anche batteri che sono sfruttati dall’uomo. Nel settore medico, ad esempio, batteri geneticamente modificati
vengono utilizzati per produrre insulina [10].
2.1.5 I batteri coliformi (Enterobatteri) I batteri coliformi, ad esempio Escherichia coli, Citrobacter freundii e le Klebsielle, appartengono alla famiglia degli
Enterobatteri (regno degli eubatteri) e sono bacilli Gram negativi. Generalmente l’habitat di questa famiglia di batteri è
l’intestino dell’uomo e degli altri animali. Alcuni di essi possono causare lievi o gravi infezioni all’ospite, a dipendenza del
germe e dell’organo colonizzato [1].
2.2 Gli antibiotici 2.2.1 Cosa sono gli antibiotici [1] Gli antibiotici sono sostanze naturalmente prodotte da microorganismi che si trovano nell’ambiente, utili per la loro
sopravvivenza. I batteri sono in competizione tra di loro e possono difendersi attraverso la produzione di queste
sostanze, che hanno un’azione antibatterica nei confronti di altre specie. La capacità di sintetizzare un determinato
antibiotico non è una caratteristica specie specifica: uno stesso antibiotico può essere prodotto da specie diverse e
microrganismi della stessa specie possono produrre uno o più antibiotici differenti. Nel settore medico, gli antibiotici sono
utilizzati per uccidere i batteri patogeni, nel caso l’uomo non sia in grado di eliminarli con le proprie difese naturali [2].
Questi hanno infatti delle proprietà che consentono di inibire e/o uccidere i batteri senza recare danni al nostro
organismo, perché interferiscono con delle funzioni metaboliche tipiche delle cellule procariote. I batteri, indeboliti o
uccisi da queste sostanze, vengono poi eliminati dal nostro sistema immunitario [1]. Gli antibiotici utilizzati nel settore
medico sono prodotti in laboratorio attraverso tecnologie diverse.
10 Anna Zimmermann
Resistenza batterica
2012/2013
2.2.2 I differenti tipi di antibiotici e le loro modalità d’azione [1,2] Gli antibiotici vengono distinti principalmente in sei famiglie in base alla loro struttura chimica: le penicilline, le
cefalosporine, le tetracicline, gli aminoglicosidi, i macrolidi e i fluorochinoloni. Vi sono poi altri agenti antibatterici
importanti, come ad esempio la Vancomicina e l’Aztreonam, che non rientrano nelle categorie sopra elencate. Come
detto in precedenza, tutti gli antibiotici sono sostanze in grado di ostacolare alcune vie metaboliche tipiche dei batteri,
inibendo la loro moltiplicazione (azione batteriostatica) e/o uccidendoli (azione battericida). Qui di seguito sono
presentate le modalità d’azione delle sei principali famiglie e dei due antibiotici Vancomicina e Aztreonam.
•
•
•
•
•
•
•
•
Le penicilline: hanno azione battericida. Interferiscono selettivamente con la sintesi del peptidoglicano,
componente della parete cellulare, bloccandone la produzione. Non hanno effetto se i batteri non sono in
condizione di riprodursi.
Le cefalosporine: hanno azione battericida. Come le penicilline, interferiscono nella produzione della
parete cellulare, bloccando la sintesi di peptidoglicano. Sono più efficaci nei confronti di batteri che
presentano resistenze ai β-lattamici (vedi cap. 2.2.3).
Le tetracicline: hanno azione batteriostatica. Inibiscono la sintesi delle proteine batteriche legandosi alla
sunbunità 30S dei ribosomi, bloccando l’accesso agli amino-acil-tRNA.
Gli aminoglicosidi: hanno azione battericida. Ostacolano la sintesi proteica dei batteri, ma a differenza
delle tetracicline, si legano irreversibilmente alla subunità ribosomiale 30S nella fase successiva
all’attacco dell’amino-acil-tRNA.
I macrolidi: hanno azione batteriostatica e battericida. Si legano alla subunità ribosomiale 50S in modo da
bloccare il processo della traslocazione durante la sintesi proteica.
I fluorochinoloni: hanno azione battericida. Inibiscono la replicazione del DNA batterico interferendo con
l’azione della DNA-girasi.
La Vancomicina: ha azione battericida. Inibisce la formazione dei fosfolipidi della parete batterica e anche
la polimerizzazione del peptidoglicano.
L’Aztreonam: ha azione battericida. Interferisce nella sintesi del peptidoglicano, componente della parete
cellulare, bloccandone la produzione.
2.2.3 I β-­lattamici [6] Figura 3 Anello tetratomico azetidinico [5] I β-lattamici costituiscono un folto gruppo di antibiotici, che ostacolano la sintesi
della parete cellulare dei batteri. Queste sostanze bloccano la produzione del
peptidoglicano, attraverso l’inibizione dell’enzima transpeptidasi. In generale sono
antibiotici più efficaci nei confronti dei batteri Gram positivi in quanto la loro parete
cellulare non presenta il rivestimento semipermeabile presente nei Gram negativi,
che ostacola l’ingresso agli antibiotici. Di questo gruppo di antibiotici fanno parte le
penicilline, le cefalosporine, l’Aztreonam e altre sostanze. Strutturalmente gli
antibiotici β-lattamici sono caratterizzati da un anello tetratomico azetidinico,
chiamato anello β-lattamico (vedi figura 3). I diversi β-lattamici differiscono nella loro
struttura chimica e nello spettro d’azione. Qui di seguito sono approfondite alcune
caratteristiche relative alle penicilline e alle cefalosporine. Le penicilline vengono
classificate secondo diverse caratteristiche.
Se si considera la loro origine si possono distinguere:
11 Anna Zimmermann
•
•
Resistenza batterica
2012/2013
le penicilline naturali, ad esempio la Penicillina G. Quest’ultima è la prima sostanza antibatterica scoperta
(nel 1928 da Alexander Fleming), ed è l’antibiotico naturale più usato in campo medico. È attiva contro la
maggior parte dei batteri Gram positivi, e ha effetto sui ceppi Gram negativi solo se utilizzata in dosi
elevate; le penicilline semisintetiche, ad esempio le meticilline, l’Oxacillina, e la Cloxacillina. Sono prodotte da
colture biologiche di Penicillium e modificate chimicamente. Agiscono principalmente sui batteri di tipo
Gram positivi. Tra queste vi sono anche penicilline ad ampio spettro, come le ampicilline, che sono attive
anche contro i batteri Gram negativi, grazie alla loro maggiore capacità di penetrazione nelle cellule
batteriche [1].
Le cefalosporine sono generalmente classificate in base al loro spettro d’azione. In questo caso si distinguono
cefalosporine di prima, seconda, terza e quarta generazione [2]:
•
•
•
•
le cefalosporine di prima generazione sono attive soprattutto nei confronti di microrganismi Gram positivi,
mentre sui Gram negativi hanno un’azione limitata;
le cefalosporine di seconda generazione hanno un efficacia maggiore rispetto a quelle di prima
generazione nei confronti di batteri Gram negativi (ad esempio Cefoxitina e Cefuroxime);
le cefalosporine di terza generazione hanno un’attività ancora maggiore sui batteri Gram negativi in
confronto alle cefalosporine di seconda generazione (ad esempio Cefpodoxime, Cefotaxima e Ceftriaxone);
le cefalosporine di quarta generazione sono efficaci sia nei confronti dei batteri Gram negativi che contro i
Gram positivi (ad esempio Cefepime).
Un altro farmaco β-lattamico è l’acido clavulanico, che ha una modesta funzione antibatterica ma è molto efficace nel
caso di batteri resistenti ai beta-lattamici. L’acido clavulanico può essere usato in combinazione alle penicilline [1].
2.3 La resistenza agli antibiotici 2.3.1 Che cosa è la resistenza agli antibiotici [1] Un battere si dice resistente quando è in grado di sopravvivere e moltiplicarsi nonostante la presenza di un antibiotico.
La resistenza è stabilita in relazione alla concentrazione dell’antibiotico: un microrganismo è detto resistente se una
specifica concentrazione di un antibiotico è in grado di inibire o uccidere altri batteri, mentre non ha effetto sulle sue
funzioni metaboliche vitali. Una tecnica che permette di riconoscere se un battere è resistente è il test
dell’antibiogramma (vedi cap. 3.2.1).
2.3.2 Cosa determina lo sviluppo della resistenza [1] Vi sono essenzialmente due fenomeni che possono determinare la resistenza batterica agli antibiotici. Il primo consiste
in una mutazione spontanea a livello del cromosoma, che può determinare lo sviluppo di geni di resistenza. Tra i geni di
resistenza vi sono ad esempio sequenze che determinano una ridotta permeabilità della parete cellulare agli antibiotici
(vedi cap 2.3.3). Il secondo fenomeno consiste nel trasferimento di geni di resistenza da un battere all’altro mediante
12 Anna Zimmermann
Resistenza batterica
2012/2013
coniugazione o, meno frequentemente, via trasduzione. Il trasferimento della resistenza tra due batteri è facilitato dal
fatto che alcuni geni di resistenza sono localizzati su trasposoni2.
2.3.3 I tipi di resistenza [9] La resistenza agli antibiotici è conferita da geni di resistenza attraverso diversi meccanismi, ad esempio:
•
•
•
•
l’alterazione della permeabilità della parete cellulare. Una permeabilità minore della parete cellulare può
limitare o impedire l’accesso dell’antibiotico al suo interno e rendere così il battere insensibile al farmaco
[5];
l’aumentato efflusso. L’antibiotico penetrato nella cellula batterica può essere allontanato attraverso
sistemi di efflusso che determinano l’espulsione del farmaco;
la ridotta affinità per il bersaglio. L’alterazione del bersaglio dell’antibiotico, ad esempio una sua modifica
enzimatica, può far si che questo non venga più riconosciuto dal farmaco. Ad esempio, la DNA girasi è il
bersaglio di alcuni antibiotici chiamati chinoloni; la modifica di un unico amminoacido in questa proteina
determina la resistenza a questa famiglia di antibiotici;
la produzione di enzimi inattivanti l’antibiotico. Questi enzimi hanno la capacità di modificare
chimicamente determinati legami presenti nelle molecole di uno o più antibiotici, annullandone l’effetto.
2.3.4 Le β-­lattamasi di tipo ESBL3 [6] Tra gli enzimi che determinano la resistenza agli antibiotici vi sono le β-lattamasi. Le β-lattamasi sono delle proteine in
grado di inattivare antibiotici di tipo β–lattamico, idrolizzando il legame amidico dell’anello β-lattamico (spezzandone cioè
il legame C-N). Questi enzimi possono essere ad ampio spettro, quindi attivi contro molti β-lattamici, o essere più
specifici, ed agire solo nei confronti di alcuni di essi. Le ESBL sono enzimi in grado di inattivare diversi β–lattamici, tra
cui le penicilline, le cefalosporine di II e III generazione e l’Aztreonam. Le ESBL sono enzimi inibitore-sensibili, cioè
enzimi che possono essere inibiti dalla presenza di sostanze specifiche. Un inibitore delle ESBL è ad esempio l’acido
clavulanico. I geni più frequenti, che codificano per questi enzimi, sono delle mutazioni dei geni TEM, SHV e il gene
CTX-M. Per i primi due geni esistono più di 100 varianti date da diverse mutazioni, che generalmente sono localizzate
sui plasmidi. Le varianti dei geni SHV e TEM possono essere presenti anche sul cromosoma. Per il gene CTX-M si
conoscono solo 30 varianti, localizzate sia su plasmidi che sul cromosoma. Ci sono altri geni che conferiscono questa
resistenza, ma sono meno frequenti, ad esempio le mutazioni del gene OXA. Anche questi geni possono trovarsi sia sui
plasmidi che sul cromosoma. Il fatto che questi geni siano presenti sui plasmidi, ne facilita il trasferimento tra ceppi
batterici. Vi sono altri batteri che possono presentare questo tipo di resistenza ad ampio spettro, ma essa è
maggiormente diffusa nella famiglia degli Enterobatteri. I ceppi ESBL sono molto diffusi nell’ambiente ospedaliero,
soprattutto in batteri patogeni come le Klebsielle. La presenza di queste resistenze rende difficile l’eliminazione dei
patogeni, in quanto restano pochi antibiotici efficaci. Uno di questi è la Vancomicina, attualmente molto importante nel
settore medico, poiché è in grado di uccidere ceppi resistenti ai β-lattamici. A causa della loro importanza clinica, i
batteri produttori di ESBL sono oggetto di approfondimento scientifico, ad esempio all’Istituto Cantonale di Microbiologia
a Bellinzona (ICM) [13].
Si tratta di elementi genetici che possono spostarsi nel genoma di un organismo; nel caso dei batteri in diverse posizioni nel cromosoma o su
plasmidi [10]. 3 -lattamasi a spettro esteso
2
13 Anna Zimmermann
Resistenza batterica
2012/2013
3. Svolgimento pratico 3.1 Materiale Agar Sangue (Dickinson, PA-254005.04)
Terreno per antibiogrammi (Mueller Hiton agar, BioMérieux, CM 51860)
Anse sterili (Sarstedt, 86.1562.050)
Becco Bunsen
Autoclave
Tryptic Soy Broth o TSB (OXOID, CM 0129)
NaCl (Fluka, 71376)
Micropipette Gilson
Strumento per misurare la torbidità in Mcfarland
Soluzione standard 0.5 Mcfarland
Vortex (Genie2, G560E)
Bastoncini di ovatta (Hartmann, 967 936/9)
Stampigliatore
Pinzette
Dischetti di antibiotici con Ampicillina, Cefoxitina, Cefuroxima, Cefpodoxima, Augmentin, Ceftriaxone, Cefotaxima,
Aztreonam, Cefepime (Becton, Dickinson)
InstaGene Matrix (Biorad, 732-6030)
Tubetti Eppendorf
Centrifuga (Eppendorf, 5417 R)
Frigorifero
Nucleo Bond PC20 (Macherey-nagel, 740571.100)
Isopropanolo
Etanolo
Acqua
Primers CTX-f, CTX-r, OXA-r, OXA-f , SHV-r, SHV-f, TEM-c, TEM-h (Microsynth)
Master Mix (Qiagen, 201443)
Termocycler Veriti (AB Prism, 9902)
Cappa UV
Agarosio (Eurobio, GEPAGA0764)
TBE (Fluka, 93349)
Acido Borico (Fluka, 15663 / B0252)
EDTA (Fluka, 03677)
Gel Red (Biotium)
Loading Buffer
Marker Peso molecolare (Roche,11721933001)
Microonde
Pettini
Lampada UV
Ceppi batterici: 38 ceppi batterici ambientali e ceppo 23/07 + ESBL
14 Anna Zimmermann
Resistenza batterica
2012/2013
I 38 ceppi utilizzati (vedi tabella 1) sono batteri coliformi isolati da quattro diversi ambienti (Lago Cadagno, acque di
scarico dell’Ospedale San Giovanni, acque del fiume Ticino prima e dopo il depuratore di Giubiasco). Questi sono stati
selezionati da 250 organismi inizialmente campionati, attraverso un terriccio per far crescere e riprodurre solamente i
microorganismi potenzialmente produttori di ESBL4.
Provenienza
Battere
Ceppo
HWW-OSG
E. coli5
55SG-CF
HWW-OSG
E. coli
56SG-CF
HWW-OSG
E. coli
58SG-CF
LEGENDA:
HWW-OSG
C.
freundii6
66SG-CF
HWW-OSG
E. cloacae7
67SG-CF
HWW-OSG
E. coli
80SG-CF
HWW-OSG
E. coli
85SG-CF
AS-DG
C. freundii
106DG-CF
AS-DG
C. freundii
108DG-CF
AS-DG
E. cloacae
109DG-CF
AS-DG
E. coli
110DG-CF
HWW: Hospital Waste Water
AS: Activated Sludge
SW: Superficial Water
TicR: Ticino River
LC: Lago Cadagno FDD: Fiume Dopo Depuratore di Giubiasco
DG: fiume prima Depuratore di Giubiasco
AS-DG
E. cloacae
115DG-CF
OSG: Ospedale San Giovanni
AS-DG
E. cloacae
117DG-CF
AS-DG
E. cloacae
120DG-CF
pneumonia8
AS-DG
K.
AS-DG
E. cloacae
123DG-CF
AS-DG
E. cloacae
124DG-CF
AS-DG
E. cloacae
125DG-CF
AS-DG
C. freundii
126DG-CF
AS-DG
E. coli
130DG-CF
AS-DG
E. coli
131DG-CF
AS-DG
E. coli
133DG-CF
AS-DG
E. coli
135DG-CF
oxytoca9
122DG-CF
AS-DG
K.
AS-DG
E. cloacae
141DG-CF
AS-DG
E. cloacae
143DG-CF
AS-DG
C. f reundii
147DG-CF
AS-DG
K. oxytoca
150DG-CF
SW-TicR-FDD
E. cloacae
157FDD-CF
SW-TicR-FDD
C. freundii
162FDD-CF
SW-TicR-FDD
E. coli
172FDD-CF
SW-TicR-FDD
C. freundii
183FDD-CF
SW-TicR-FDD
C. freundii
185FDD-CF
SW-TicR-FDD
E. coli
189FDD-CF
136DG-CF
La selezione è stata effettuata attraverso piastre cromogene per la selezione di batteri ESBL (chromID ESBL, Biomerieux, Ref 43481). Le piastre
contengono un antibiotico -lattamico che, se degradato dalle ESBL, cambia colore. La selezione è stata effettuata dall’équipe dell’ICM.
5 Escherichia coli
6 Citrobacter freundii
7 Enterobacter cloacae
8 Klebsiella pneumoniae
9 Klebsiella oxytoca 4
15 Anna Zimmermann
Resistenza batterica
SW-TicR-FDD
C. f reundii
197FDD-CF
SW-LC
E. coli
213LC-CF
SW-LC
E. cloacae
248LC-CF
2012/2013
Tabella 1 Illustra la provenienza, la specie e il nome dei ceppi batterici ambientali analizzati nell’esperimento Il ceppo 23/07 + ESBL, che è stato testato in precedenza dall’équipe dell’ICM, è produttore di ESBL e contiene tre geni
caratteristici della resistenza ricercata.
3.2 Procedimento L’identificazione di batteri con geni per le ESBL avviene tramite diversi esperimenti: la crescita di batteri su terreno
selettivo (eseguito in precedenza per la selezione dei 38 ceppi), l’antibiogramma (ricerca di sinergie), la PCR ( ricerca di
geni di resistenza) e il sequenziamento dei geni di resistenza (non eseguito in questo lavoro).
3.2.1 Preparazione dei 38 ceppi all’antibiogramma e antibiogramma Le operazioni descritte in seguito sono state eseguite in condizione di
sterilità, vicino a un becco Bunsen. I 38 ceppi sono stati scongelati
dall’equipe dell’ICM e personalmente inoculati con la modalità dello
strisciamento per settori10 nel terriccio agar sangue. Lo strisciamento per
settori viene eseguito in tre fasi. Si effettua un primo strofinamento fitto che
arrivi all’incirca a metà della capsula in modo da scaricare l’ansa. L’ansa
deve percorrere tutto il diametro della capsula. Per il secondo strisciamento
è necessario girare la capsula di 60 gradi e con l’ansa si esegue uno
sfregamento leggero, disegnando una linea a zig-zag. Infine, per il terzo e Figura 4 Fotografia che illustra la crescita del ultimo strisciamento, si compie una nuova rotazione sempre di 60 gradi e si ceppo E.coli 189FFD inoculato secondo la tecnica dello strisciamento per settori (ICM) esegue lo sfregamento come il precedente (vedi figura 4). Le capsule sono
poi state messe in incubazione a 37°C per una notte. L’agar sangue è un terreno composto da sostanze altamente
nutritive, che permette a tutti i batteri di crescere e prosperare, a differenza di un terreno selettivo, che consente solo a
determinati batteri di moltiplicarsi. È fondamentale eseguire questo passaggio per avere microrganismi freschi per
eseguire gli antibiogrammi. I risultati possono così essere interpretati con le tabelle di riferimento, ottenute con batteri
nelle stesse condizioni. Se i batteri fossero ad esempio morti, l’esito del test potrebbe essere falsato. Una volta cresciuti,
i batteri sono stati inoculati in un terreno liquido, in questo caso un brodo di arricchimento standard (TSB). A tal fine sono
state prelevate 2-3 colonie batteriche con un’ansa sterile e sono state stemperate in 2 ml di brodo. Dopo questa
operazione le provette sono state messe in incubazione a 37°C per favorire la moltiplicazione batterica. Secondo i
metodi standard [4], l’inoculo che viene utilizzato per l’antibiogramma deve avere una concentrazione di batteri non
superiore a 0.5 McFarland11 (circa 108 UFC12/ml).
Per ottenere questa concentrazione, dopo circa 3 ore d’incubazione, le colture batteriche sono state diluite con una
soluzione 0.9% di cloruro di sodio, che mantiene le cellule vitali. Esistono due metodi che permettono di ottenere la
Una possibile tecnica per l’inoculazione su piastra dei batteri.
È l’unità di misura che si utilizza per misurare la concentrazione dei batteri. Indica quante cellule batteriche vi sono per ml di sospensione in base
alla sua torbidità.
12 Unità Formanti Colonie 10
11
16 Anna Zimmermann
Resistenza batterica
2012/2013
giusta concentrazione di batteri. Il primo, non molto preciso, consiste nell’inserire i batteri nella soluzione salina e
contemporaneamente confrontare l’opacità della miscela che si ottiene con quella di una provetta di controllo 0.5
McFarland, fino al punto in cui le provette presentano la stessa torbidità. La seconda tecnica utilizza una macchina
capace di misurare esattamente la concentrazione dei batteri, sempre in McFarland, man a mano che i batteri vengono
introdotti nella provetta. In questo esperimento è stato utilizzato prevalentemente il primo metodo, per alcuni ceppi è
stato utilizzato il secondo. Raggiunta la giusta concentrazione, i batteri sono stati inoculati nelle capsule per
l’antibiogramma. A tal fine è stato preso un batuffolo di ovatta sterile ed è stato immerso nella sospensione batterica. Il
batuffolo impregnato di batteri è stato strisciato per 3 volte in modo fitto, girando per ogni strisciamento la capsula di 60
gradi. Infine è stato passato il batuffolo sul margine interno della capsula. A questo punto le capsule sono state lasciate
riposare per una decina di minuti e in seguito sono stati inseriti, utilizzando una pinzetta sterile e a volte lo
stampigliatore13, i 9 dischetti di antibiotici. Sono stati inseriti i seguenti antibiotici: Ampicillina (AM, penicillina
semisintetica), Cefoxitina (FOX, cefalosporina di II generazione), Cefuroxime (CX, cefalosporina di II generazione),
Cefpodoxima (CPD, cefalosporina di III generazione), Augmentin (AMC, contiene Amoxicillina, penicillina semisintetica e
acido clavulanico, inibitore ESBL), Ceftriaxone (CRO, cefalosporina di III generazione), Cefotaxima (CTX, cefalosporina
di III generazione), Aztreonam (ATM, monobattame14), Cefepime (FEB, cefalosporina di IV generazione). I dischetti sono
stati inseriti nella sequenza indicata sopra. Questo permette di osservare l’eventuale presenza di sinergie o di
antagonismi ricercati. Le sinergie sono presenti quando due antibiotici a contatto producono un effetto maggiore sui
batteri rispetto all’effetto che avrebbero se fossero presi singolarmente. Gli antagonismi consistono nel fenomeno
inverso, cioè quando due antibiotici a contatto presentano un effetto minore che se da soli. I dischetti sono stati messi ad
una distanza reciproca maggiore ai 24 mm, per impedire interferenze tra i vari antibiotici. Le piastre sono state messe in
incubazione alla temperatura di 37°C per una notte. Il giorno dopo sono state analizzate per determinare le resistenze
dei ceppi analizzati. Per stabilire se il battere è sensibile o resistente ad un dato antibiotico ci si è basati sui valori di
riferimento, che indicano il diametro dell’alone d’inibizione di crescita attorno ai diversi dischetti (vedi tabella 2) . Sono
poi state cercate eventuali sinergie e antagonismi, visibili attraverso variazioni delle forme degli aloni di inibizione.
13
14
Si tratta di una macchina nella quale si mettono i dischetti d’antibiotico, che devono essere applicati alla piastra.
Nome della famiglia di antibiotici di cui fa parte l’Aztreonam. 17 Anna Zimmermann
Antibiotico
Resistenza batterica
Concentrazione
dell’antibiotico
applicata al
dischetto (µg)
Diametro
dell’alone
< Resistente
2012/2013
Diametro
dell’alone
≥ Sensibile
1
Ampicillina (AM)
10
14 mm
14 mm
2
Cefoxitina (FOX)
30
19 mm
19 mm
3
Cefuroxime (CX)
30
18 mm
18 mm
4
Cefpodoxime (CPD)
10
21 mm
21 mm
5
Ceftriaxone (CRO)
30
20 mm
23 mm
6
Augmentin (AMC)
20/1015
17 mm
17 mm
7
Aztreonam (ATM)
30
24 mm
27 mm
8
Cefepime (FEB)
30
21 mm
24 mm
9
Cefotaxima (CTX)
5
18 mm
21 mm
Tabella 2 Indica per i diversi antibiotici utilizzati (colonna 1), la concentrazione dell’antibiotico sul dischetto (colonna 2), le dimensioni dell’alone d’inibizione nel caso il ceppo sia resistente all’antibiotico (terza colonna) e nel caso sia sensibile (quarta colonna). I valori si riferiscono agli enterobatteri. Per verificare se i batteri possiedono i geni per la produzione delle ESBL, è stata osservata la loro risposta fenotipica. In
caso positivo, nell’antibiogramma deve evidenziarsi una sinergia tra gli antibiotici Ceftriaxone, Augmentin e Cefotaxima e
una resistenza per l’antibiotico Cefoxitina. Il riscontro di queste due caratteristiche è il fattore più importante. I risultati per
gli altri cinque antibiotici, benché non siano determinanti per la ricerca di batteri ESBL positivi, sono comunque stati
raccolti. La risposta fenotipica ricercata è spiegata di seguito. Gli antibiotici Ceftriaxone e Cefotaxima sono cefalosporine
di III generazione. Augmentin contiene l’antibiotico Amoxicillina, che fa parte delle penicilline semisintetiche, e l’acido
clavulanico, un inibitore degli enzimi ESBL. La Cefoxitina fa parte delle cefalosporine di II generazione. I quattro
antibiotici presi in considerazione sono suscettibili agli enzimi ESBL, quindi in loro presenza vengono degradati e il
battere risulta resistente. La presenza di acido clavulanico inibisce però l’attività di questi enzimi e di conseguenza, se i
batteri sono ESBL positivi, si manifesta un livello minore di resistenza ai tre antibiotici e si ha la sinergia. Per evidenziare
la sinergia ricercata, il dischetto con l’antibiotico Augmentin deve essere messo tra gli altri due in quanto contiene
l’inibitore acido clavulanico. Nelle capsule la sinergia si manifesta nel seguente modo: gli aloni esterni non appaiono
come cerchi perfetti, ma presentano una sagoma ovale in cui la parte rivolta all’antibiotico centrale è più grande (vedi
Figura 8). Con l’antibiogramma non si identificano tutti i ceppi aventi i geni della resistenza per le ESBL. È possibile
infatti che alcuni batteri lo possiedano, ma che questo non venga espresso a causa della presenza di un altro gene,
l’AmpC. Il prodotto del gene AmpC, un’altra β-lattamasi a spettro esteso, non viene inibito dall’acido clavulanico e quindi
non è visibile la sinergia. I batteri aventi i geni per le ESBL, che non vengono identificati tramite l’antibiogramma, sono
detti “falsi negativi”. Per verificare se i batteri contengono il gene AmpC vi sono diverse procedure. Si possono ad
esempio usare delle piastre Mueller Hinton (MH) dove nel terreno viene aggiunto l’antibiotico Cloxacillina (una penicillina
semisintetica), che è in grado di inibire il gene AmpC. In pratica si fanno degli antibiogrammi con un unico dischetto
dell’antibiotico Cefoxitina che viene degradata dal prodotto dell’AmpC, sia su piastre MH normali che su piastre MH con
Cloxacillina. In caso di presenza del gene AmpC, i diametri di inibizione che si andranno a leggere sulle due capsule,
dovranno essere diversi. Nella capsula con la Cloxacillina, l’alone dovrà essere almeno 5 mm più grande rispetto a
quello misurato nella capsula senza l’antibiotico. Un'altra tecnica per verificare se il battere possiede il gene AmpC
consiste nel ricercarne direttamente il gene, cioè svolgere lo stesso lavoro presentato in seguito per i geni TEM, SHV,
OXA e CTX-M. Può anche capitare che un battere possieda il gene AmpC, ma che questo non venga espresso. In
15
20 mg Ampicillina e 10 mg Acido Clavulanico
18 Anna Zimmermann
Resistenza batterica
2012/2013
questo caso l’acido clavulanico è ancora in grado di inibire le di ESBL. In altre parole il gene è presente nel genoma del
battere ma visto che è inattivo è come se non ci fosse. L’analisi dei “falsi positivi” è stata eseguita in un secondo tempo
dall’équipe dell’ICM.
3.2.2 Estrazione di DNA totale dal ceppo di controllo 23/07 + ESBL Il ceppo di controllo produce ESBL e contiene tre dei quattro geni di resistenza ricercati (TEM, SHV e CTX-M). Lo scopo
di questa estrazione è quello di avere a disposizione del DNA, con il quale controllare il corretto funzionamento del
termociclatore16. Capita infatti spesso che con la PCR17 si ottengano dei risultati errati. L’estrazione del DNA totale di un
battere consiste nell’estrarne sia il cromosoma che i plasmidi. Alcune colonie del ceppo 23/07 + ESBL (2/3 colonie) sono
state prelevate con un’ansa sterile e stemperate in 200 µl di InstaGene Matrix in un tubetto Eppendorf da 1,5 ml. Questa
matrice, a contatto con i batteri, ne causa la lisi e quindi la liberazione del DNA. In seguito il tubetto è stato messo
sull’agitatore Vortex per 15 secondi, per sciogliere bene le colonie ed è stato scaldato per 10 minuti alla temperatura di
95°C, per facilitare la lisi cellulare. Al termine del riscaldamento è stato utilizzato nuovamente l’agitatore Vortex per 15
secondi, sempre per sciogliere bene le colonie e avere una miscela omogenea. Il campione è poi stato centrifugato per
10 minuti a 13000 rpm al fine di far precipitare tutto il materiale di scarto, tra cui le proteine e i residui delle pareti e delle
membrane cellulari batteriche. Il DNA rimasto in sospensione è stato conservato a -20°C.
3.2.3 Estrazione dei plasmidi dai 38 ceppi d’interesse Per l’estrazione dei plasmidi da ogni ceppo è stato usato un apposito kit (kit Nucleobond), nel quale sono contenuti tutti i
materiali e le spiegazioni necessarie per l’esecuzione del test. Tramite questo kit è possibile estrarre tre diverse quantità
di DNA – Mini (AX18 20), Midi (AX 100) e Maxi (AX 500) – a dipendenza delle quantità di tamponi che si utilizzano. In
questo esperimento sono state osservate le indicazioni per un’estrazione Mini in quanto per il procedimento successivo,
la PCR, è sufficiente una piccola quantità di DNA. Il primo passo per l’estrazione dei plasmidi è stato quello di
stemperare 2-3 colonie batteriche in 400 µl di Buffer S1+RNase A in un tubetto da 1,5 ml. Questa miscela di sostanze
permette di risospendere le cellule. In seguito sono stati aggiunti 400 µl di Buffer S2 che, oltre ad avere delle proprietà
tamponanti (quindi minimizzanti le variazioni di pH), permette la lisi delle cellule batteriche. Il campione è poi stato
mescolato per inversione molto lenta, per non creare danni al DNA. Questo movimento è stato ripetuto per 15 volte e poi
la miscela è stata incubata a 18°- 25°C ( temperatura ambiente) per 2-3 minuti. Il passo successivo è stato l’aggiunta di
400 µl della soluzione Buffer S3, in precedenza conservata nel frigo a 4°C, per far precipitare gli scarti dovuti alla lisi
batterica. Gli scarti sono costituiti di proteine, residui della parete e della membrana cellulare. Il tutto è stato di nuovo
mescolato per inversione 15 volte, fino ad ottenere una sospensione omogenea. A questo punto il DNA cromosomiale
ha iniziato a precipitare, visibile sotto forma di filamenti biancastri. Per facilitare tale processo la miscela è stata messa
nel ghiaccio per 5 minuti; in seguito è stata centrifugata a 12,000 x g per 5-10 minuti alla temperatura di 4°C, per far
depositare gli scarti della lisi e il DNA cromosomiale. Il passo finale è stato l’isolazione del DNA plasmidico attraverso un
NucleoBond®. Si tratta di una colonnina in cui c’è una resina, che dopo attivazione tramite il contatto con il Buffer N219,
riesce a trattenere i plasmidi isolandoli dal resto della soluzione. Il surnatante ottenuto dalla centrifugazione è stato
introdotto nella colonnina. Questa è stata poi lavata con 3 ml di Buffer N3 per eliminare eventuali impurità. I plasmidi
Il termociclatore è la macchina che esegue l’amplificazione del DNA via Reazione Polimerasica a Catena o PCR.
Reazione Polimerasica a Catena, che permette l’amplificazione del DNA.
18 La sigla AX serve per l’identificazione della colonnina Nucleobond® da utilizzare nei diversi casi. 19
Determina un cambiamento del pH che permette la formazione di legami chimici tra resina e DNA. 16
17
19 Anna Zimmermann
Resistenza batterica
2012/2013
sono stati recuperati dalla resina attraverso l’eluizione con 1 ml di Buffer N5 in un tubetto da 2 ml. Il DNA plasmidico in
soluzione è stato fatto precipitare con 750 µl di isopropanolo. Il campione è stato in seguito centrifugato a 15,800 x g per
30 minuti sempre alla temperatura di 4°C ed il surnatante è stato allontanato dal pellet contenente l'estratto plasmidico.
Successivamente sono stati aggiunti 500 µl di etanolo al 70% per disidratare il DNA e il tubetto è stato centrifugato
nuovamente a 16,100 x g a 18-25°C (temperatura ambiente) per circa 30 minuti. Infine il DNA del pellet è stato
risospeso in 40 µl d’acqua.
3.2.4 Preparazione dei campioni alla PCR e PCR per i geni TEM, SHV, CTX-­M e OXA Sono stati effettuati due tipi di PCR: la PCR normale, che
amplifica un unico gene, e la PCR Multiplex, che amplifica
diversi geni contemporaneamente. In questo esperimento è
stata utilizzata la PCR normale per amplificare i geni CTX-M
e OXA. Per i geni TEM e SHV è stata effettuata la PCR
Multiplex, in quanto le condizioni di temperatura per
l’ibridazione dei primers per questi due geni sono molto
simili.
Innanzitutto sono stati scongelati i primers CTX-f, CTX-r,
OXA-r, OXA-f, SHV-r, SHV-f, TEM-c, TEM-h, le cui
sequenze nucleotidiche sono illustrate nella figura 5.
Figura 5 sequenze nucleotidiche dei primers [5] 3.2.4.1 Preparazione dei campioni per la PCR normale per i geni CTX-­M e OXA Per l’amplificazione di ogni gene sono stati preparati diversi campioni, seguendo le indicazioni sulle quantità riportate
sotto. Per il gene OXA sono stati preparati in totale 41 campioni: 38 campioni per i ceppi batterici in esame, 1 campione
per il controllo negativo (invece del DNA plasmidico sono stati utilizzati 2,5 µl d’acqua) e 2 campioni per il controllo
positivo, con DNA proveniente dal ceppo ESBL + 23/07 (uno con 2,5 µl DNA plasmidico e uno con 2,5 µl DNA
genomico). Per il gene CTX-M sono stati preparati 40 campioni: 38 campioni per i ceppi batterici in esame, 1 campione
per il controllo negativo (invece del DNA plasmidico sono stati utilizzati 2,5 µl d’acqua) e un solo campione per il
controllo positivo (con 2,5 µl di DNA plasmidico 23/07 + ESBL).
Composizione campioni per la PCR normale
12,5 µl Master Mix
0,75 µl primer-f
0,75 µl primer-r
8,5 µl H2O
2,5 µl DNA plasmidico
La preparazione dei campioni è stata effettuata sotto cappa, al fine di evitare qualsiasi tipo di contaminazione. Per la
preparazione di ogni campione è stata prelevata prima la sostanza necessaria in quantità maggiore, quindi la soluzione
20 Anna Zimmermann
Resistenza batterica
2012/2013
Master Mix, poi sono state aggiunte le altre sostanze secondo quantità decrescente e per finire è stato inserito il DNA,
cromosomico o plasmidico.
3.2.4.2 Preparazione dei campioni per la PCR multiplex per i geni TEM e SHV Per l’amplificazione dei geni TEM e SHV sono stati preparati, seguendo le indicazioni sulle quantità riportate sotto
rispettivamente 40 e 41 campioni: 38 campioni per i ceppi batterici, 1 campione per il controllo negativo (invece del DNA
plasmidico sono stati utilizzati 2,5 µl d’acqua) e 1 campione per il controllo positivo (con 2,5 µl di plasmidico del ceppo
23/07 + ESBL). Per il gene SHV è stato preparato anche un secondo controllo positivo con DNA genomico del ceppo
23/07+ESBL. I campioni per la PCR multiplex sono stati preparati seguendo le stesse indicazioni riportate per la PCR
normale ma con quantità diverse delle diverse sostanze.
Composizione campioni per la PCR Multiplex
12,5 µl Master Mix
2,5 µl mix primers
7,5 µl H2O
2,5 µl DNA plasmidico
3.2.4.3 Amplificazione del DNA I campioni sono stati introdotti nel termociclatore, cioè la macchina dove i geni vengono amplificati. Le condizioni applicate per l’ampliamento dei geni TEM e SHV mediante la PCR multiplex sono state le seguenti.
La fase di denaturazione iniziale, che ha permesso di attivare la Taq polimerasi presente nella Master Mix, è stata di 15
minuti a 95°C. In seguito sono stati impostati 32 cicli comprendenti una denaturazione di 30 secondi a 94°C,
un’ibridazione dei primers a 54°C per 30 secondi, un’elongazione da parte della Taq polimerasi a 72°C per 1 minuto e
un’elongazione finale a 72°C per 10 minuti . Le stesse condizioni sono state applicate anche per l’ampliamento del gene
OXA mediante PCR normale. Le condizioni a cui sono stati sottoposti i campioni per l’amplificazione del gene CTX-M
sono state invece le seguenti. La fase di denaturazione iniziale è stata di 5 minuti alla temperatura di 95°C.
Successivamente sono stati impostati 35 cicli comprendenti una denaturazione di 1 minuto a 95°C, un’ibridazione dei
primers a 58°C per 1 minuto, un elongazione della Taq polimerasi a 72°C per 1 minuto e un elongazione finale a 72°C
per la durata di 7 minuti. In un secondo tempo è stata effettuata la PCR normale anche per i geni TEM e SHV, in quanto
la PCR Multiplex ha dato esiti errati20 . Queste due amplificazioni sono stata effettuate dall’équipe dell’ICM.
3.2.5 Preparazione dei campioni per elettroforesi su gel e elettroforesi su gel Inizialmente è stato preparato il gel di agarosio. Per la realizzazione del gel sono stati pesati 0,75 g di agarosio e sciolti
in 50 ml di TBE 1x. Il TBE è un tampone di corsa che serve a mantenere il valore del pH intorno all'8 e aiuta il passaggio
della corrente durante l'elettroforesi. La miscela è poi stata riscaldata per permettere all’agarosio di sciogliersi nel
L’elettroforesi del controllo positivo ha evidenziato il gene TEM della resistenza batterica in una posizione corrispondente a 400 paia di basi (pb)
quando il gene in realtà ne ha 516.
20
21 Anna Zimmermann
Resistenza batterica
2012/2013
tampone e ottenere una soluzione omogenea. In seguito è stata raffreddata brevemente e vi sono stati inseriti 4 microlitri
di GelRed. Questo è un intercalante del DNA che si inserisce tra le sue basi e ne permette la visualizzazione ai raggi
UV. Il liquido è poi stato versato nello stampo, in cui sono stati messi dei pettini per lasciar impressi dei pozzetti. L’ultimo
passo della preparazione del gel è stato lasciarlo solidificare per venti minuti. Il gel è quindi stato caricato con i diversi
campioni. In ogni pozzetto sono stati inseriti 10 µl dei campioni contenenti DNA amplificato e 4 µl di loading Buffer. Il
loading Buffer è stato usato per colorare i geni ampliati e per appesantirli, in modo da facilitare la loro migrazione e
consentire il corretto caricamento del pozzetto. Una volta inseriti i campioni, l’apparecchio per l’elettroforesi (vedi figura
6) è stato allacciato alla corrente. Per permettere la migrazione dei pezzetti di DNA sono stati necessari circa 30 minuti.
Nel gel è stato riservato anche un pozzetto per introdurvi un marcatore di taglia da 100 pb21, così da poter riconoscere il
gene attraverso la sua lunghezza (TEM 516 pb, SHV 392 pb, CTX-M 580 pb e OXA 619 pb).
Figura 6 In questa immagine si può osservare l’apparecchio per l’elettroforesi su gel (ICM) 3.3 Risultati 3.3.1 Risultati antibiogrammi Nella tabella seguente sono riportati i risultati ottenuti negli antibiogrammi, per i diversi ceppi batterici analizzati.
21
paia di basi 22 Anna Zimmermann
Resistenza batterica
2012/2013
Cefepime FEP
Aztreonam ATM
Cefotaxima CTX
Augmentin AMC
Ceftriaxone CRO
Cefpodoxime
CPD
Cefuroxime CX
Cefoxitina FOX
Ampicillina AM
Antibiotici e
diametro dell’alone d’inibizione misurato
con i diversi antibiotici (mm)
Provenienza
Specie
Ceppo
HWW-OSG
E. coli
55SG-CF
6
23
13
10
17
16
13
13
21
HWW-OSG
E. coli
56SG-CF
6
18
16
10
ND
ND
15
14
26
HWW-OSG
E. coli
58SG-CF
6
20
18
23
ND
15
27
30
29
HWW-OSG
C. freundii
66SG-CF
6
6
9
6
12
8
11
21
31
HWW-OSG
E. cloacae
67SG-CF
6
6
6
6
11
9
6
10
26
HWW-OSG
E. coli
72SG-CF
6
20
ND
ND
ND
ND
ND
ND
ND
HWW-OSG
E. coli
80SG-CF
6
20
NE
NE
NE
NE
NE
NE
NE
HWW-OSG
E. coli
85SG-CF
6
17
16
12
20
19
18
12
25
AS-DG
C. freundii
106DG-CF
9
8
9
6
15
11
9
19
27
AS-DG
C. freundii
108DG-CF
6
6
10
6
15
9
6
6
29
AS-DG
E. cloacae
109DG-CF
6
6
13
13
23
9
18
23
30
AS-DG
E. coli
110DG-CF
6
ND
ND
ND
ND
ND
ND
ND
ND
AS-DG
E. cloacae
115DG-CF
6
6
6
6
15
9
9
15
30
AS-DG
E. cloacae
117DG-CF
6
6
6
6
11
9
6
11
25
AS-DG
E. cloacae
120DG-CF
6
6
6
6
12
6
6
12
24
AS-DG
K. pneumoniae
122DG-CF
NE
NE
NE
NE
NE
NE
NE
NE
NE
AS-DG
E. cloacae
123DG-CF
6
6
6
6
12
9
6
16
21
AS-DG
E. cloacae
124DG-CF
6
6
6
6
12
9
6
12
26
AS-DG
E. cloacae
125DG-CF
6
6
6
6
15
9
8
16
29
AS-DG
C. freundii
126DG-CF
6
6
6
6
13
10
6
17
25
AS-DG
E. coli
130DG-CF
ND
ND
ND
ND
ND
ND
ND
ND
ND
AS-DG
E. coli
131DG-CF
NE
NE
NE
NE
NE
NE
NE
NE
NE
AS-DG
E. coli
133DG-CF
ND
ND
ND
ND
ND
ND
ND
ND
ND
AS-DG
E. coli
135DG-CF
ND
ND
ND
ND
ND
ND
ND
ND
ND
AS-DG
K. oxytoca
136DG-CF
ND
ND
ND
ND
ND
ND
ND
ND
ND
AS-DG
E. cloacae
141DG-CF
6
6
14
15
18
13
18
22
30
AS-DG
E. cloacae
143DG-CF
6
6
6
6
13
8
6
15
26
AS-DG
C. freundii
147DG-CF
6
6
8
6
13
10
6
18
25
AS-DG
K. oxytoca
150DG-CF
ND
ND
ND
ND
ND
ND
ND
ND
ND
SW-TicR-FDD
E. cloacae
157FDD-CF
6
6
6
6
16
10
6
12
28
SW-TicR-FDD
C. freundii
162FDD-CF
6
6
6
6
11
9
6
14
24
SW-TicR-FDD
E. coli
172FDD-CF
6
22
6
6
12
14
11
19
19
SW-TicR-FDD
C. freundii
183FDD-CF
6
6
10
6
14
9
6
19
27
SW-TicR-FDD
C. freundii
185FDD-CF
6
6
9
6
15
6
6
18
27
SW-TicR-FDD
E. coli
189FDD-CF
6
ND
ND
ND
ND
ND
ND
ND
ND
SW-TicR-FDD
C. freundii
197FDD-CF
6
6
9
6
15
6
6
19
27
SW-LC
E. coli
213LC-CF
ND
ND
ND
ND
ND
ND
ND
ND
ND
SW-LC
E. cloacae
248LC-CF
6
6
6
6
17
6
16
18
29
23 Anna Zimmermann
Resistenza batterica
2012/2013
Tabella 3 Illustra i risultati ottenuti dagli antibiogrammi dei 38 ceppi per i 9 antibiotici utilizzati (rosso: resistente, verde: 22
sensibile, NE: Non Eseguito e ND : Non Definito). Gli antibiotici in rosso sono quelli che devono manifestare la sinergia. In diverse capsule i batteri sono risultati sensibili a tutti o quasi a tutti gli antibiotici testati (vedi figura 7). Si tratta dei
ceppi che nella tabella presentano l’indicazione ND. Invece i ceppi batterici definiti NE non sono stati testati. Dalla tabella emerge che i ceppi analizzati sono in maggioranza resistenti ai primi otto antibiotici testati. Per l’antibiotico
Cefepime si registra invece un grado di resistenza sensibilmente minore; solo quattro dei ceppi analizzati sono risultati
resistenti a questo antibiotico.
I risultati percentuali per i diversi antibiotici, determinati senza prendere in considerazione i ceppi non definiti (ND) e non
eseguiti (NE), sono riportati di seguito:
•
•
•
Ampicillina (penicillina semisintetica): 100% Resistenti
Cefoxitina (cefalosporina di II generazione): 82% Resistenti
Cefuroxime (cefalosporina di II generazione): 96% Resistenti
•
Cefpodoxime (cefalosporina di III generazione): 96% Resistenti
•
•
•
Ceftriaxone (cefalosporina di III generazione): 95% Resistenti
Augmentin (Amoxicillina, penicillina semisintetica e acido clavulanico, inibitore delle β-lattamasi): 96 %
Resistenti
Cefotaxima (cefalosporina di III generazione): 96% Resistenti
•
•
Aztreonam (monobattame): 96% Resistenti
Cefepime (cefalosporina di IV generazione): 15% Resistenti
Figura 7 Fotografia dell’antibiogramma del ceppo 110DG-­‐CF. Il battere risulta sensibile a 8 dei 9 antibiotici testati. Gli antibiotici presentano il seguente ordine: 1. Ampicillina, 2. Cefoxitina, 3. Cefuroxima, 4. Cefpodoxima, 5. Ceftriaxone, 6. Augmentin, 7. Cefotaxima, 8. Aztreonam e 9. Cefepime. Questo ceppo presenta nella tabella l’indicazione ND (ICM)
Per la ricerca di batteri produttori di ESBL sono stati considerati i seguenti fattori: presenza di sinergia tra gli antibiotici
Cefotaxime, Augmentin e Cetriaxone e sensibilità all’antibiotico Cefoxitina.
22
I ceppi con l’indicazione ND sono risultati sensibili a tutti o quasi a tutti gli antibiotici testati.
24 Anna Zimmermann
Resistenza batterica
2012/2013
Dei 38 ceppi testati, solo due hanno dato risultati caratteristici del fenotipo ESBL. Negli antibiogrammi dei due ceppi si
può infatti notare la sinergia ricercata (vedi figura 8). Inoltre questi due ceppi non sono sensibili all’antibiotico Cefoxitina
e questa è un’altra caratteristica dei batteri con fenotipo ESBL. Questi due ceppi sono E.coli 55SG-CF e E.coli 172FFDCF.
Figura 8 Fotografia dell’antibiogramma del ceppo 55SG-­‐CF. Nell’antibiogramma è visibile la sinergia tra gli antibiotici Ceftriaxone (5), Augmentin (6) e Cefotaxima (7). Inoltre nella foto si può osservare la sensibilità alla Cefoxitina (2). Gli antibiotici presentano il seguente ordine: 1. Ampicillina, 2. Cefoxitina, 3. Cefuroxima, 4. Cefpodoxima, 5. Ceftriaxone, 6. Augmentin, 7. Cefotaxima, 8. Aztreonam e 9. Cefepime (ICM) Gli altri 36 ceppi batteri che sono stati testati non hanno evidenziato le caratteristiche ricercate. Osservando gli
antibiogrammi, si è notato che in diverse capsule (circa 22 su 38) i batteri hanno formato delle piccole colonie all’interno
dell’alone di inibizione, in genere intorno ai 3 dischetti con i quali i batteri produttori di ESBL mostrano delle sinergie
(circa 16 su 22 capsule).
25 Anna Zimmermann
Resistenza batterica
2012/2013
3.3.2 Risultati della PCR per i geni TEM, SHV, CTX-­M e OXA 3.3.2.1 Risultati ricerca gene TEM Il gene TEM è costituto da 516 paia di basi. Nell’immagine, che riporta la fotografia del gel d’agarosio per la migrazione
dei frammenti ottenuti dalla PCR per il gene TEM (Figura 9), si può osservare che unicamente 7 dei 38 batteri analizzati
possiedono questo gene: si tratta dei ceppi 58SG-CF, 72SG-CF, 115DG-CF, 124DG-CF, 130DG-CF, 141DG-CF e
189FFD-CF. Il risultato è stato dedotto dalla lunghezza della sequenza del gene amplificato, che corrisponde a quella
del controllo positivo. Figura 9 Fotografia del gel d’agarosio utilizzato per la ricerca del gene TEM. Nella porzione in alto sono stati caricati 15 campioni nella seguente sequenza: controllo positivo, controllo negativo, DNA plasmidico dei ceppi 1-­‐13; nella porzione intermedia altri 26 Anna Zimmermann
Resistenza batterica
2012/2013
15 campioni: DNA plasmidico dei ceppi 14-­‐28; nella porzione in basso 10 campioni: DNA plasmidico dei ceppi 29-­‐38. Per ogni porzione di gel è stato caricato al centro il marcatore di peso molecolare (ICM) Nell’immagine sono visibili della bande aspecifiche (indicate con l’asterisco nella Figura 9), che sono state differenziate
dal gene TEM grazie al loro peso molecolare23. Si tratta di sequenze nucleotidiche che i primers hanno ampliato
assieme al gene TEM e che l’elettroforesi ha permesso di mettere in evidenza.
3.3.2.2 Risultati ricerca gene CTX-­M Il gene CTX-M è formato da 580 paia di basi. La PCR ha evidenziato la presenza del gene CTX-M in uno solo dei 38
ceppi analizzati: il ceppo batterico 172FFD-CF. Il risultato è stato dedotto dalla lunghezza della sequenza nucleotidica
del frammento, che presenta lo stesso peso molecolare del gene ricercato (vedi Figura 10).
Figura 10 Fotografia del gel d’agarosio utilizzato per la ricerca del gene CTX-­‐M. Nella porzione in alto sono stati caricati 15 campioni nella seguente sequenza: controllo negativo, controllo positivo, DNA plasmidico dei ceppi 1-­‐13; nella porzione intermedia altri 15 campioni: DNA plasmidico dei ceppi 14-­‐28; nella porzione in basso 10 campioni: DNA plasmidico dei ceppi 29-­‐
38. Per ogni porzione di gel è stato caricato al centro il marcatore di peso molecolare (ICM) 23
Il numero di paia di basi del gene TEM è infatti diverso da quello delle bande aspecifiche.
27 Anna Zimmermann
Resistenza batterica
2012/2013
Anche in questo caso (vedi risultati gene TEM) sono visibili degli aspecifici non rilevanti, che si differenziano dal gene
ricercato per le dimensioni del frammento amplificato (vedi asterischi in Figura 10 ).
3.3.2.3 Risultati ricerca gene OXA Il gene OXA è costituito da 619 paia di basi. Da questo test è risultato che solo 2 dei 38 ceppi batterici presentano
questo tipo di gene: si tratta dei ceppi 55SG-CF e 124DG-CF. Il risultato è stato dedotto dalla lunghezza delle sequenze
nucleotidiche del gene amplificato, che corrisponde a quella del controllo positivo (vedi Figura 11). Figura 11 Fotografia del gel d’agarosio utilizzato per la ricerca del gene OXA. Nella porzione in alto sono stati caricati 15 campioni nella seguente sequenza: controllo positivo genomico, controllo negativo, controllo positivo plasmidico, DNA plasmidico dei ceppi 1-­‐12; nella porzione intermedia altri 15 campioni: DNA plasmidico dei ceppi 13-­‐27; nella porzione in basso 11 campioni: DNA plasmidico dei ceppi 28-­‐38. Per ogni porzione di gel è stato caricato al centro il marcatore di peso molecolare (ICM) 28 Anna Zimmermann
Resistenza batterica
2012/2013
Anche in questo caso (vedi risultati gene TEM) sono visibili degli aspecifici non rilevanti, che si differenziano dal gene
ricercato per le dimensioni del frammento amplificato (vedi asterischi in Figura 11).
3.3.2.4 Risultati ricerca gene SHV Il gene SHV è costituito da 392 paia di basi. La PCR ha segnalato che 4 ceppi batterici dei 38 analizzati presentano il
gene SHV: si tratta dei ceppi 55SG-CF, 56SG-CF, 85SG-CF e 122DG-CF. Il risultato è stato dedotto dalla lunghezza
della sequenza nucleotidica del gene (vedi Figura 12 ).
Figura 12 Fotografia del gel d’agarosio utilizzato per la ricerca del gene SHV. Nella porzione in alto sono stati caricati 15 campioni nella seguente sequenza: controllo positivo plasmidico, controllo positivo genomico, controllo negativo, DNA plasmidico dei ceppi 1-­‐12; nella porzione intermedia altri 15 campioni: DNA plasmidico dei ceppi 13-­‐27; nella porzione in basso 11 campioni: DNA plasmidico dei ceppi 28-­‐38. Per ogni porzione di gel è stato caricato al centro il marcatore di peso molecolare (ICM) Anche in questo caso (vedi risultati gene TEM) sono visibili degli aspecifici non rilevanti, che si differenziano dal gene
ricercato per le dimensioni del frammento amplificato (vedi asterischi in Figura 12).
29 Anna Zimmermann
Resistenza batterica
2012/2013
3.3.3 Sintesi dei risultati In sintesi, 14 dei 38 ceppi analizzati presentano geni che potrebbero determinare la produzione di ESBL, più
precisamente 7 presentano il gene TEM, 1 presenta il gene CTX-M, 2 presentano il gene OXA e 4 presentano il gene
SHV. Per stabilire se i ceppi risultati positivi alla ricerca dei geni TEM, SHV e OXA sono realmente produttori di ESBL,
sono necessarie più informazioni perché questi geni sono presenti in più varianti. Se i geni TEM, SHV e OXA rilevati
sono di prima generazione, cioè non presentano mutazioni, i batteri non sono in grado di produrre ESBL. Per contro la
presenza del gene CTX-M da una maggior certezza, in quanto generalmente è indice del fenotipo ricercato.
Come già indicato nel procedimento, per definire se un battere è produttore di ESBL è quindi necessario prendere in
considerazione diversi test. Alcuni test sono stati eseguiti in questo lavoro (antibiogramma e ricerca dei geni), altri sono
stati eseguiti in precedenza (selezione mediante piastre cromogene) e altri sono da effettuare (ad esempio il
sequenziamento dei geni amplificati dalla PCR) per avere la certezza che i batteri siano realmente produttori di ESBL.
Prendendo in considerazione i risultati della selezione iniziale, dell’antibiogramma e della ricerca dei geni, si è arrivati a
definire che solamente 2 dei 38 batteri testati, sono produttori di ESBL: i ceppi 172FDD-CF (E. coli) e 55SG-CF (E. coli).
Questi ceppi hanno infatti dato risultati caratteristici del fenotipo ESBL sia nell’antibiogramma (sinergia e resistenza alla
Cefoxitina), che nella ricerca dei geni di resistenza (presenza del gene CTX-M per il ceppo 172FDD-CF e presenza dei
geni OXA e SHV per il ceppo 55SG-CF. Tutti i 38 ceppi batterici, sono stati in seguito testati con un ulteriore
esperimento di conferma, che evidenzia i batteri ESBL: L’E-Test24. Questo test ha segnalato altri 2 batteri produttori di
ESBL: il ceppo 56SG-CF( E. coli ), già risultato positivo alla ricerca del gene SHV e il ceppo 58SG-CF (E. coli) che in
questo lavoro non ha dato nessun risultato positivo.
3.4 Discussione Per fare un confronto con la prima ipotesi riportata nell’introduzione, cioè che i batteri provenienti da ambienti con forte
presenza di antibiotici presentano maggiormente il fenomeno della resistenza, ci si è basati sui risultati ottenuti dalla
prima selezione su piastre cromogene. Questo test ha permesso di avere un’indicazione sulla diffusione della resistenza
agli antibiotici β-lattamici nei 4 ambienti testati, partendo da un campionario di 250 ceppi. Le riflessioni di seguito
proposte sono state quindi effettuate su un numero ridotto di ceppi batterici analizzati e sono solo in relazione a questo
tipo di resistenza. L’ipotesi iniziale prevedeva che nelle acque prima del depuratore di Giubiasco e nelle acque di scarico
dell’ospedale San Giovanni, si dovesse riscontrare un’alta percentuale di batteri resistenti contrariamente agli altri due
ambienti, il Lago Cadagno e il fiume dopo il depuratore, nei quali ci si aspettava di non trovare resistenze.
I risultati hanno evidenziato una maggior quantità di batteri resistenti nelle acque del fiume Ticino prima del depuratore
di Giubiasco rispetto agli altri tre ambienti (19 ceppi). Nei campioni delle acque di scarico dell’ospedale San Giovanni e
nelle acque del fiume Ticino dopo il depuratore di Giubiasco si è trovato un numero inferiore di ceppi resistenti (in
entrambi i casi 7 ceppi) e solo due ceppi resistenti in quelli provenienti dal Lago Cadagno. L’ipotesi che nelle acque
prima del depuratore ci fosse una maggior presenza del fenomeno resistenza ha avuto conferma. Probabilmente gli
antibiotici e i vari farmaci scaricati dalle varie abitazioni e dall’ospedale favoriscono la promozione per selezione naturale
della resistenza. Anche per l’ambiente Lago Cadagno i risultati confermano l’ipotesi iniziale. Il fatto che vi siano
comunque, in questo ambiente, dei batteri resistenti può essere spiegato con il verificarsi di mutazioni spontanee oppure
con il fatto che queste acque non sono forse cosi incontaminate come si crede. I risultati differiscono dalle ipotesi invece
per quanto concerne gli altri due ambienti. Ci si aspettava infatti un’alta percentuale di resistenza anche nelle acque di
24
Test eseguito dall’équipe dell’ICM.
30 Anna Zimmermann
Resistenza batterica
2012/2013
scarico dell’ospedale e una bassa, se non nulla, percentuale di batteri resistenti nelle acque dopo la stazione di
depurazione di Giubiasco. Questi risultati potrebbero essere spiegati dal fatto che gli antibiotici che si trovano nelle
acque di scarico dell’Ospedale San Giovanni provengono tutti dalla medesima fonte, ovvero il già citato ospedale.
Queste acque non possono essere comparate a quelle del fiume prima del depuratore, nel quale affluiscono acque
contenti antibiotici provenienti da molte più fonti, come ad esempio l’ospedale, abitazioni e ditte farmaceutiche. Per
questo motivo nel secondo ambiente si rileva probabilmente una quantità maggiore di resistenza.
Per quanto riguarda il ritrovamento di ceppi resistenti, anche se pochi, nelle acque dopo il depuratore di Giubiasco, lo si
può spiegare con il fatto che il depuratore è in grado tramite dei processi chimici, biologici, fisici, di eliminare la maggior
parte di batteri, ma non tutti [8]. In generale, questi risultati sembrano confermare l’ipotesi iniziale, che il diffondersi del
fenomeno della resistenza sia influenzato dalla presenza nell’ambiente di antibiotici.
Prendendo ora in considerazione gli esiti degli antibiogrammi, quasi tutti i batteri analizzati sono risultati resistenti alla
maggior parte degli antibiotici testati. Questo può essere spiegato con il fatto che i 38 ceppi analizzati sono stati
precedentemente selezionati per la ricerca del fenotipo ESBL, selezione che ha probabilmente escluso dai batteri rimasti
la presenza di batteri sensibili a penicilline e cefalosporine di II e III generazione, nei confronti dei quali i batteri ESBL
sono resistenti. L’unico antibiotico al quale i batteri sono risultati in maggioranza sensibili è il Cefepime che, essendo
una cefalosporina di IV generazione e quindi recente, è per ora in grado di uccidere i batteri analizzati. È però probabile
che, con il diffondersi del suo utilizzo, i batteri sviluppino la resistenza anche a questo antibiotico.
Un altro dato che emerge dai risultati degli antibiogrammi è il livello leggermente minore di resistenza registrato nei
confronti dell’antibiotico Cefoxitina (cefalosporina di II generazione). Questo dato potrebbe essere spiegato dal fatto che
questi 38 ceppi sono stati selezionati dai 250 ceppi prelevati dagli ambienti, tramite un test che escludeva tutti i batteri
che non potevano assolutamente produrre ESBL. Una delle caratteristiche dei batteri produttori di ESBL è la sensibilità
all’antibiotico Cefoxitina. Vi è un unico battere che è risultato sensibile a diversi degli antibiotici somministrati; si tratta del
ceppo batterico E. coli 58SG-CF. Dai test effettuati in questo lavoro, non è emerso come produttore di β-lattamasi a
spettro esteso, ma a seguito dell’E-test è stato identificato come tale. Una spiegazione possibile a questo risultato
potrebbe essere che il battere 58SG-CF non esprima una variazione del gene TEM, nonostante la possieda. Dunque
questo battere ha la capacità di produrre delle β-lattamasi, ma siccome la mutazione del gene TEM è inattiva, o forse
inibita dalla presenza di altri elementi, non manifesta il fenotipo ESBL. Per quanto concerne la diffusione del fenotipo
ESBL positivo negli ambienti considerati si può notare che solo 4 ceppi lo presentano. Nei ceppi batterici presi in esame
da questo studio, la capacità di produrre ESBL non è molto diffusa. Dato il numero ridotto di batteri analizzati non si
possono però trarre conclusioni definitive. Fatto interessante è che tre di questi ceppi sono stati isolati dalle acque di
scarico dell’ospedale e uno nelle acque in entrata al depuratore. Questo potrebbe essere spiegato dalla presenza
potenziale di antibiotici β-lattamici, i quali favoriscono lo sviluppo di questo tipo resistenza.
La seconda ipotesi invece, non ha potuto essere confermata. Il tipo di lavoro che si voleva utilizzare per un confronto era
diverso da questo, pertanto non è stato possibile fare il paragone. Il problema essenzialmente è dato dal fatto che lo
studio sui ceppi clinici ha ricercato la distribuzione di geni di resistenza considerando solamente ceppi ESBL, mentre
l’obiettivo di questo lavoro era di individuare in un campionario di batteri ambientali quali fossero ESBL.
I ceppi ESBL che sono stati individuati in questo lavoro (55SG-CF e 172FDD-CF) e quelli trovati dall’E-test (56SG-CF e
58SG-CF) hanno come geni di resistenza rispettivamente OXA e SHV il primo, CTX-M il secondo, SHV il terzo e TEM il
quarto. Con un solo gene di resistenza per tipo non è possibile fare statistiche. Nel lavoro sui ceppi clinici, nel quale
sono stati riscontrati più batteri ESBL con geni TEM (34 ceppi), OXA (29 ceppi) e alcuni con il gene SHV (5 ceppi), è
invece stato possibile. Sempre nello studio sui ceppi clinici non sono stati ricercati geni di resistenza CTX-M,
diversamente da quanto fatto per i batteri ambientali. L’unica considerazione che può essere fatta è che in questo studio
tutti i batteri produttori di ESBL sono della specie E. coli, che è la stessa specie in cui la resistenza ESBL era risultata
più frequente nel lavoro sui ceppi clinici. Probabilmente i batteri di questa specie sono più propensi a sviluppare questo
31 Anna Zimmermann
Resistenza batterica
2012/2013
tipo di resistenza. Dai risultati si può inoltre notare che certi batteri possono presentare più di un gene di resistenza che
conferisce il fenotipo ESBL.
4. Conclusione Durante lo svolgimento del mio lavoro di maturità ho avuto modo di approfondire il fenomeno della resistenza batterica,
focalizzandomi su una di queste: la resistenza ai β-lattamici. Ho avuto la possibilità di scoprire alcuni aspetti dei batteri,
tra i quali le diverse modalità con cui essi sviluppano la resistenza. Inoltre ho potuto studiare i diversi tipi di antibiotici e i
loro meccanismi d’azione. In particolare le mie conoscenze si sono ampliate nell’ambito della resistenza ai β-lattamici,
tema centrale del mio lavoro. Ad esempio sono venuta a conoscenza del fatto che nell’ambiente ci sono dei batteri
produttori di enzimi ESBL. Questi batteri mettono a dura prova il settore medico, in quanto pochi farmaci sono in grado
di eliminarli. Per far fronte alla resistenza occorre sintetizzare sempre nuovi farmaci e sempre più potenti. Per quanto
riguarda l’esecuzione della parte sperimentale, eseguita in una settimana all’istituto cantonale di microbiologia a
Bellinzona, si è svolta molto bene. Questa esperienza è stata molto interessante e del tutto nuova, poiché ho avuto la
possibilità di utilizzare sofisticati macchinari di laboratorio. I risultati ottenuti differiscono in parte dalla mia ipotesi sulla
distribuzione della resistenza batterica nei diversi ambienti. Inoltre non hanno permesso una comparazione esaustiva
con il lavoro di Miguel Fernandez “Caratterizzazione molecolare del fenotipo ESBL in Enterobatteriacee ed altri bacilli
Gram negativi di interesse clinico”.
Credo di aver portato a termine un buon lavoro, in quanto mi sono impegnata molto, investendo tanto tempo e tante
energie nella sua realizzazione.
5. Ringraziamenti Vorrei terminare il mio lavoro di maturità esprimendo la mia gratitudine verso coloro che hanno reso possibile il suo
svolgimento, mettendo a mia disposizione la loro esperienza e la loro attenzione. Pertanto ci tengo a fare i dovuti
ringraziamenti alla docente che ha seguito il mio progetto durante questi mesi, Vania Della Chiesa e ai collaboratori
dell’istituto di microbiologia, che mi hanno dato la possibilità e mi hanno aiutata a svolgere la parte sperimentale, in
particolare alla dottoressa Antonella de Marta, Federica Mauri e Alessandro Carnelli.
32 Anna Zimmermann
Resistenza batterica
2012/2013
6. Bibliografia •
•
•
•
•
•
[1] La Placa, M.. 2001. I principi della microbiologia medica. Esculapio. Bologna. Pp. 173-194, pp. 203-207 e
pag. 281.
[2] Fisher, R. D., e Harvey, R., e Champe, P. C.. 2008. Le basi della microbiologia. Zanichelli. Bologna. Pp. 4650.
[3] Campbell, N., e Reece, J., e Mitchell, L., e Taylor, M.. 2009. Immagini della biologia. Volumi B, E. Seconda
edizione. Zanichelli. Bologna. Pag. 59, pp. 226-227, pp. 726-727.
[4] Pini K. 2011/2012. Resistenza multibatterica agli antibiotici nel Canton Ticino. Lavoro di maturità. Liceo
Bellinzona.
[5] Fernandez Parra, F.M.. 2007/2008. Caratterizzazione molecolare del fenotipo ESBL in Enterobatteriacee
ed altri bacilli gram negativi di interesse clinico. Lavoro di diploma. Scuola Specializzata Superiore Medico
Tecnica Locarno.
[6] De Marta, A., e Ruggeri N. (2010). Proposta di una metodica per la detezione fenotipica di β-lattamasi a
spettro esteso (ESBL) e AmpC in batteri Gram negativi patogeni. Rapporto ESBL. Istituto Cantonale di
Microbiologia Bellinzona.
Sitografia • [7] Todar, K. (2009). Meccanismi di patogenità.
http://translate.google.ch/translate?hl=it&langpair=en%7Cit&u=http://textbookofbacteriology.net/themicrobialwo
rld/pathogenesis.html (17.10.12)
• [8] Consorzio Depurazione Acque Lugano e Dintorni. 2011. Il percorso dell’acqua. http://www.cdaled.ch/
(22.11.12)
• [9] Meccanismi di resistenza agli antibiotici. http://www.microbiologia.unige.it/dpb/Appunti/M-gener05.htm (22.11.12)
Comunicazioni verbali •
•
•
•
•
[10] Luca Tito Bertini, docente biologia Liceo Lugano 1
[11] Lisa Palme, docente biologia Liceo Lugano 1
[12] Antonella Demarta, Dottoressa in biologia, collaboratrice all’istituto cantonale di microbiologia
[13] Federica Mauri, collaboratrice all’istituto cantonale di microbiologia
[14] Alessandro Carnelli, collaboratore all’istituto cantonale di microbiologia
Fonte immagine copertina:
http://www.google.ch/imgres?q=evolution+bacteria&um=1&hl=it&sa=X&tbo=d&biw=1280&bih=614&tbm=isch&tbnid=DfV
RPR6X2euatM:&imgrefurl=http://whyfiles.org/2001/evolutioneverywhere/&docid=qj0DLwL1PHGWlM&imgurl=http://whyfiles.org/shorties/085fast_evolution/images/bacteria.jpg&w=15
4&h=200&ei=21zsUPCmBozDswaqmICQCA&zoom=1&iact=rc&dur=220&sig=109978370248423610390&page=1&tbnh
=148&tbnw=114&start=0&ndsp=20&ved=1t:429,r:7,s:0,i:106&tx=83&ty=29
33