
Ignazio Pio Gallo
QUINDI RICAPITOLANDO, L’ANTIGENE 0 SI FORMA GRAZIE ALL’ENZIMA H, CHE AGGIUNGE UN FUCOSIO.
MODIFICHE DELL’ANTIGENE 0, IN PRESENZA DI ALLELI A OPPURE B, INDUCONO LA FORMAZIONE DI ANTIGENI
A E B. IN PARTICOLARE, L’AGGIUNTA DI UN’N-ACETILGLUCOSAMINA INDUCE LA FORMAZIONE
DELL’ANTIGENE A, MENTRE L’AGGIUNTA DI UN ULTERIORE GALATTOSIO, INDUCE LA FORMAZIONE
DELL’ANTIGENE B.
L’ANTIGENE 0 VERRA’ QUINDI SEMPRE RICONOSCIUTO COME SELF, PERCHE’ PRESENTA UNA STRUTTURA
COMUNE AGLI ALTRI.
FENOTIPO BOMBAY
È una possibile configurazione del gruppo sanguigno.
I gruppi A, B e AB sono caratterizzati dalla presenza di antigeni sulla superficie degli eritrociti. Tali antigeni
sono generati dal legame tra una molecola H ed uno zucchero complesso, che si forma per mezzo di enzimi A
o B. Quando tali enzimi sono assenti, la sola presenza della molecola H, non basta a generare l’antigene.
In questa rara condizione, una mutazione in un gene non correlato, impedisce l’espressione dei fenotipi A e
B. Negli individui omozigoti per un allele recessivo h, non può avvenire l’assemblaggio degli antigeni A e B
sulla superficie delle cellule, quindi sono fenotipicamente 0, ma genotipicamente hanno gli alleli 𝐈 𝐀 o 𝐈𝐁 .
In questo caso, essere omozigoti per l’allele h (hh), impedisce l’espressione fenotipica degli alleli 𝐈 𝐀 o 𝐈𝐁 , e
questo altro non è che un’interazione genica epistatica. (Vedi EPISTASI successivamente).
Quindi hh, che è mutato in omozigosi, impedisce la manifestazione degli alleli IA o IB, perché è epistatico.
- Ma perché è epistatico?
Il gene H è in grado di produrre la sostanza H a partire dal polisaccaride di partenza. Questa sostanza H, infatti,
viene prodotta aggiungendo fucosio al glucide di partenza. L’aggiunta di fucosio avviene grazie alla fucosiltransferasi, un enzima codificato dal gene H.
Come abbiamo già detto, l’aggiunta di n-acetilgalattosamina o di un ulteriore molecola di galattosio a questo
glucide di base, porta alla formazione di antigene A o B.
Se, come nel caso del fenotipo Bombay, abbiamo una mutazione omozigote recessiva (hh), non si verrà mai
a formare questa sostanza h, e di conseguenza, anche se è presente l’allele IA, che codifica per l’enzima IA
(in grado di aggiungere n-acetil galattosamina), questa non verrà mai aggiunta, dunque fenotipicamente non
ci sarà l’antigene A. Per lo stesso motivo non sarà possibile formare l’antigene B.
‘’In queste condizioni, quindi, qualunque assetto genico in AB0 non verrà mai manifestato, perché il gene H
mutato in omozigosi, non permette la sintesi della sostanza di partenza, e la conseguente aggiunta di altri
zuccheri che costituiscono antigeni A e B. Dunque il gene h è epistatico rispetto ad IA e IB, che sono geni
ipostatici’’.
www.facebook.com/AppuntiMedicina.Farmacia.CTF
19
Ignazio Pio Gallo
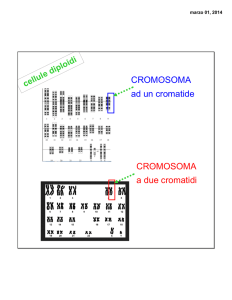
Il DNA dei centromeri è costituito da sequenze di DNA
altamente ripetute.
Originariamente, furono identificati 7 gruppi, indicati
con le lettere maiuscole dalla A alla G, all’interno dei
quali i cromosomi erano numerati in ordine decrescente
rispetto alla lunghezza.
Nella classificazione, però, è presente un errore. Infatti,
il cromosoma 21, sebbene sia leggermente più piccolo
del cromosoma 22, fu indicato inizialmente come più
grande. Tutt’oggi, si trova in questa posizione errata.
Dato che alcuni cromosomi sono morfologicamente
molto simili tra loro, negli anni ’70 furono introdotte
delle particolari tecniche di analisi come il BANDEGGIO.
Le tecniche di bandeggiatura, permettono di
distinguere un cromosoma dall’altro, facendo
riferimento alla colorazione di ciascuno di essi,
secondo un’alternanza di bande trasversali
chiaramente distinguibili. Queste bande, che
appaiono chiare e scure, permettono di
indentificare ogni singolo cromosoma. Gli
omologhi presentano un pattern praticamente
identico. Si ritiene che ogni banda (detta banda
G), contenga circa 100 geni.
Per quanto riguarda, invece, la nomenclatura specifica e
standard, per i cromosomi, nel ’71, in un convegno a
Parigi, si stabilì che:
‘’Ogni cromosoma risulta costituito da un braccio corto
(p – petit) e un braccio lungo (q – queu -coda), separati
da un centromero’’.
OGGI – si fa riferimento al Sistema Internazionale per la
Nomenclatura in Citogenetica umana (ISCN), secondo
cui ogni cromosoma, oltre ad essere distinto in un
braccio corto (p) ed uno lungo (q), presenta (in ogni
braccio) delle regione numerate con i numeri arabi, a
partire dal centromero e andando verso la parte
terminale (p1-p2…).
www.facebook.com/AppuntiMedicina.Farmacia.CTF
45
Ignazio Pio Gallo
4. Traslocazione – è lo spostamento di tratti di DNA, secondo 3 modalità:
a. Traslocazione intracromosomica non reciproca – spostamento di un tratto di DNA in un’altra
zona dello stesso cromosoma.
b. Traslocazione intercromosomica non reciproca – spostamento di un tratto di DNA su un
cromosoma diverso.
c. Traslocazione intercromosomica reciproca – scambio di tratti non omologhi tra 2 cromosomi.
CROMOSOMA PHILADELPHIA
Un esempio di traslocazione è rappresentata dal cromosoma Philadelphia. È un caso di traslocazione
reciproca che coinvolge i cromosomi 9 e 22. In particolare, succede che il proto-oncogene abl, in seguito a
questo scambio reciproco, passa dal cromosoma 9 al cromosoma 22. Questo processo causa una
trasformazione neoplastica che conduce alla leucemia mieloide cronica con crescita incontrollata dei
mioblasti. Sembra che, in seguito allo spostamento del gene abl, venga sintetizzata una proteina di fusione
che presenta elevata attività tirosin-chinasica.
Questo tipo di traslocazione è Reciproca e cambia la morfologia dei cromosomi, poiché il segmento del 22 che
si sposta sul 9, è più grande del segmento
del 9 che si sposta sul 22.
Questo tipo di traslocazione è stata la
prima anomalia cromosomica correlata
direttamente alla patogenesi di una
malattia neoplastica come la leucemia
mieloide cronica. È stato dimostrato che,
nel 95% o 100% dei casi, tutte le leucemie
mieloidi presentano un gene di fusione
chiamato BCR-ABL. Questo prodotto
chimerico (chimera: fusione di due geni)
deriva da una traslocazione bilanciata
(reciproca) tra i cromosomi 9 e 22.
-
ABL – è una tirosin-chinasi, che però ha anche un dominio di auto-inibizione, e di conseguenza passa
da uno stato fosforilato, ad uno defosforilato. Possedendo un’attività fosfatasica, dunque, si alterna
tra lo stato attivo ed inattivo.
-
BCR – Break-Point Cluster Region, ovvero punti di rottura localizzati sul cromosoma 22, che hanno
attività serin-tirosin-chinasica.
www.facebook.com/AppuntiMedicina.Farmacia.CTF
60
Ignazio Pio Gallo
MOSAICISMO CROMOSOMICO
Come si forma il mosaico 45, X - 46, XY?
- Esso deriva da uno zigote maschio che in una delle divisioni mitotiche post-zigotiche (primissime fasi
embrionali) ha subito una non disgiunzione mitotica di Y e così si formano i due cariotipi differenti.
MOSAICISMO E CHIMERISMO
Mosaicismo – è la coesistenza di cloni cellulari con assetto genetico differente ma che provengono dallo stesso
organismo.
Chimerismo – è la coesistenza di popolazioni cellulari geneticamente diverse, che provengono da due individui
diversi. Per esempio nei trapianti, l'organo ha un patrimonio genetico diverso da quello del corpo che lo ospita.
DISOMIA UNIPARENTALE (UPD)
È un fenomeno piuttosto raro, in cui, in uno zigote, i 2 cromosomi di una coppia di omologhi non vengono
ereditati uno dal padre ed uno dalla madre, ma essi sono ereditati entrambi da uno solo dei genitori.
L’individuo ha comunque un corredo cromosomico corretto, che può essere 46, XX o 46, XY. Se il genitore da
cui eredita i due cromosomi è affetto da una malattia autosomica recessiva, il figlio con disomia la presenterà
sicuramente nonostante l’altro genitore sia sano.
Questo particolare fenomeno può essere causato da:
a) Non disgiunzioni in entrambi i genitori – per cui un gamete ha due copie di un cromosoma e l'altro
non ne ha nessuna. Si ottiene uno zigote con disomia (complementazione gametica).
b) Non disgiunzione di un genitore seguita da duplicazione dell'embrione – il cromosoma generico viene
dal padre per esempio e manca quello della madre o viceversa. Si forma lo zigote che compie una
duplicazione post-zigotica presentando così la disomia (recupero monosomia).
c) Perdita del cromosoma nello zigote seguita da una duplicazione – quindi lo zigote è normale, perde
un cromosoma e poi duplica quello rimanente presentando così la disomia (recupero monosomia).
d) Non disgiunzione in un genitore e perdita del cromosoma nell'embrione – lo zigote che si forma
presenta una trisomia, questo zigote poi perde un cromosoma e presenta così una disomia (recupero
trisomia).
Diversi meccanismi che portano alla disomia uniparentale:
www.facebook.com/AppuntiMedicina.Farmacia.CTF
72
PEDIGREE DI UNA PATOLOGIA AUTOSOMICA DOMINANTE
Ignazio Pio Gallo
IPERCOLESTEROLEMIA FAMILIARE
Patologia autosomica dominante molto diffusa (1/500 individui),
caratterizzata da colesterolemia elevata, appunto, dovuta all’accumulo di
colesterolo lipoproteine a bassa densità (LDL), le lipoproteine
trasportatori del colesterolo, apolari, che circolano nel flusso ematico.
Le lipoproteine sono classificate in base alla densità in:
- ALTA – HDL
- BASSA – LDL
Il colesterolo, tramite le lipoproteine, viene trasportato nelle cellule, dove
poi viene utilizzato come componente di membrana (essendo un
regolatore di fluidità di membrana), e per la sintesi di ormoni steroidei e
Sali biliari. Le HDL trasportano il colesterolo al fegato, dove, viene portato
ai tessuti ad opera delle LDL.
Gli individui affetti da ipercolesterolemia familiare, sono soggetti a
patologie cardiache, dovute alla formazione di placche causate da
accumulo di colesterolo. I soggetti omozigoti (AA) manifestano un quadro
clinico più grave rispetto agli eterozigoti (Aa), in quanto presentano livelli
di colesterolo particolarmente elevati. Questa comune patologia è causata da assenza o alterazione dei
recettori di superficie che legano e regolano l’ingresso delle LDL nelle cellule.
Il gene che codifica il recettore delle
LDL, localizzato sul cromosoma 19,
risulta mutato, codificando, così, per
recettori difettosi, che hanno scarsa
affinità per le LDL.
www.facebook.com/AppuntiMedicina.Farmacia.CTF
75
Ignazio Pio Gallo
Come diretta conseguenza, le malattie genetiche dovute a mutazioni nei geni mitocondriali:
- Sono ereditate esclusivamente per via materna, e danno luogo ad una particolare modalità di
trasmissione;
- Tutti i figli di madri malate, sono malati. Le femmine malate, trasmettono, infatti, la patologia a tutti i
figli, sia maschi, che femmine, ma i maschi non la trasmettono a nessun figlio.
Essendo (i mitocondri) le centrali energetiche della cellula, una possibile mutazione dei geni mitocondriali,
conduce alla produzione di una minore quantità di energia.
In genere le malattie da mutazione di DNA mitocondriale, sono tutte malattie progressive dei tessuti ad alto
consumo energetico, quindi sono delle patologie che si esplicano in maniera molto severa sul cuore, cervello
(SNC), muscoli, che sono ovviamente gli organi che più degli altri hanno bisogno di essere sostenuti da un
metabolismo ossidativo.
Le patologie di questo tipo che colpiscono i muscoli, sono raggruppate sotto il nome di miopatie
mitocondriali; mentre quelle che riguardano sia i muscoli, sia il sistema nervoso, prendono il nome di
encefalomiopatie mitocondriali.
-
Miopatie mitocondriali – debolezza muscolare e morte del tessuto muscolare, con conseguente
paralisi dei muscoli oculari, palpebre cadenti, difficoltà nella deglutizione e nella fonazione.
-
Encefalo-miopatie mitocondriali – ai problemi muscolari, si affiancano problemi nervosi.
‘’Ogni cellula contiene da 2 a 1000 mitocondri, ma
da cosa può dipendere questa grossa variabilità?
Esistono cellule che hanno un metabolismo
ossidativo più fiorente e chiaramente
presenteranno più mitocondri (cellule epatiche) e
cellule che hanno, invece, un metabolismo più
ridotto, e che ovviamente avranno un numero
inferiore di mitocondri. Sostanzialmente,
coesistono numerosi mitocondri, ed in ciascuno di
essi, più genomi, da 2 a 10’’.
www.facebook.com/AppuntiMedicina.Farmacia.CTF
91
Ignazio Pio Gallo
La precipitazione viene stimolata grazie alle basse temperature (-80°C) e aggiungendo Sali come 𝑵𝒂+ , in modo
da neutralizzare i fosfati e rendere meno solubile l’acido nucleico, che allora precipita più velocemente.
‘’Alla fine, sostanzialmente, il prodotto ottenuto dal processo di purificazione, è un precipitato di DNA.
Quindi la differenza di solubilità delle fasi organiche e delle fasi acquose, permette di recuperare il DNA’’.
Il DNA, dopo disidratazione, viene risospeso in una soluzione diluita di Sali, alla concentrazione desiderata.
Oggi per l’estrazione degli acidi nucleici, specialmente di DNA, si utilizza una cromatografia su colonna di
affinità. La colonna ha diverso diametro, a seconda che si voglia isolare DNA o RNA, e diversi buffer di
eluizione più o meno acidi. È presente un filtro microporoso, che permette di eluire le proteine e di
raccogliere l’acido nucleico che rimane adeso.
ENZIMI PER LA MANIPOLAZIONE DEGLI ACIDI NUCLEICI
Gli enzimi che permettono di
manipolare gli acidi nucleici in vitro,
possono essere raggruppati in 3
categorie:
a. DNA Polimerasi;
b. Nucleasi;
c. Ligasi.
A. DNA POLIMERASI
Le DNA polimerasi effettuano sintesi di DNA a partire da uno stampo di DNA o RNA. È un enzima
multifunzionale che sintetizza ex novo un filamento di DNA in direzione 5’3’, copiando lo stampo in
direzione opposta (3’5’) e partendo da un innesco, il PRIMER. Inoltre, la DNA polimerasi, contiene anche
attività esonucleasiche 3’5’ e 5’3’. Queste attività esonucleasiche sono sito di correzione di bozze.
www.facebook.com/AppuntiMedicina.Farmacia.CTF
105
Ignazio Pio Gallo
VETTORI DI ESPRESSIONE
Partendo dalla struttura di base dei plasmidi utilizzati per il clonaggio, sono state apportate delle modifiche,
per permettere, ad esempio, la sintesi di RNA o proteine corrispondenti al DNA clonato. Affinché ciò sia
possibile, i vettori devono avere determinate caratteristiche:
- Devono contenere specifici segnali corrispondenti:
o Al tipo di prodotto da esprimere, quindi RNA o proteina;
o All’ambiente, quindi eucariotico o procariotico.
Ad esempio, i vettori per la sintesi di RNA in ambiente procariotico, contengono gli stessi segnali usati per la
clonazione, ma in più, al 5’ della regione polylinker, è localizzato un promotore per la trascrizione. Alcuni
vettori possiedono un promotore anche al 3’, in modo da originare, dallo stesso frammento di DNA, un
trascritto senso, oppure antisenso. Questi trascritti, definiti sonde a RNA, vengono prodotti in vitro, usando
un plasmide purificato e linearizzato con enzima di restrizione che possiede un unico sito di taglio a valle del
sito di inserzione del DNA esogeno.
L’architettura di un vettore di espressione prevede allora:
a. ORI – origine di replicazione;
b. Marcatore di selezione – per la manipolazione
in E. coli;
c. Polylinker;
d. Promotore costitutivo forte eucariotico al 5’ –
che permette un’elevata espressione in
un’ampia gamma di cellule di mammifero;
e. Segnale di poliadenilazione e di terminazione
al 3’.
f. RBS – quando presente, è un sito di legame per
il ribosoma, che rende più efficiente la
traduzione.
Esiste quindi un’ampia gamma di vettori utilizzabili a seconda delle esigenze del ricercatore. Tra questi, i
vettori retrovirali e virali. Nel caso dei retro-virali, si lavora sul cDNA, che copia tramite la trascrittasi inversa
dell’RNA. In genere, i vettori virali e retro-virali sono molto più efficienti di quelli plasmidici, dato che
permettono l’inserzione di frammenti molto più grandi (fino a 23 Kb).
Tuttavia, dal punto di vista della sicurezza, l’utilizzo di tali vettori risulta rischioso. L’inserimento di alcuni virus
nel nostro genoma, infatti, potrebbe creare situazioni potenzialmente oncogeniche.
‘’Quindi con i sistemi di clonaggio si utilizza sostanzialmente un sistema artificiale, per far esprimere
all’interno di un sistema cellulare, il gene di nostro interesse. In questo caso, tutte le cellule batteriche che
hanno ricevuto un plasmide funzionante costituiscono dei cloni cellulari, in cui questo gene si è praticamente
amplificato, nel senso che è posseduto da tutte le cellule che derivano dalla cellula progenitrice, in cui
l’inserzione è avvenuta’’.
AMPLIFICAZIONE DI DNA IN VITRO
Il sistema della reazione a catena della polimerasi (PCR), ha rivoluzionato la ricerca, permettendo il
conseguimento di obiettivi fino a poco tempo prima impensabili, come l’identificazione personale su base
genetica, le diagnosi di malattie genetiche o di infezioni da patogeni in tempi molto brevi.
PCR = (Polymerase Chain Reaction)
Il metodo rappresenta un’amplificazione del processo di replicazione del DNA, permettendo la replicazione
ripetitiva di una specifica regione di DNA. In particolare, la PCR, permette di poter sequenziare un particolare
gene e di amplificarlo selettivamente. La scoperta si deve ad uno studente di 23 anni, laureato in Biochimica,
KARY MULLIS, che qualche anno dopo ricevette il Nobel.
www.facebook.com/AppuntiMedicina.Farmacia.CTF
111




![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)

![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)




