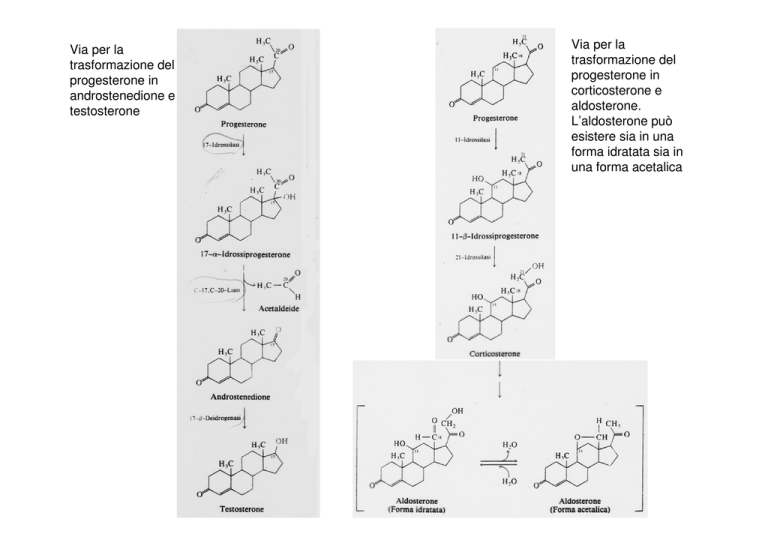
Via per la
trasformazione del
progesterone in
androstenedione e
testosterone
Via per la
trasformazione del
progesterone in
corticosterone e
aldosterone.
L’aldosterone può
esistere sia in una
forma idratata sia in
una forma acetalica
Via per la trasformazione del
testosterone in β-estradiolo
17-OH
Estradiolo
Sequenza di reazione che porta all’aromatizzazione degli androgeni ad estrogeni
Receptors for steroid hormones and thyroid hormones contain similar DNA-binding
domains and different hormone-binding domains.
Struttura di due domini presenti nei recettori nucleari. I Recettori nucleari contengono
due comini conservati: (1) un dominio di legame al DNA in una zona centrale della sequenza proteica
e (2) un dominio di legame al ligando in prossimità del terminale carbossilico.
Un modello per l’azione del recettore per gli estrogeni (ER).
Abbr: E, estrogen; R, receptor; ERE, estrogen response element; GF, growth factor; TBP, TATA
binding protein; TAFs, TBP-associated factors; pol II, RNA polymerase II.
Riconoscimento specifico di sequenze da parte dei recettori per gli steroidi.
Sono mostrate le sequenze degli aminoacidi nel dominio di legame al DNA del recettore per
glucocorticoidi.
Gli aminoacidi nel P-box e D-box sono importanti, rispettivamente, per il riconoscimento e la
dimerizzazione con il DNA
Stabilizzazione da parte del recettore per gli steroidi del complesso di pre-inizio della trascrizione
Sequenze consenso dei siti di DNA, chiamate
elementi di risposta, che si legano al recettore
per i glucocorticoidi (GRE), al recettore per gli
estrogeni (ERE), al recettore per la vitamina D3
(VDRE), al recettore per l’ormone tiroideo
(TRE) e al recettore per l’acido retinoico
(RARE). Le ripetizioni invertite in GRE e ERE e
le ripetizioni dirette in VDRE, TRE e RARE
sono indicate con le frecce.
Ligand-dependent recruitment of multiple coactivator complexes.
Upon ligand binding, the receptors recruit different coactivator complexes. The complex CBP/p160/PCAF
possesses histone acetyltransferase activity, the SWI/SNF complex possesses ATP-dependent chromatin
remodeling activity, and the TRAP/DRIP complex may recruit the RNA polymerase II (RNAP II) holoenzyme.
Recruitment of the complexes may be sequential or combinatoria. It is conceivable that chromatin remodeling
complexes are initially recruited to the promoter. These factors may relieve the repression imposed by highorder chromatin structure and allow a second acetylation-dependent step on gene activation. Activation would
require the combinatorial of subsequent action of additional complexes that include the TRAP/DRIP complex.
Hypotetical minimal model of a
non-DNA binding form of a
steroid receptor.
This form of the receptor cannot
bind to DNA because the DNA
binding site is blocked by the 94
kDa hsp protein or by some other
constituent. Molecular weight of this
complex is approximately 300 kDa.
Ligand binding to nuclear hormone receptor. The ligand lies completely surrounded
within a pocket in the ligand binding domain. The last alpha helix, folds into a groove on
the side of the structure on ligand binding
Coactivator recruitment.
The binding of a nuclear
hormone receptor induces
a conformational change in
the ligand-binding domain.
This change in
conformation generates
favorable sites for the
binding of a coactivator
Estrogen receptor-tamoxifen complex.
Tamoxifen binds in the pocket normally
occupied by estrogen. However, part of the
tamoxifen structure extends from this pocket,
and so helix 12 cannot pack in its usual
position. Instead, this helix blocks the
coactivator-binding site.
Enzymes that catalize such reactions are called Histone acetyltransferases (HATs)
Structure
of
acetyltranserase.
the
Histone
The ammino-terminal tail of the
histone H3 extends into a pocket in
whicha lysine side chain can accept an
acetyl group from acetyl CoA bound in
a adjacent side.
SH
SH
SH
SH
DAG
PKC
DAG
PKC
SH
p110
Scaffold
SH
SH
IP3
α
GTP
γ
Ras-GTP c-Raf
β
Shc
P
c-Raf
cAMP
PI3-4-5 P
Src
Pi
AC
AKT P
p85
Sr
c
PIP3 PLC
P
P
Pi
Shc Grb2
PKA
P
Sos
PI3-Kinase
Signal transduction systems
PKA
PKC
PLC
PI3K
Ras/MAPK
E-NOS
EGFR/matrix metalloproteinase
Rapid response outcomes
Ion channels
Transcription
Translation
Protein kinase/phosphatase
Structural protein
Signalling enzymes
Feddback regulation
SH
Steroid hormone
Schematic diagram of a steroid hormone interacting with four classes of membrane receptors to generate second messengers
linking to variety of signal-transduction systems. A) Three classes of membrane receptor are shown illustrating the classic nuclear
steroid-hormone receptor associated with a caveola. Aa) The receptor is technically outside the cell and is associated with the
outer surface of the plasma membrane in the flask of the caveola. Ab) The receptor is tethered by a scaffolding protein to the
plasma membrane on the inner surface of the caveola. Ac) The receptor is tethered to the caveolae by a palmitic acid molecule
that is esterified to a recoptor Ser or Thr with the fatty-acid side chain “inserted” into the membrane (palmitoylation). B) A G-protein
coupled receptor with its ligand binding domain on the outside of the cell and a seven-membrane spanning peptide transition
followed by an intracellular peptide domain that can bind G alpha beta and gamma proteins. C) A single-spanning membrane
receptor with intrinsic kinase activity that might be functional as a monomer. D) Same as C except a homodimer. Caveolae are
flask-shaped membrane invaginations present in the outer cell membrane of many cells; they are believed to serve as a platform
to accumulate or dock signal transduction-related molecules. The signal transduction systems are listed as candidates for
madiating rapid responses to steroid and are based to published data. The details remain to be defined on the basis of careful
experimentation. The two ovals with Ras-GTP and c-Raf are to suggest that c-Raf was recruited to the complex. AC adenylyl cyclase;
DAG, diacylglycerol; EGFR, epidermal groth factor receptor; e-NOS, endothelial nitric oxide synthase; IP3, inositol triphosphate; MAP, mitogen-activated
protein; PI3K, phosphatidylinositol 3-kinase; PIP3, phosphatidylinositol triphosphate; PKA, protein kinase A; PKC, protein kinase C; PLC, phospholiphase C
Extracellular
Intracellular
Endoplasmic
reticulum
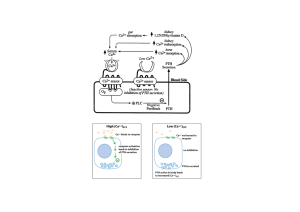
Diagram illustrating the manner in which Ca2+ acts as an extracellular messenger. Activation of the Ca2+-sensing receptor (by
binding of Ca2+ to negatively charged regions) activates phospholipase C (PLC; possibly via Gq protein), leading to increased
intracellular levels of diacyglycerol (DG) and inositol 1,4,5-trisphosphate (IP3), and concomitant release of Ca2+ from internal
stores (e.g. the endoplasmic reticulum). The rise in Ca2+i is sustained by influx of Ca2+ through channels in the plasma
membrane. The Ca2+-sensing receptor can also reduce receptor-mediated increases in cAMP levels (possibly via Gi protein).
The Ca2+-induced changes in the activities of these second messenger systems leads to changes in the activities of a series of
kinases (e.g. PKC and PKA), which in turn alter the biological activities of the cell. AC, adenylate cyclase; PIP2,
phosphatidylinositol bisphosphate.
Correlazione strutturale fra
vilamina D3 colecalciferolo e
vitamina D2 ergocalciferolo rispetto
alle loro rispettive pro-vitamine e al
colestrerolo. Le due
rappresentazioni strutturali
mostrate in basso per la vitamina
D3 e D2 sono equivalenti; sono
semplicemente modi diversi per
mostrare la stessa molecola.
Si sottolinea che la vitamina D3 è
la forma naturale della vitamina; è
prodotta dal 7-deidrocolesterolo
che è presente nel sotto cute ed è
modificato dall’azione dei raggi
solari. La vitamina D2 che è
efficace in maniera equivalente
nell’uomo e molti mammiferi ma
non negli uccelli, è prodotta a
livello commerciale e deriva
dall’irradiazione UV delle piante
Colecalciferolo-25-idrossilasi
P450-MITOCONDRIO
Trasformazione della vitamina D3
in 1,25-diidrossivitamina D3
25-Idrossicolecalciferolo-1α-idrossilasi
P450-MITOCONDRIO
Ipocalcemia
Ca2+
FEGATO
Ipofosfatemia
Pi
25-idrossilasi
7-DEIDROCOLESTEROLO
(pro-vitamina D3)
25(OH)D3
PTH
Radiazioni UV
pelle
RENE
1-idrossilasi
+
+
24-idrossilasi
VITAMINA D3
1-α,25(OH)2 D3
24,25(OH)2 D3
+
1,24,25(OH)3 D3
Organi bersaglio
Vie di formazione dei metaboliti della Vitamina D3.
La provitamina D3 viene attivata
fotochimicamente sulla pelle e trasformata in una
struttura aperta detta colecalciferolo. Questa
molecola subisce l’idrossilazione nel fegato in
C25 e quindi nel rene viene idrossilata in C1.
L’enzima 1 idrossilasi renale è attivato dall’ipocalcemia, mediante azione del PTH e dalla ipofosfatemia. I livelli di
1,25(OH)2 D3 possono attivare la 24 idrossilasi per formare dei metaboliti idrossilati inattivi.
ORMONI TIROIDEI
3,5,3’,5’ TETRAIODOTIRONINA (TIROXINA) o T4
3,5,3’ TRIIODOTIRONINA o T3
Modello del metabolismo dello
ione
ioduro
nei
follicoli
tiroidei.
La figura mostra una cellula
follicolare posta tra il lume
follicolare
e
lo
spazio
extracellulare (in basso).
Lo ioduro entra nella tiroide
soprattutto
grazie
ad
un
trasportatore. La sintesi degli
ormoni tiroidei avviene nello
spazio follicolare attraverso una
serie di reazioni molte delle quali
sono mediate da perossidasi. Gli
ormoni tiroidei vengono rilasciati
dalla tireoglobulina per idrolisi di
questa
molecola
(Tgb,
tireoglobulina;
MIT,
monoiodotirosina;
DIT,
diiodotirosina; T3, triiodotironina;
T4, tetraiodotironina).
Gli asterischi indicano le tappe o i
processi che risultano alterati per
deficit enzimatici, responsabili del
gozzo congenito e spesso
associati a ipotiroidismo
In vitro:
2I- + 2H+ + 2H2O
I2 + 2H2O
Iodination of tyrosine.
Iodine is oxidized by thyroid peroxidase in a 2eoxidation step, forming I+. The phenolate anion of
tyrosine is in equilibrium with its quinoid form. Iodinium
and tyrosine quinoid react to form an iodinated quinoid
intermediate that forms MIT by electronic
rearrangement. (b) Iodine is oxidized by thyroid
peroxidase in a 1e- step forming a free radical (I0). The
quinoid anion of tyrosine is oxidized to a free radical by
peroxidase or by another I0. The quinoid free radical
reacts with I0 to form MIT by electronic rearrangement.