Principi di genetica - Robert J. Brooker
Copyright © 2010 – The McGraw-Hill Companies srl
Soluzioni ai problemi del Capitolo 13
Domande concettuali
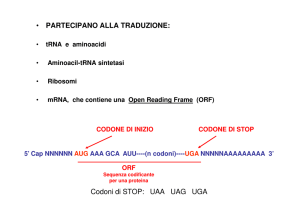
C1. Il codone di inizio parte dal quinto nucleotide. La sequenza aminoacidica sarà Met Gly Asn Lys
Pro Gly Gln STOP.
C2. Quando si dice che il codice genetico è degenerato, si intende che più codoni possono
specificare uno stesso aminoacido. Per esempio, GGG, GGC, GGA e GGU specificano tutti glicina.
In generale il codice genetico è quasi universale, perché viene usato nello stesso modo da virus,
procarioti, funghi, piante e animali. Come viene discusso nella Tabella 13.3, le eccezioni sono
poche e riguardano soprattutto i protisti e i mitocondri di lievito e di mammifero.
C3.
A. Vero
B. Falso
C. Falso
C4.
A. Questo tRNA mutante dovrebbe riconoscere i codoni della glicina nell’mRNA, ma inserirebbe
triptofano nelle posizioni del polipeptide dove ci si attende la glicina.
B. Questa mutazione ci dice che l’aminoacil-tRNA sintetasi riconosce principalmente regioni del
tRNA diverse da quelle dove risiede l’anticodone. Se così non fosse, ti potresti aspettare che la
glicil-tRNA sintetasi riconosca questo tRNA mutante e aggiunga glicina, cosa che non succede.
C5. Come si vede nella Figura 13.7, l’energia deriva dall’ATP. Questo passaggio spiega la
definizione di “tRNA carico”.
C6.
A. La risposta è tre. Ci sono 6 codoni della leucina: UUA, UUG, CUU, CUC, CUA, e CUG.
L’anticodone AAU potrebbe riconoscere UUA e UUG. Avresti bisogno di due altri tRNA per
riconoscere efficientemente gli altri quattro codoni. Questi potrebbero portare gli anticodoni GAG e
GAU oppure GAA e GAU.
Principi di genetica - Robert J. Brooker
Copyright © 2010 – The McGraw-Hill Companies srl
B. La risposta è uno. C’è solo un codone, AUG, per cui hai bisogno di un solo tRNA con
anticodone UAC.
C. La risposta è tre. Ci sono sei codoni della serina: AGU, AGC,UCU, UCC, UCA, e UCG. Ci
sarebbe bisogno di un solo tRNA per riconoscere AGU e AGC. Questo tRNA potrebbe avere
anticodone UCG o UCA. Avresti poi bisogno di altri due tRNA per riconoscere con efficienza gli
altri quattro codoni, e questi potrebbero avere gli anticodoni AGG e AGU oppure AGA e AGU.
C7. Ci sono quattro codoni della prolina, quattro della glicina, uno della metionina e sei della serina.
Applicando la regole del prodotto si risolve il problema:
4 × 4 × 1 × 6 = 96
C8. 3'–CUU–5' or 3'–CUC–5'
C9. Il codone è 5’-CCA-3’, che specifica per prolina.
C10. Può riconoscere 5'–GGU–3', 5'–GGC–3', e 5'–GGA–3'. Tutti specificano glicina.
C11. Un anticodone 3’-UUG-5’ riconoscerebbe due codoni. Per riconoscere 5’-AAA-3’ esso
dovrebbe essere modificato in 3’-UUI-5’.
C12. Tutte le molecole di tRNA hanno delle caratteristiche in comune. Essi hanno tutti una struttura
a trifoglio con tre anse. La seconda ansa contiene la sequenza dell’anticodone, che riconosce il
codone nell’mRNA. All’estremità 3’ c’è il braccio accettore, con sequenza CCA, che funge da sito
di aggiunta dell’aminoacido. La maggior parte dei tRNA contengono anche delle basi modificate
nelle loro sequenze nucleotidiche.
C13. Si trovano molto distanti, alle estremità opposte della molecola.
C14. Il ruolo dell’aminoacil-tRNA sintetasi è quello di riconoscere specificamente le molecole di
tRNA e aggiungervi il corretto aminoacido. Questa capacità viene definita talvolta secondo codice
genetico perché la specificità dell’aggiunta è un passo cruciale nella lettura del codice genetico. Per
esempio, se il tRNA ha un anticodone 3'–GGG–5', esso riconosce il codone 5'–CCC–3' che
specifica prolina. E’ essenziale che la prolil-tRNA sintetasi riconosca questo tRNA e aggiunga la
Principi di genetica - Robert J. Brooker
Copyright © 2010 – The McGraw-Hill Companies srl
prolina alla sua estremità 3’. Le altre aminoacil-tRNA sintetasi non dovrebbero riconoscere questo
tRNA.
C15. Nel contesto della traduzione, un aminoacido attivato ha avuto un AMP legato ad esso. Questo
ha fornito l’energia necessaria per l’aggiunta dell’aminoacido al corretto tRNA.
C16. Le basi che sono state modificate chimicamente possono trovarsi in varie posizioni della
molecola di tRNA. Il significato di queste modificazioni non è del tutto noto. Tuttavia all’interno
dell’anticodone la modificazione delle basi altera l’appaiamento, e consente a uno stesso anticodone
di riconoscere due o più codoni diversi.
C17. Un gruppo formile viene legato covalentemente alla metionina dopo che la metionina è stata a
sua volta aggiunta al tRNA che contiene l’anticodone UAC.
C18. Non lo è. A causa delle regole del vacillamento, in un tRNA la base al 5’ dell’anticodone può
riconoscere due o più basi nella terza posizione (3’) dell’mRNA. Perciò, ogni tipo cellulare
sintetizza molto meno di 61 tipi di tRNA.
C19. La traduzione ha bisogno di mRNA, tRNA, ribosomi, proteine quali fattori di inizio, e molte
piccole molecole. ATP e GTP sono piccole molecole contenenti legami ad alta energia. Gli mRNA,
tRNA e le proteine sono macromolecole. I ribosomi sono grandi complessi di macromolecole.
C20. Il processo di assemblaggio è molto complesso a livello molecolare. Negli eucarioti, per
formare la subunità 40S servono 33 proteine e un rRNA. Per formare la subunità 60S servono 49
proteine e tre rRNA. L’assemblaggio avviene nel nucleolo.
C21. La subunità di una proteina è un polipeptide Una subunità ribosomale è un complesso molto
più grande che è composto di RNA e molte proteine. Una subunità ribosomale è una struttura molto
più grande in confronto alla subunità proteica.
C22.
A. Sulla superficie della subunità 30S e all’interfaccia tra le due subunità.
B. Dentro la subunità 50S.
C. Dalla subunità 50S.
Principi di genetica - Robert J. Brooker
Copyright © 2010 – The McGraw-Hill Companies srl
D. Con la subunità 30S.
C23.
Inizio: L’mRNA, il tRNA iniziatore e i fattori di inizio si associano con la subunità ribosomale
minore. Poi si associa la subunità maggiore.
Allungamento: Il ribosoma si sposta di un codone alla volta lungo l’mRNA; aggiungendo
aminoacidi alla catena polipeptidica.
Terminazione: Viene raggiunto un codone di stop e un fattore di terminazione si lega al sito A.
L’idrolisi del GTP avvia una serie di eventi che conducono al disassemblaggio del ribosoma e al
rilascio della catena polipeptidica.
C24. Molti mRNA batterici contengono una sequenza di Shine-Dalgarno, necessaria per il legame
dell’mRNA alla subunità minore del ribosoma. Questa sequenza, UUAGGAGGU, è complementare
a quella dell’rRNA 16S. A causa della complementarietà queste sequenze formeranno legami
idrogeno tra di loro durante la fase di inizio della traduzione
C25.
A. Il tRNA iniziatore non si legherebbe alla subunità ribosomale 40S.
B. L’mRNA non si legherebbe alla subunità ribosomale 40S, e/o il codone di inizio non sarebbe
riconosciuto perché la struttura secondaria dell’mRNA non sarebbe stata aperta.
C. La subunità 60S non si assemblerebbe dopo l’identificazione del codone di inizio.
C26. Il ribosoma si lega all’estremità 5’ dell’mRNA e poi inizia la scansione in direzione 3’ in cerca
del codone di inizio AUG. Se ne trova uno che ragionevolmente ubbidisce alle regole di Kozak, la
traduzione avrà inizio da quel sito. A parte il codone di inizio AUG, altre due caratteristiche
importanti sono la guanosina in posizione +4 e una purina in posizione –3.
C27.
1. GCCACCAUGG
2. GACGCCAUGG
3. GCCUCCAUGC
4. GCCAUCAAGG
L’ultima sequenza non ha un codone di inizio, per cui non potrebbe funzionare. La terza verrebbe
tradotta, ma debolmente.
Principi di genetica - Robert J. Brooker
Copyright © 2010 – The McGraw-Hill Companies srl
C28. Il sito A (aminoacilico) è la posizione in cui si lega un tRNA che porta un aminoacido. La sola
eccezione è il tRNA iniziatore, che si lega al sito P (peptidilico). La catena polipeptidica nascente
viene rimossa dal tRNA nel sito P, e trasferita all’aminoacido attaccato al tRNA nel sito A. Il
ribosoma trasloca nella direzione 3’, con il risultato che i due tRNA nei siti P e A si spostano
rispettivamente nei siti E (exit) e P, e il tRNA privo di aminoacido nel sito E viene rilasciato.
C29. La sequenza aminoacidica è: metionina-tirosina-glicina-alanina. La metionina è alla estremità
N-terninale, l’alanina a quella C-terminale. I legami peptidici sono come in Figura 13.3b.
C30. La fase di inizio implica il legame della sequenza di Shine-Dalgarno all’rRNA nella subunità
30S. La fase di allungamento implica il legame degli anticodoni del tRNA ai codoni dell’mRNA.
C31. Il nucleolo è la regione del nucleo eucariotico dove avviene l’assemblaggio delle subunità
ribosomali.
C32.
A. Il sito E e il sito P. Nota che un tRNA privo di aminoacido si trova solo brevemente nel sito P,
subito prima che avvenga la traslocazione.
B. Il sito P e il sito A. Nota che un tRNA con una catena polipeptidica attaccata si trova solo
brevemente nel sito A, subito prima che avvenga la traslocazione.
C. Di solito il sito A, a parte il tRNA iniziatore che si trova nel sito P.
C33. Un polisoma è una molecola di mRNA batterico con molti ribosomi legati ad esso.
C34. I tRNA si legano all’mRNA perché i loro anticodoni sono complementari ai codoni. Quando il
ribosoma trasloca in direzione 5’→ 3’, i tRNA restano legati ai loro codoni complementari, e i due
tRNA si spostano dal sito A al sito P e dal sito P al sito E. Se il ribosoma si muovesse nella
direzione opposta, il tRNA perderebbe la propria posizione e andrebbe a trovarsi in una nuova
posizione non necessariamente complementare all’mRNA.
C35.
A. Falso
B. Vero
Principi di genetica - Robert J. Brooker
Copyright © 2010 – The McGraw-Hill Companies srl
C36. 52
C37. Significa che la traduzione può iniziare prima che la sintesi dell’mRNA sia stata completata.
Questo non avviene nelle cellule eucariotiche perché la trascrizione e la traduzione avvengono in
compartimenti distinti. Nelle cellule eucariotiche la trascrizione avviene nel nucleo, la traduzione
nel citoplasma.
Domande sperimentali
S1. Un codone contiene tre nucleotidi. Siccome G e C sono presenti al 50% ciascuno, se
moltiplichiamo 0,5 x 0,5 x 0,5 otteniamo un valore di 0,125 ossia 12,5% per ciascuno dei possibili
otto codoni. Se consultiamo la Tabella 13.2 ci attendiamo il 25% di glicina (GGG e
GGC), il 25% di alanina (GCC e GCG), il 25% di prolina (CCC e CCG), e il 25% di arginina
(CGG e CGC).
S2.
A. Ci potrebbero essere state altre scelte, ma questo stampo avrebbe dovuto contenere un codone
della cisteina, UGU, e nessuno dell’alanina.
B. Non potresti usare
35
S perché la marcatura sarebbe rimossa durante i trattamenti con il nickel
Raney.
C. Non ci sarebbe una quantità significativa di radioattività incorporata nel polipeptide di nuova
sintesi, con o senza trattamento con nickel Raney. Il solo aminoacido marcato nell’esperimento era
la cisteina, che viene aggiunta al tRNAcys. Quando vengono esposte al nickel Raney, queste cisteine
sono convertite in alanina ma solo dopo essere state aggiunte al tRNAcys. Se non ci fossero codoni
della cisteina nello stampo di mRNA, il tRNAcys non lo riconoscerebbe. Perciò non ci aspettiamo di
trovare molta radioattività nei polipeptidi neosintetizzati.
S3. La treonina è stata sostituita da serina. Sulla base delle loro strutture, deve essere avvenuta la
demetilazione della treonina. In altre parole il gruppo metilico è stato sostituito da un idogeno.
S4. La fase di inizio della traduzione è molto diversa nei batteri e negli eucarioti. Un mRNA
batterico non sarebbe tradotto molto efficientemente in un sistema eucariotico, perché non possiede
il cappuccio al 5’. Un mRNA eucariotico non avrebbe la sequenza di Shine-Dalgarno alla sua
estremità 5’, per cui non sarebbe tradotto molto efficientemente in un sistema procariotico.
Principi di genetica - Robert J. Brooker
Copyright © 2010 – The McGraw-Hill Companies srl
S5. Capire la struttura del ribosoma può essere di grande aiuto per comprendere il processo della
traduzione. Per esempio il sequenziamento dell’rRNA 16S batterico ha dimostrato l’esistenza della
sequenza complementare a quella di Shine-Dalgarno. Ciò ha aiutato i biologi molecolari a capire
che l’mRNA si lega inizialmente alla subunità minore del ribosoma.
S6. Guardando alla figura, l’estremità 5’ dello stampo di DNA è verso la destra. L’estremità 5’
dell’mRNA è la più lontana dal DNA; e la 3’ la più vicina. I codoni di inizio sono a valle
dell’estremità 5’ dell’RNA.
S7.
S8.
S9. Il campione della corsia 2 deriva da un soggetto omozigote per una mutazione che ha introdotto
un codone di stop nella sequenza codificante. Come si vede nella corsia 2, la proteina è più corta di
quella normale. Il campione nella corsia 3 deriva da un soggetto omozigote per una mutazione che
impedisce l’espressione del polipeptide. Potrebbe essere una mutazione nel promotore che riduce
l’efficienza della trascrizione, oppure una mutazione nella sequenza codificante che provoca la
rapida degradazione della proteina. Il campione nella corsia 4 deriva da un individuo omozigote per
una mutazione che ha prodotto una sostituzione aminoacidica, ossia una mutazione di senso. Tali
Principi di genetica - Robert J. Brooker
Copyright © 2010 – The McGraw-Hill Companies srl
mutazioni non sono sempre rilevate su gel, ma alterano la funzione della proteina, cosa che
potrebbe spiegare il fenotipo albino.
S10. A. Se l’utilizzo dei codoni fosse significativamente diverso tra cellule di canguro e di lievito,
questo inibirebbe il processo di traduzione. Per esempio, se nel canguro il codone preferito fosse
CUU, la traduzione probabilmente non procederebbe nel sistema di lievito. Ci aspettiamo che il
sistema in vitro di lievito contenga soprattutto tRNA della leucina con anticodone AAC, perché
questo tRNA riconosce il codone preferito per la leucina nel lievito, che è UUG. Nel sistema di
traduzione di lievito non ci sarebbe probabilmente una grande quantità di tRNA con l’anticodone
GAA, che riconoscerebbe il codone preferito dei canguri, CUU. Per questo motivo l’mRNA di
canguro non sarebbe tradotto appropriatamente nel sistema in vitro anche se in qualche misura
sarebbe comunque tradotto.
B. Il vantaggio dell’uso preferenziale dei codoni è che una cellula può contare su una popolazione
più piccola di tRNA per tradurre con efficienza le proteine. Uno svantaggio è che le mutazioni, che
non cambiano la sequenza di aminoacidi ma cambiano un codone (per esempio UUG in UUA), può
inibire la produzione di un polipeptide se il codone preferenziale viene cambiato in un codone non
preferenziale.