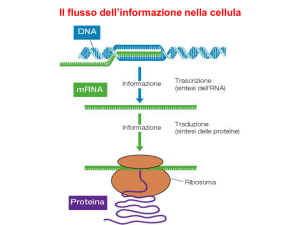
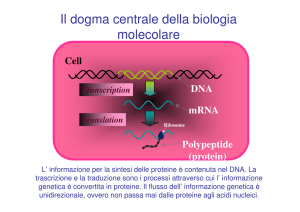
Il flusso dell’informazione:
l’espressione genica
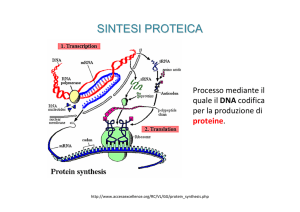
La traduzione
L’espressione genica: la “messa in atto”
dell’informazione contenuta nel DNA
La traduzione (sintesi proteica)
è il processo energeticamente
più dispendioso per la cellula
batterica.
Bersaglio di numerosi
antibiotici.
Struttura del gene
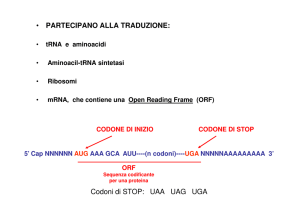
• Gene
– Unità base dell’informazione genica
– Sequenza di DNA che codifica per un polipeptide
oppure per un tRNA o un rRNA
– Sequenza nucleotidica definita da un punto di
inizio e uno di fine (per geni codificanti per
proteine codoni di inizio e di fine)
– Negli mRNA ogni codone corrisponde ad un dato
amino acido
• Fase di lettura (reading frame)
– Ogni elica possiede 3 fasi possibili di lettura, dei
quali solo una corrisponde alla corretta
informazione del gene
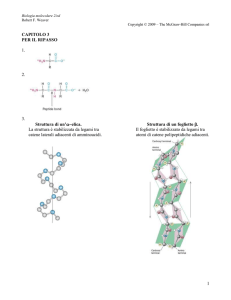
La trascrizione è un processo
„asimmetrico“
Template strand/elica stampo
b
w
b‘
a
Coding strand/elica senso
Importanza della fase di lettura
Geni che codificano per proteine
(polipeptidi)
• L’elica stampo (template strand) è usata per la sintesi
dell’RNA messaggero
– È letta 3’-5’ e consente la sintesi di un mRNAs 5’-3’ con
sequenza nucleotidica complementare all’elica stampo ed
“identica” alla sequenza dell’elica senso
– Il gene termina con un codone di stop
• Il trascritto continua fino ad una sequenza di terminazione
• Il trascritto possiede una sequenza leader trascritta ma
non tradotta
La trascrizione (DNA>RNA) è
comunque finalizzata alla traduzione
I tre tipi di RNA trascritti
mRNA (RNA messaggero): contiene la sequenza
nucleotidica del gene per la sua conversione a proteina. La
sua sequenza nucleotidica viene letta in triplette secondo il
codice genetico
tRNA (RNA transfer): RNA stabile, specifico per ogni
codone “senso”, viene caricato con l’amino acido
corrispondente
rRNA (RNA ribosomale): circa il 90% di tutto l’RNA presente
nella cellula. Costituisce con una cinquantina di proteine il
ribosoma, su cui avviene la sintesi proteica
Il Codice Genetico
• colinearità
– Corrispondenza tra sequenza nucleotidica nel DNA e sequenza
amino acidica nella proteina
– Codoni
Unità elementari del codice genetico, corrispondono ad un
aminoacido
Decifrato da Marshall Nirenberg negli anni 60
L’universalità del codice genetico è considerata, assieme all’uso
del DNA come molecola per la conservazione del materiale
genetico, la prova principale dell’evoluzione di tutti gli organismi
viventi da un unico progenitore comune
Il codice genetico
E’ “degenerato”…..
(più di un singolo codone può avere lo stesso significato)
Il codice genetico
I codoni che specificano per aminoacidi sono detti codoni “senso”
I tre codoni non senso (UAA, UAG, UGA) segnalano la fine della traduzione
Che cosa consente che ad un codone
corrisponda un dato aminoacido?
3’
5’
Il tRNA.
Sequenza nucleotidica mRNA (es. GGC, codifica per
glicina)
5’
3’
Esistono 61 molecole diverse di tRNA, ognuna in grado
di legare i codoni senso complementari tramite la loro
sequenza anticodone
All’estremità 3’ del tRNA viene attaccato l’amino acido
corrispondente al codone (nell’esempio, la glicina)
Il tRNA ha una struttura complessa con
tratti a doppio filamento
Regioni a doppio
filamento
Complementare al
codone nell’ mRNA
Sito a cui viene attaccato
l’amino acido
“Peculiarità” dell’RNA transfer
• Presenza di regioni a doppio filamento
• Presenza di un legame esterico aa-nt
• Presenza di basi azotate modificate
(metiluridina(meU), pseudouridina(y))
Aminoacil-tRNA Sintetasi
Legame esterico
Aminoacido
attivato
(aa-AMP)
tRNA
La aa-tRNAs sintetasi (21 per cellula)
“Wobble”
• Scarsa fedeltà
nell’appaiamento
delle basi
– 3° posizione del
codone è meno
importante delle
prime due
• Permette ad un
unico tRNA di
interagire con più
codoni che
codificano per lo
stesso aa
Lo stesso tRNAs può interagire con due
diversi codoni per la glicina
Il ribosoma: sede e “cervello” della
sintesi proteica
• Il ribosoma è un
complesso costituito da
due subunità distinte e
composto da rRNA (3
molecole) e proteine
(circa 55)
• Nelle cellule procariote
rappresenta la struttura
citoplasmatica più
complessa
Inizio della sintesi proteica
• Coinvolge le
subunità ribosomali
• tRNA d’inizio
• Fattori di inizio
(IFs)
Inizio della sintesi proteica
Il tRNA iniziatore è caricato con
formil-metionina
Archea ed eucarioti
usano tRNA a metionina
Inizio della sintesi proteica
• Il posizionamento del
tRNA d’inizio ha luogo
ad un codone AUG
• Il riconoscimento del
ribosoma di un codone
AUG di inizio è favorito
dalla presenza di una
sequenza detta di
Shine-Dalgarno
…AGGAGG……AUG
5-8 nt
I tre siti di legame per il tRNA sul
ribosoma
• Al termine del ciclo di inizio il
ribosoma si è ricostituito e vi
sono riconoscibili tre siti di
legame per il tRNA
• peptidil-tRNA (sito P)
– E’ il sito che lega il tRNA d’inizio e
il tRNA attaccato alla catena
peptidica
• Sito dell’aminoacil-tRNA (sito
A)
– Dove entrano i nuovi aa-tRNA
• Sito di uscita (Exit site)
– Dove si posizione il tRNA scarico
L’allungamento (elongazione) della
sintesi proteica
•
•
•
•
I successivi aa-tRNA vengono
posizionati nel sito A del ribosoma
da una proteina specifica detta
EF-Tu ( idrolisi 1 GTP)
L’EF-Tu resta legato al GDP ma
viene ricaricato a EF-Tu/GTP da
un altro fattore, EF-Ts.
L’azione della peptidil-trasferasi
del ribosoma consente il
trasferimento dell’aa (o catena
peptidica) dal sito P all’ aa-tRNA
del sito A
La catena peptidica nascente
viene traslocata al sito P tramite
l’intervento di un altro fattore, EFG (idrolisi di 1 GTP)
La fase di allungamento della sintesi proteica è il
bersaglio di un grande numero di antibiotici
• Peptidil-trasferasi: Puromicina,
cloramfenicolo
• EF-Tu: kirromicine, pulvomicine
• EF-G: acido fusidico
• Ribosoma: tetraciclina, streptomicina,
kanamicina
La terminazione avviene in
corrispondenza dei codoni di stop
• Richiede I fattori di
rilascio (RF)
– Permettono il
riconoscimento dei
codoni di stop
– 3 RFs nei procarioti
– solo 1 RF attivo negli
eucarioti
Trascrizione e traduzione sono
contemporanee nei procarioti
Step 2: dalla trascrizione alla proteina
Gli enzimi sono attivi solo se ripiegati
nella conformazione corretta (folding)
Nel processo di folding hanno luogo
modificazioni post-traduzionali:
es. Folding ossidativo
Soltanto il corretto appaiamento di
residui di cisteina porta alla
conformazione biologicamente attiva
DnaK
„Mettersi in riga“ o venire eliminati: il ruolo delle chaperonine
Struttura del complesso GroEL/GroES
(una della maggiori chaperonine citoplasmatiche)
A) Veduta laterale
B) Veduta apicale e basale
Nel citoplasma troviamo anche….
• Sostanze di riserva immagazzinate in
corpi di inclusione
• Es.:
– Granuli di glicogeno (polimero di glucosio)
– Poli-idrossi-alcanoati (PHA)
– “Bolle di gas”