SELEZIONE ASSISTITA CON MARCATORI MOLECOLARI
Che cosa è un marcatore genetico?
E’ un fenotipo (una qualsiasi caratteristica morfologica) strettamente associato al
carattere di interesse, che consente di selezionare piante che portino il carattere di
interesse
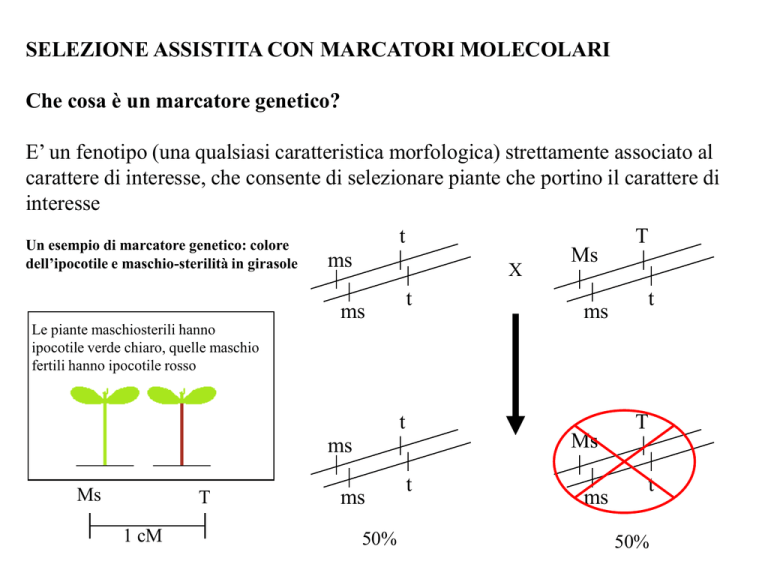
Un esempio di marcatore genetico: colore
dell’ipocotile e maschio-sterilità in girasole
t
ms
X
t
ms
Ms
ms
T
t
Le piante maschiosterili hanno
ipocotile verde chiaro, quelle maschio
fertili hanno ipocotile rosso
t
Ms
ms
Ms
T
1 cM
ms
50%
t
ms
T
t
50%
Che cosa sono i marcatori molecolari?
Un marcatore molecolare è una sequenza di
DNA o una proteina facilmente
analizzabile, la cui eredità da una
generazione all’altra possa essere
monitorata. Un pattern di marcatori
molecolari è un vero e proprio “fenotipo”
Perché sono necessari i marcatori
molecolari?
I fattori ambientali possono influenzare le
caratteristiche morfologiche
I marcatori molecolari sono espressione diretta
del materiale genetico e consentono di valutare
le variazioni genetiche
I marcatori molecolari possono essere usati per:
misurare la diversità genetica
mappature genetiche
selezione di germoplasma in programmi di miglioramento genetico
Polimorfismo delle proteine:
Polimorfismo del DNA:
proteine di riserva del seme
isoenzimi
nucleare
citoplasmatico
Caratteristiche desiderabili di un marcatore molecolare
Polimorfico
A eredità codominante
Che copre tutto il genoma
Facile, veloce ed economico da studiare
Riproducibile
Trasferibile ad altri genotipi della stessa specie
Nessun singolo
marcatore
risponde a tutte
queste
caratteristiche
Esempio di codominanza: superossido dismutasi di girasole
MARCATORI MOLECOLARI (a DNA)
Basati sull’amplificazione
mediante PCR
Basati su enzimi di restrizione,
Southern blot e ibridazione
AFLP
SNP
MARCATORI BASATI SUGLI RFLP
Gli RFLP esaminano differenze nella taglia di specifici frammenti di restrizione del
DNA; consentono di osservare variazioni anche minime della sequenza del DNA;
le variazioni sono ereditate come caratteri mendeliani e possono quindi funzionare
come marcatori genetici, anche di caratteri quantitativi. Si realizzano (di solito) sul
DNA genomico totale. Richiedono DNA purificato, ad alto peso molecolare.
Metodologia:
tagliare il DNA in frammenti
separarli per elettroforesi su gel
trasferirli su membrana
Differenze nei pattern di restrizione a seconda dell’enzima usato
Differenze di sequenza evidenziate da un enzima di restrizione
1
2
3
4
5
6
MARCATORI BASATI SULLA PCR
La “polymerase chain reaction” serve per amplificare specifiche sequenze
di DNA utilizzando cambiamenti ciclici delle temperature di reazione:
94°C per la denaturazione
55°C per l’appaiamento (annealing) dell’oligonucleotide (primer)
72°C per la sintesi (extension) del DNA con l’uso della Taq Polymerase
Ad ogni ciclo raddoppia
il numero di frammenti
prodotti
Dopo l’elettroforesi, il numero delle bande ottenibili in un individuo dipende dal tipo di primer:
1) Aspecifici
Es.: R.A.P.D.
(primer lunghi 1015 nt, aspecifici)
2) Specifici (si legano ad una sequenza nota, specifica, del genoma)
[lunghezza > di 18 nt]:
2a) specifici per sequenze ripetute (es. retrotrasposoni)
2b) specifici per sequenze a singola copia (es. geni)
multilocus (il numero di
bande dipende dal
numero di ripetizioni nel
genoma)
single locus (danno 1 banda
negli omozigoti e due negli
eterozigoti [per la lunghezza
del frammento])
MARCATORI SINGLE LOCUS: MICROSATELLITI
-Microsatelliti (simple sequence repeats = SSR):
utilizzano primers progettati su sequenze fiancheggianti i microsatelliti
evidenziano variazioni di lunghezza dei microsatelliti (o assenza dei primers)
ACGTCTGCTAATCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCGGGTCTGGA
ACGTCTGCTAATCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCTCGGGTCTGGA
ACGTCTGCTAATCTCTCTCTCTCTCTCTCTCTCTCGGGTCTGGA
Ind.#1 100101
Ind.#2 011011
Ferogrammi relativi alle lunghezze dei loci VVMD6, VVMD17,
VVMD21 VVMD24 di tre genotipi di vite: Chianti 2000/1, Nielluccio
189 e V. champini.
Lunghezze dei frammenti di DNA amplificati (in bp) con 10 microsatelliti in 22 accessioni di vite; per ogni locus è
stato riportato il valore relativo ai due alleli. Dove non è stato riportato nessun valore, le coppie di primers non hanno
dato nessun prodotto di amplificazione.
AFLP (Amplified-Fragment-Lenght-Polymorphism)
Una combinazione di RFLP e PCR
Procedura:
1)digestione con enzimi di restrizione
2)attacco alle estremità dei frammenti di adattatori per l’enzima usato +
sequenza specifica
3)PCR con primers omologhi alla sequenza specifica
MARCATORI SNP
I marcatori molecolari possono essere trattati come geni mendeliani
(es. F2 di un diibrido)
… e utilizzati per fare mappe genetiche
Mappa genetica-citologica-molecolare di
Medicago truncatula
MARKER ASSISTED SELECTION (MAS) PER CARATTERI
QUANTITATIVI
I caratteri quantitativi sono determinati da componenti genetiche ed ambientali. Il
fenotipo può essere molto “distante”da quelle che sono le sue componenti
genetiche.
La selezione è basata su metodi biometrici (medie e varianze) e spesso non può
stimare con precisione sufficiente le componenti genetiche.
Con i marcatori molecolari si può cercare di ricondurre i caratteri quantitativi a
caratteri (quasi) qualitativi, facilitando così la selezione.
Q.T.L. = quantitative trait locus
“locus controlling part of the phenotypic variation of a quantitative trait”
(Gelderman, 1975)
I QTL sono loci (posizioni) sul cromosoma che individuano le sequenze
coinvolte nella determinazione di caratteri quantitativi, NON
NECESSARIAMENTE codificanti prodotti genici precisi.
Questi loci possono individuare regioni cromosomiche in cui si hanno:
1) Geni maggiori, che hanno particolare importanza nella determinazione del
carattere
2) Gruppi di geni coinvolti nello stesso carattere quantitativo e strettamente
vicini sul cromosoma
Il QTL è una sequenza di DNA che identifica la posizione del carattere
quantitativo in virtù della sua ASSOCIAZIONE (più o meno stretta) a geni che
controllano quel carattere.
In questo senso, può essere costituita da DNA genico, codificante, o anche non
codificante (che individua segmenti intergenici).
UN QTL ANTE LITTERAM
Sax K. (1923) The association of size differences with seed-coat pattern and
pigmentation in Phaseolus vulgaris.
Gli individui vengono misurati per il carattere quantitativo: il QTL viene
evidenziato se si osserva una differenza significativa tra le medie fenotipiche
delle sottopopolazioni x, y e z LD (Linkage Disequilibrium)
A LIVELLO MOLECOLARE:
Se dalla F2 faccio una serie di autofecondazioni, alla fine raggiungerò in tutti
gli individui l’omozigosi, con questi quattro possibili genotipi:
RECOMBINANT INBRED LINES (R.I.L.)
A = alta
B = bassa
A
B
A
B
B
A
A
B
A
A
A
Esempio: Single Marker Analysis
(SMA) - ANOVA
1) Individui divisi in due classi genotipiche per tutti i marcatori disponibili (Aa vs aa)
2) ANOVA o t-test per vedere se le medie fenotipiche sono diverse (ipotesi H0:
µaa) - differenza = (µAa – µaa) (1-2r)
3) Effetto come semplice differenza delle medie:
d = µAa – µaa
yij = µ + αi + Eij
Dove:
yij: valore fenotipico dell’individuo j per la classe genotipica i
µ: media fenotipica
αi : effetto del locus marcatore di genotipo i (che può essere scomposto in effetto
additivo e di dominanza)
Eij: variabile residuale
µAa =
dove:
M: marcatore (fattore)
E: fattore residuale
ni: numero di individui di genotipo i (j = 1,2,…, ni)
g: numero di genotipi possibili
N: numero totale di individui della discendenza
Il valore di questo rapporto viene confrontato con i
valori soglia della distribuzione F per (g-1) e (N-g) gradi
di libertà e un rischio α (corretto secondo Bonferroni)
PEV (proporzione della varianza fenotipica
dovuta a quel particolare QTL)
LOD (z) = log10 del rapporto di verosimiglianza tra l’ipotesi di presenza di un QTL
in una posizione z e quella di assenza del QTL
sono le stime di massima verosimiglianza sotto l’ipotesi di un solo
QTL nella posizione z
Nel caso di assenza di QTL, i fenotipi sono indipendenti con
distribuzione identica N(µ, )
Come si stabilisce la
soglia di LOD? Ad ogni
valore di LOD dobbiamo
associare un rischio α (o
“p-value”) che ci dia
un’idea della significatività statistica del QTL (in
genere al 5% di ogni
esperimento)
Quali dati si ottengono da una mappatura di QTL?
1) Numero e posizione dei QTL: quanti QTL controllano il carattere e
dove sono
2) Livello di significatività e intervallo di confidenza sulla posizione
di ognuno di essi
3) Effetto positivo o negativo sul fenotipo (cioè se la sostituzione
allelica fa aumentare o diminuire il valore del fenotipo)
4) Effetto sul fenotipo: PEV (proporzione della varianza fenotipica
dovuta a quel particolare QTL)
5) Effetti epistatici (interazione fra QTL e pleiotropia)
“Genome-scan”: il test di LOD viene ripetuto su tutto il genoma
Risultato di una mappatura di un QTL
Dai marcatori molecolari indeterminati si può passare a marcatori basati sui geni e
infine agli SNP (Single Nucleotide Polymorphisms) per ottenere i QTN (Quantitative
Trait Nucleotides)
I caratteri complessi vengono scomposti nei singoli fattori mendeliani che li determinano
Esempi di QTL correlati con geni nelle piante (Paran e Zamir, 2003)
Un esempio di QTL (resistenza al freddo in orzo)
Map positions of some dehydrin genes
in barley. Traditional barley
chromosome designations are shown in
parentheses below the Triticeae
genome designations. The map
positions of genes detected by
Himalaya probes dhn1 through dhn6
are indicated. Loci detected by dhn1
and dhn2 (red bar) on linkage group
5H are within the 99% confidence
interval (yellow box) of a QTL
contributing to winterhardiness,
photoperiod response, and heading
date in a number of studies (Pan et al.,
1994). This region of the group 5
chromosomes also houses the barley
Sh2 locus, which is a determinant of
winter or spring growth habit.
A cosa può corrispondere un QTL - Il locus tb1 di mais
Doebley et al. (1997): il mais è molto diverso morfologicamente dal teosinte (Zea mays ssp.
parviglumis, o Zea mays ssp. mexicana, suo progenitore selvatico) ma è molto simile nella struttura
dei geni nei cromosomi e nelle sequenze nucleotidiche ed è perfettamente incrociabile con il teosinte.
Si suppone quindi che il mais si sia originato dal teosinte per mutazione in 5 loci maggiori, fra cui:
gene tga1 su cromosoma 4 - gene tb1 su cromosoma 1
(a) pianta di teosinte (Zea mays ssp mexicana) con
una branca laterale primaria (b) mostrante fiori
maschili terminali (T) e sete (S) che emergono dalle
foglie che avvolgono la pannocchia. (c) pianta di
mais selvatico, infiorescenza femminile laterale
selvatica (d). Una pianta mutante di tipo tb1(e) con
le branche laterali che portano anch’esse i fiori
maschili, ma non la spiga femminile (f)
Il locus tb1 di mais
È stato accertato che l’effetto fenotipico del gene tb1 (teosinte branched 1) su diversi
caratteri della spiga dipende soprattutto da una regione cis regolatoria a monte di oltre
60 Kb, diverse nelle varie linee anche per la presenza di elementi retrotrasponibili.
Il locus tb1 di mais. Linee di
introgressione di teosinte in
mais con diversa struttura nel
locus tb1. Aspetti della
sequenza sono indicati in
alto; gli effetti
dell’introgressione sono
indicati a destra. La regione
critica che influenza il
branching basale e alcuni
caratteri della spiga è
indicata al centro con la
colonna in grigio (Clark et
al. 2006).
QTL di resistenza a lepidotteri in mais
Putative chromosomal cluster
containing quantitative trait loci
(blue ovals) involved in corn borer
resistance of temperate and tropical
maize with genes involved in cell
wall fortification.
RIASSUMENDO:
Cosa occorre per fare una mappatura di un QTL:
1) Popolazioni derivate da incroci controllati
(F2, BC, RIL, NIL e altre)
2) Mappa di associazione
3) Indice fisiologico, biochimico ecc., espressione del carattere
quantitativo di interesse da misurare in ogni individuo della progenie
dell’incrocio controllato
4) Analisi statistiche: a) per la ricerca di marcatori molecolari legati
geneticamente a QTL per il carattere quantitativo analizzato; b) per la
stima dei parametri genetici dei QTL individuati
Quanto deve essere “accurato” un QTL? Cioè, quanto deve essere stretta la sua
associazione al carattere?
Linkage disequilibrium in funzione dell’età della popolazione o della specie
A seconda del livello di linkage disequilibrium possiamo identificare i QTL con
dei marcatori molecolari oppure è necessario analizzare dei geni “candidati”
Approccio dei geni candidati
Identificazione di geni candidati
CGs
Screening dei geni candidati mediante:
a) Studi di associazione in popolazioni naturali tra polimorfismi del GC
e variabilità del carattere
b) Analisi della co-segregazione con eventuali QTL che controllano
parte della variabilità del carattere
Verifica dei geni candidati mediante mappature comparative, analisi
fisiologiche e (se possibile) trasformazione.












