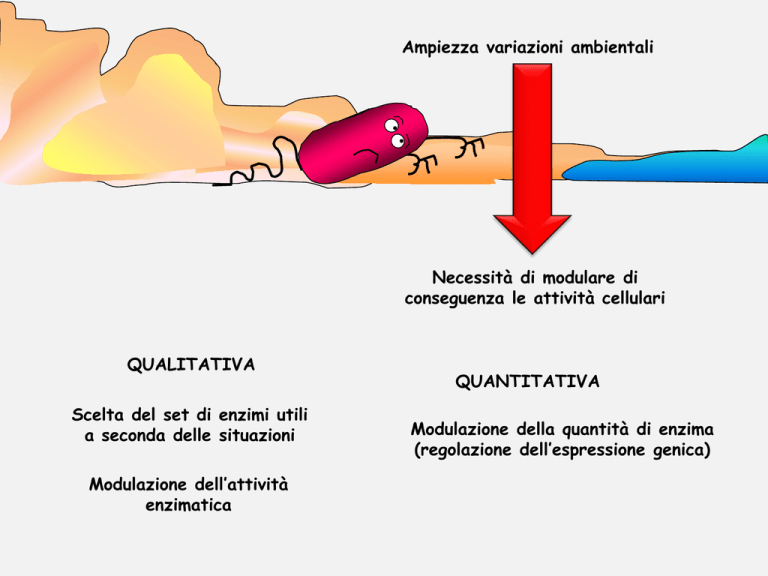
Ampiezza variazioni ambientali
Necessità di modulare di
conseguenza le attività cellulari
QUALITATIVA
Scelta del set di enzimi utili
a seconda delle situazioni
Modulazione dell’attività
enzimatica
QUANTITATIVA
Modulazione della quantità di enzima
(regolazione dell’espressione genica)
MODULAZIONE DELL’ATTIVITÀ ENZIMATICA
le proteine possono cambiare conformazione
ESSERE INIBITE
ESSERE ATTIVATE
ACQUISIRE CAPACITA’
NUOVE (LATENTI)
ATTRAVERSO IL LEGAME
CON EFFETTORI
ALLOSTERICI (LIGANDI)
positivi
negativi
Il sito di legame per l’effettore non
coincide con il sito catalitico dell’enzima
Il legame con
l’effettore modifica
Velocità di reazione
(Vmax)
Affinità per il substrato
(Km)
REGOLAZIONE DELL’ATTIVITA’
ENZIMATICA
Inibizione a
FEED BACK
Se il prodotto finale di una biosintesi è in eccesso o se è
disponibile nell’ambiente, il processo non è più conveniente
QUANDO IL PRODOTTO
SI ACCUMULA
la reazione enzimatica cessa
e non si forma più prodotto
si lega a uno degli enzimi della
catena, causandone l’inattivazione
Non tutti i geni sono espressi
nello stesso modo
Enzimi particolari
Poco espressi
Proteine house-keeping: molto espresse
Regolazione dell’espressione genica
Facilitarla (regolazione positiva)
regolazione della trascrizione
Adattarsi variando la quantità di
mRNA disponibile per la traduzione in
proteine significa risparmio energetico
Ostacolarla (regolazione negativa)
Evita sintesi inutili di
nucleotidi e proteine
Regolazione positiva
Attivatore: se promuove il
legame della RNA-polimerasi
la trascrizione avviene
Regolazione negativa
Repressore: se si
lega al promotore
la trascrizione non
avviene
Stimoli ambientali possono modificare l’attivatore, ottenendo una forma attiva,
attraverso un SISTEMA A DUE COMPONENTI
il sensore cambia conformazione e
manifesta una capacità enzimatica prima
latente (proteina-chinasi)
sensore
+
segnale
sensore
Una proteina sensore si lega alla
molecola segnale (STIMOLO)
sensore
P-K
P
P-K
Si autofosforila
P
+
E trasferisce il fosfato al
regolatore, attivandolo
Il regolatore (effettore) è ora in grado di
legarsi a una sequenza specifica al 5’ del gene
da regolare, attivando la trascrizione
L’attivatore viene poi
disattivato da una fosfatasi
P
P-asi
In questo modo la cellula può rispondere a nuovi stimoli e
cessa di rispondere quando lo stimolo viene a mancare
Nella maggior parte dei casi questo
tipo di regolazione è positiva
REGOLAZIONE POSITIVA
La trascrizione parte solo dopo
il legame di un attivatore
AraC* interagisce con la regione I
(iniziatore) e attiva l’espressione
se è presente, l’arabinosio
si lega a AraC e l’attiva
La struttura dei promotori regolati positivamente
è spesso poco aderente al consensus
araC
O
Il legame dell’attivatore ne permette il
riconoscimento da parte della RP
I
araB
araA
araD
Le basi molecolari della regolazione
positiva sono poco chiare
Il sito di legame dell’attivatore può
essere vicino al promotore
ABS
P
P
ABS
Ma anche molto lontano
in questi casi si suppone
che si formi un’ansa
P
ABS
REPRESSIONE DELLA TRASCRIZIONE
Regolazione negativa
Es. Operone “lac”
I
P O
Z
Y
A
lacZ (β-galattosidasi) scinde il lattosio
lacY (permeasi) ingresso del
lattosio nella cellula batterica
lacA (transacetilasi) funzione
non pienamente compresa
Glucosio + galattosio
lacI (repressore) è a monte del promotore pLac
ed è trascritto in modo costitutivo
Regolazione negativa
Operone “lac”
I
P
O
Z
Y
A
??
Il repressore si lega a una
sequenza di DNA (OPERATORE)
Di conseguenza la RNA polimerasi non
può legarsi alla sequenza da trascrivere
La trascrizione non può avere luogo
una molecola che, con la sua presenza, causa la produzione
dell’enzima deputato a metabolizzarla si definisce INDUTTORE
L’induttore di “Lac” è il lattosio; molecole affini, capaci di agire da induttori ma
non metabolizzate, si dicono induttori gratuiti ( es.IPTG)
L’induttore di “Lac” è un metabolita
del lattosio (allolattosio)
Il repressore ha due siti di legame: quando l’induttore si lega a uno di questi,
modifica l’altro impedendo l’interazione con l’operatore
Quando il repressore non può legarsi
la trascrizione avviene
LIVELLO BASALE
Y
Z
allolattosio
lattosio
Molti geni regolati negativamente si esprimono
a basso livello nella cellula batterica
Grazie all’espressione basale qualche molecola di LacY e LacZ
è presente quando il lattosio si rende disponibile
La loro azione permette di ottenere
l’induttore necessario per la piena espressione
Molecole che agiscono da induttori ma non sono metabolizzate
dai geni indotti, sono chiamate induttori gratuiti
Nel caso del promotore Plac l’induttore è
Non è metabolizzato
la sua concentrazione può
essere tenuta sotto controllo
ESPRESSIONE COSTITUTIVA
(anche in assenza dell’induttore)
Mutazioni nell’operatore possono impedire il legame del
repressore, causando una espressione COSTITUTIVA
I
P
O
Z
Y
A
Anche Mutazioni del repressore, nel sito di legame all’operatore (Ic)
causano una espressione costitutiva
I
P
O
Z
Y
A
SUPER REPRESSIONE
Se una Mutazione del repressore (Is), modifica il sito di
legame all’induttore ne consegue una super repressione
I
P
O
Z
Y
A
Il legame del repressore non può essere
impedito e la trascrizione non avviene
CO-REPRESSORE
Operone trp
In altri casi, la molecola
effettrice è un “co-repressore”
R
P
O
e
d
c
b
a
Da solo, il repressore è inattivo
diventa capace di impedire la trascrizione
solo quando si lega al triptofano
P
O
e
d
c
b
a
REGOLAZIONE PER SMALL RNA (sRNA)
sRNA complementari alla
regione del promotore
Possono legarvisi impedendo la
trascrizione
P
sRNA
??
O si possono legare al mRNA
impedendone la TRADUZIONE
sRNA
Un meccanismo di questo tipo regola la
produzione di alcune proteine ribosomali
Gli sRNA possono anche legarsi agli mRNA
scatenandone la degradazione precoce
sRNA
ATTENUAZIONE DELLA
TRASCRIZIONE
(operone triptofano)
L’operone Trp è regolato negativamente da
TrpR, il co-repressore è il triptofano
+
Trp
La repressione riduce l’espressione della via
biosintetica di circa 70 volte
Ma il triptofano innesca anche un altro meccanismo,
che contribuisce a inibire ulteriormente la biosintesi:
ATTENUAZIONE DELLA TRASCRIZIONE
Shhh....!
Trp
All’inizio dell’operon c’è una sequenza che
codifica un piccolo peptide (leader-14 AA)
trpR
P
O
trpE
Leader
trpD
trpC
trpB
trpA
162 nt
Nella sequenza leader (1) si trovano
due codoni “Trp” consecutivi
Nel mRNA sono presenti altre
sequenze nucleotidiche (“2”-”3”-”4”)
complementari, in grado di formare
strutture ansa-stelo
codoni trp
1
2
3
4
mRNA
(peptide leader)
Stop-codon
attenuatore
La regione “1” si appaia con la “2”
la regione “3” si appaia con la “4”
2
1
3
4
la “2” si appaia con la “3”
Ma nel caso in cui la regione
“1” non sia disponibile,
!
?
1
2
3
4
Lasciando la “4” non
appaiata
Si possono dunque verificare due
diverse conformazioni
1
2
3
L’attenuatore si forma
(ansa stelo seguita da UUUUU)
la trascrizione termina
4
UUUUUUU
2
1
L’attenuatore NON si forma
(ansa-stelo NON seguita da UUUUUU)
la trascrizione prosegue
3
4
La disposizione degli appaiamenti tra le regioni dipende dalla
posizione del ribosoma.. che dipende dalla disponibilità di Trp
Se è disponibile Trp, il ribosoma sintetizza
il peptide leader, fino al codone di stop
peptide leader
AUG
1
UGA
2
3
4
UUUUUU
il ribosoma occupa il segmento “1” ma sporge sul
segmento “2” che non si può appaiare a “3”
“3” e” 4” formano l’attenuatore e la
trascrizione termina
mRNA
Shhh..!
3
4
UUUUUU
Se Trp manca, il ribosoma si ferma in
corrispondenza dei due successivi codoni “trp”
peptide leader
AUG
UGA 2
1
3
4
mRNA
il segmento “2” è libero di appaiarsi con “3”
“3” e” 4” non formano l’attenuatore e la
trascrizione prosegue
2
3
4
I determinanti di resistenza all’Eritromicina (geni erm)
Sono stati scoperti in Enterococcus
Il gene usato sui vettori per Bacillus è
ermC, che conferisce il fenotipo MLSR
Il meccanismo con cui i geni erm conferiscono la
resistenza a questo gruppo di antibiotici è detto
ATTENUAZIONE TRADUZIONALE
L’attenuazione traduzionale permette di ottenere un mRNA
molto stabile (emivita ~ 40’) a seguito dello stallo del ribosoma
Nel messaggero sono presenti
due sequenze SD
SD2
SD2
Peptide
SD1 leader
SD1 è normalmente
accessibile per la traduzione
In assenza di induzione quindi il solo
prodotto è il piccolo peptide
ermC
Ma SD2 è sequestrata dal
ripiegamento del messaggero, dovuto
all’appaiamento di basi complementari
Quando l’eritromicina si lega al
ribosoma (il suo bersaglio)
Ne provoca lo stallo prima che la
traduzione del peptide sia terminata
Lo stallo del ribosoma provoca
un cambiamento della
conformazione del
messaggero, che espone SD2
SD2
ermC
SD2
Peptide
EmSD1 leader
ErmC
La traduzione di ermC può essere portata
a compimento da un altro ribosoma non
legato dall’eritromicina o già metilato
CONTROLLO GLOBALE
insieme di più geni o operoni sotto il
controllo della stessa proteina regolatrice
Regulone:
i geni appartengono allo stesso pathway
Modulone:
i geni appartengono a pathway diversi
Molti geni e operoni che sono sotto un controllo globale possono essere
contemporanemente regolati, positivamente o negativamente con altre strategie
REGULONE DEL MALTOSIO
L’attivatore MalT è inattivo
La forma attivata di MalT
interagisce con 4 operoni,
attivandone l’epressione
Quando è presente, il maltosio si
lega a MalT e lo attiva
REPRESSIONE DA CATABOLITA un controllo globale su diversi
operoni catabolici (es. Lac, ara..)
Se è disponibile glucosio come fonte di energia, la sintesi
degli enzimi catabolici per altre fonti viene inibita
La cellula può così scegliere tra le diverse fonti di energia e/o
carbonio disponibili, la più facile da utilizzare
La molecola effettrice nella
repressione da catabolita è cAMP
cAMP si lega alla proteina CAP, che si
attiva e si comporta da regolatore
AMP ciclico
cAMP
O
HO-P=O
CAP
CH2
H
Adenina
O
H
H
O
OH
OH
Ognuno degli operoni controllati da
CAP è controllato anche da una
proteina regolatrice specifica
ATP
Adenilato-ciclasi
glucosio
cAMP
Adenilato-ciclasi
Se è disponibile glucosio non si
produce cAMP e CAP non si attiva
I geni regolati non possono
esprimersi
Se NON è disponibile glucosio si
produce cAMP e CAP si attiva
I geni sono liberi di
esprimersi
SECONDO LA REGOLAZIONE
SPECIFICA
L’operone Lac (regolato negativamente in modo specifico)
È regolato positivamente da CAP
Se il glucosio è ASSENTE gli operoni Ara e Lac
sono liberi di essere trascritti
cAMP
I
CAP
P
SE è presente lattosio
Z
O
A
Y
Glucosio + lattosio
espressione minima
OPPURE SE è presente
arabinosio
cAMP
CAP
araC
O
I
araB
araA
araD
RISPOSTA STRINGENTE
risposta globale con cui i batteri affrontano
situazioni di emergenza
la quantità di ribosomi e di
tRNA si riduce
sintesi di nuove proteine
Duplicazione del DNA
Attivazione/repressione
di operoni biosintetici o
catabolici
Il segnale è la presenza di tRNA scarichi
durante la traduzione
AA
RISPOSTA STRINGENTE
REL-A
TRADUZIONE NORMALE
REL-A
GTP
ATP
ppGpp
l’interazione di questi tRNA con il ribosoma
induce la produzione di un nucleotide inusuale
(ppGpp - allarmone) che agisce da regolatore
FATTORI SIGMA
ALTERNATIVI
Ogni fattore sigma riconosce una classe di promotori con
specifiche sequenze consensus -35 e -10
E. coli
.
Geni trascritti
Segnale
σ70
maggior parte dei geni
σ 32
geni heat-shock
temperatura elevata
σ 55
geni regolati dall’azoto
carenza di ammonio
σ 38
geni da stress ossidativi
agente ossidante
SPORULAZIONE
La formazione dell’endospora (8-10h) è controllata da diversi meccanismi tra
cui la sintesi di fattori sigma alternativi, particolarmente importanti
Pro-σE
σE
σA + σH
Fase vegetativa
σF
Sigma precoci
al segnale di scarsità di nutrienti nell’ambiente, si avviano i
processi che portano alla sintesi di fattori sigma alternativi
Pro-σK
σK
σG
Sigma tardivi
L’inizio della sporulazione è controllato da Spo0A- P che riceve il segnale
dai sensori KinA,B attraverso la fosforilazione di Spo0F,B
e induce o reprime ~ 500 geni
KinA,B
P
Spo0F
P
Spo0B
P
Spo0A
σG
σF
REGOLAZIONE QUORUM SENSING
La regolazione Quorum-sensing permette la
sincronizzazione dell’espressione in cellule diverse
eccomi
eccomi
ECCOCI !!!!!!
I batteri avvertono la presenza e la densità di altri batteri, grazie a una molecola
segnale che loro stessi producono (AUTO-INDUTTORE)
una volta raggiunta una soglia critica, la popolazione batterica
assume comportamenti uniformi
I entrambi i gruppi i batteri producono
continuamente gli autoinduttori
Ehi!
Ehi!
Ehi!
Ehi!
Ehi!
Ehi!
Nei batteri didermi il messaggio è portato da Nacyl-omoserina-lattoni (N-AHL)
HBHL
N-(3-hydroxybutanoyl)-HL
HHL
OHHL
N-(3-oxooctanoyl)-HL
BHL
N-hexanoyl- HL
N-butanoyl- HL
differiscono leggermente da specie a specie
per la catena laterale acilica
L’autoinduttore passa liberamente
attraverso gli involucri esterni,
LuxI
In condizioni di non
attivazione l’induttore è
prodotto a livelli basali
LuxR
A bassi livelli non si lega
con l’attivatore LuxR
se la concentrazione del segnale è
elevata, l’induttore attiva il gene luxR
RISPOSTA
LuxI
si lega all’attivatore trascrizionale LuxR,
che ne è il prodotto
Il complesso induce LuxI, che
produce nuove molecole segnale,
e mantiene la risposta finché
persiste lo stimolo.
LuxR
E attiva la risposta
fenotipica
Nei monodermi la molecola segnale è
un oligopeptide, processato nella
cellula e rilasciato nell’ambiente
A
B
C
precursore
S
REG
A
B
C
È percepito da chinasi sensore che
trasferiscono il segnale all’effettore
corrispondente
precursore
S
REG
RISPOSTA !
Gli streptomiceti usano γ-butirrolattoni con una struttura simile a
quella degli N-AHL ma non è mai
stato dimostrato alcun tipo di
comunicazione tra i due gruppi
LUX-S
Oltre a LuxR/I è stato scoperto un altro
sistema in Vibrio harvey
la sintasi LuxS non omologo di LuxI
E l’autoinduttore è il peptide AI2
MONODERMI
Il sistema LuxS è presente in
DIDERMI
AI2
LUX-S è probabilmente dedicato a
comunicazioni interspecifiche e
presumibilmente più antico di LuxR/I
Più sistemi nella stessa cellula
Gerarchici (Pseudomonas)
alternativi (Bacillus)
sinergici (Vibrio)
Si inducono in sequenza
amplificando progressivamente
l’effetto fenotipico
Si escludono a vicenda, inducendo
comportamenti differenti
Inducono la medesima risposta
fenotipica, determinandola
simultaneamente
Es. in Vibrio harvey, (3 sistemi)
La risposta dipende dal bilancio tra le attività chinasiche o
fosfatasiche dei diversi recettori
AI-1
LuxR..
LuxM
LuxP-Q
sRNA x 5
P
AI-2
CAI-1
LuxS
LuxP-Q
CqsA
CqsS
P
P
LuxO
+P
La concentrazione dell’attivatore chiave
LuxR, dipende da quella di 5 piccoli RNA
regolatori, la cui sintesi è determinata dallo
stato di fosforilazione di LuxO
concentrazione di auto induttori bassa
i recettori funzionano come chinasi
fosforilano LuxO
LuxO-P
sintesi dei 5 sRNA
LuxR non si esprime
NO risposta QS
LuxM
Se gli autoinduttori sono abbondanti, i
recettori si comportano da fosfatasi
AI-1
LuxP-Q
P
sottraggono fosfati a LuxO
LuxS
LuxO
P
LuxP-Q
AI-2
i piccoli RNA NON sono sintetizzati
P
LuxR si esprime RISPOSTA QS
sRNA x 5
LuxR
LuxR!!
CqsA
CqsS
CAI-1
La degradazione degli AHL provoca
l’uscita dalla risposta di QS
In ambienti con pH
intorno a 6, tuttavia
Gli AHL si degradano spontaneamente
perdendo la catena laterale
Gli AHL sono stabili
In queste condizioni uscire dalla risposta
può essere un problema
Sono stati recentemente scoperti enzimi che degradano gli AHL
(AHL-lattonasi)
Potrebbero avere una notevole importanza nell’equilibrio tra
specie e nell’ecologia microbica
AHL-lattonasi
Molte specie possiedono più
sistemi QS nella stessa cellula
Gerarchici (Pseudomonas, Yersinia)
Si inducono in sequenza
amplificando progressivamente
l’effetto fenotipico
alternativi (Bacillus)
Si escludono a vicenda, inducendo
comportamenti differenti
sinergici (Vibrio)
Inducono entrambi la medesima
risposta fenotipica
Meccanismi di evoluzione batterica
Mutazione
trasferimento genico laterale (LGT)
e ricombinazione genica
Nel cromosoma batterico
possono verificarsi
MUTAZIONI
La sequenza delle basi nel
genoma di un organismo
cambia in modo ereditario
I cambiamenti genetici provocati dalle
mutazioni sono di entità limitata
Il fenotipo corrispondente alla
sequenza normale è “selvatico” (wild)
sostituzione
microinserzione
wild
mutante
microdelezione
Nessuna
mutazione
Sostituzione silente
Le mutazioni provocano la
comparsa di “mutanti”
RETROMUTAZIONE
ripristino del fenotipo wild
Revertante dello
stesso sito
seconda mutazione
nello stesso sito
Reversione di
“frameshift”
Reversione di
“nonsense”
ripristino della sequenza
nucleotidica “wild”
REVERSIONE VERA
SOPPRESSIONE
ripristino del fenotipo wild
mutazione in un
ALTRO sito
La seconda mutazione COMPENSA
l’effetto della prima
Altro sito stesso gene:
ripristino di “frameshift”
Altro gene
Ripristino della funzione
del gene mutato
Altro prodotto che sostituisce
quello del gene mutato
MUTAZIONI
sostituzione
UNA BASE
(puntiformi)
microinserzione
microdelezione
MOLTE PAIA DI BASI
DELEZIONI: provocano la perdita di centinaia
o migliaia di paia di basi
non revertono (a meno di ricombinazioni)
INSERZIONI: aggiungono nuove basi inattivando il gene in cui accadono
sono spesso dovute a sequenze di DNA specifiche, lunghe 700-1400 bp,
(sequenze di inserzione). Le mutazioni da inserzione possono revertere.
Sostituzione di una base
AG
GA
TC
CT
5’...TAC...
...ATG...5’
TRANSIZIONE
AG
TC
TC
AG
TRASVERSIONE
Replicazione normale
TAC
ATG
UAC (Tyr)
mutazione
AAC
TTG
AAC (Asp)
mutazione
TAG
ATC
UAG (Stop) Proteina tronca
Mutazione nonsenso
mutazione
TAT
ATA
UAU (Tyr)
Proteina difettosa
Mutazione missenso
Proteina normale
Mutazione silente
LE MUTAZIONI
POSSONO ESSERE
OCCASIONALI
ERRORI DI LETTURA
INDOTTE DA AGENTI
CHIMICI O FISICI
ORIGINATE DA
SISTEMI DI
RIPARAZIONE
Che le mutazioni abbiano un’origine spontanea lo dimostra il
TEST DI FLUTTUAZIONE
TERRENO
CON
ANTIBIOTICO
TERRENO
SENZA
ANTIBIOTICO
REPLICA-PLATING
AGENTI CHIMICI E FISICI CHE
POSSONO INDURRE MUTAZIONI
MUTAGENI CHIMICI-1
ANALOGHI DELLE BASI
O
O
CH3
H N
Br
H N
Es. 5-BrU: a differenza della Timina,
ha un legame stabile con la Guanina
O
O
H
H
Timina
H 2N
N
N H
5-BromoUracile
N
N
Adenina
N H
N
H 2N
C
N
G
G
N
5-BrU
2-Aminopurina
5-BrU
A
A
T
A:T G:C
MUTAGENI CHIMICI-2
trasformano le basi alterandone
le proprietà di appaiamento
AGENTI ALCHILANTI
Inducono mutazioni con frequenza
molto più alta degli analoghi delle basi
Nitrosoguanidina, Acido nitroso,
Idrossilamina; etilmetanosulfonato
A
Es acido nitroso deamina:
U
citosina
uracile
adenina
ipoxantina
C
IPX
MUTAGENI CHIMICI-3
AGENTI INTERCALANTI
Arancio di acridina, proflavina;
bromuro di etidio
si inseriscono tra due basi
del DNA, separandole
Causano lo slittamento della DNA
polimerasi, provocando microinserzioni e
microdelezioni durante la replicazione
Molti composti chimici possono
essere mutageni (cancerogeni)
IL TEST DI AMES
+
Salmonella
His-
controllo
+ Composto
da analizzare
Pochi
retromutanti
Frequenza invariata
Il composto NON è
mutageno
Frequenza aumentata:
il composto E’
MUTAGENO
Terreno privo
di istidina
Terreno privo
di istidina
Terreno privo
di istidina
MUTAGENI FISICI-1
RADIAZIONI NON
IONIZZANTI (UV)
I nucleotidi assorbono i raggi UV
con picco a a 269 nm
T
T
C
C
UV260 provoca la formazione di
DIMERI di PIRIMIDINE
Tra due C o T adiacenti si
formano legami covalenti
i dimeri aumentano la probabilità che la DNA
polimerasi inserisca un nucleotide errato
MUTAGENI FISICI-2
Radiazioni ionizzanti
Radiazioni a onda corta
molto penetranti
RAGGI
gamma
ionizzazione di H2O e
altre sostanze
X
cosmici
radicali liberi es OH-
azione mutagena
indiretta
ELEVATA FREQUENZA DI MUTAZIONI
MALFUNZIONAMENTO
DELLA CELLULA
ECCESSIVA DERIVA
DELLA POPOLAZIONE
LA FREQUENZA DELLE MUTAZIONI OCCASIONALI E’
LIMITATA DALL’AZIONE DELLA CORREZIONE DI BOZZE E
DEL SISTEMA MMR (CNFR DUPLICAZIONE DEL DNA)
ALCUNI DANNI PROVOCATI DAGLI AGENTI
MUTAGENI RENDONO IMPOSSIBILE LA
DUPLICAZIONE DEL DNA
SISTEMI DI RIPARAZIONE
Riparazione DNA
diretta
Riparazione della base
modificata
Es. Metilguanina transferasi
CH3
O
HN
H 2N
N
N
Trasferisce su di sé
il gruppo metile
N
H
Guanina
Separazione dei
dimeri di pirimidine
Assorbe radiazioni
nel visibile
(fotoliasi)
TT
Rompe i legami covalenti e
ricostruisce la struttura corretta
RISPOSTA SOS
DANNO AGLI ACIDI NUCLEICI
(UV o ROS)
Blocco della forca replicativa
nel punto del danno
ssDNA
Attività di co-proteasi
su LexA
Espressione coordinata di
diversi pathway (>30 geni)
Avvio di autoproteolisi
in LexA
Blocco della repressione
Rec-A
Rec-A*
LexA reprime molti
geni tra cui
parziale
RecA
recA
parziale
Error-free
uvrA
LexA
lexA
Error-prone
umuCD
IN CONDIZIONI NORMALI recA e lexA sono trascritti e
tradotti a bassa concentrazione
Le cassette SOS a monte di geni diversi hanno affinità differenti per il repressore
Non indotta
parzialmente indotta
pienamente indotta
recA
recA
recA
lexA
lexA
lexA
uvrA
uvrA
uvrA
sulA
sulA
sulA
umuCD
umuCD
umuCD
dinD
dinD
dinD
Riparazione DNA
Indiretta (escissione e ricostruzione)
Il “vuoto” è chiamato sito “AP”
La base mutata è estratta e
sostituita con quella giusta
Es: DNA-glicosilasi individua
l’uracile derivato dalla deaminazione
della citosina e lo rimuove
dC
U
La DNA polimerasi stabilizza
la base nell’elica
AP-endonucleasi e
deossiribofosfodiesterasi introducono
la base corretta nel sito AP
SINTESI TRANS-LESIONE
Le DNA-mutasi inseriscono un nucleotide qualsiasi
in corrispondenza di una lesione
x
E proseguono la sintesi
Quando il DNA è riparato
non ci sono più ssDNA
RecA si disattiva
La proteolisi di LexA
cessa
I sistemi di
riparazione tornano
ad essere repressi
I diversi geni hanno affinità differenti
per il repressore
diversa affinità dei
promotori per LexA
Induzione in quantità e in TEMPI diversi
I sistemi error free (es. uvrA) sono
indotti precocemente
Pieno successo: riparazione e
cessazione del segnale
Tasso di mutazione contenuto
Insufficienti a contenere il danno
Il segnale prosegue, sono indotte le
polimerasi error prone
(Pol. V e IV: DNA-MUTASI)
La frequenza di mutazione aumenta maggiore variabilità maggiori
possibilità di sopravvivenza di fronte a uno stress esteso
Mentre la riparazione procede, la risposta
SOS blocca la divisione cellulare
SulA, normalmente
represso da LexA
Si esprime
Si lega a FtsZ e impedisce la
formazione dell’anello
F
F
F
F
SulA
A riparazione completa, la
repressione si ripristina
La proteasi Lon degrada SulA
liberando FtsZ e la divisione
ricomincia