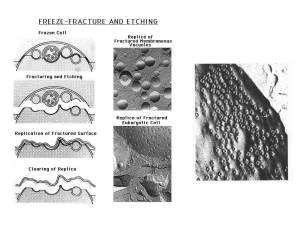
Duplicazione dell’informazione genetica
mediante replicazione del DNA
In questo processo le due catene di una doppia elica di DNA
sono separate e ciascuna serve da stampo per la sintesi
della nuova catena compementare
Dal DNA alla proteina
L’informazione genetica è letta per
mezzo di un processo a due passi
Nel primo passo, la trascrizione, segmenti
di DNA sono utilizzati per dirigere la
sintesi di molecole di RNA
Nelle traduzione quindi le molecole di
RNA sono utilizzate nella sintesi di
proteine
Trascrizione
Procarioti
DNA
mRNA Proteina
Regioni di DNA trascritte
ma non utilizzate nella traduzione
Regioni non-codificanti
Alcune Mutazioni nelle regioni non-codificanti
possono impedire al gene di esprimersi
DNA
Trascrizione
mRNA
Traduzione
Proteina
Eucarioti
Geni interrotti Non-colinearità Trascrizione
Esoni
Introni
Regioni codificanti
Regioni non-codificanti
Qual è la funzione degli Introni?
DNA
Trascrizione
RNA-precursore
Maturazione o
splicing
mRNA (maturo)
Traduzione
Proteina
Operone Lac E-coli: il livello di glucosio e lattosio contrallano l’inizio della
trascrizione agendo sul CAP (catabolite activator protein) e sul repressore
(schema semplificato).
LacZ: codifica per l’enzinma bgalattosoossidasi che separa il
lattoso in galattoso e glucosio
L’aumento di lattosio
accrece la
concentrazione di
allolattosio che si lega
la repressore e lo
rimuove dal DNA
Figure 7-39 Molecular Biology of the Cell (© Garland Science 2008)
Figure 7-37 Molecular Biology of the Cell (© Garland Science 2008)
Promoter (gene eucariote)
Figure 7-44 Molecular Biology of the Cell (© Garland Science 2008)
trascrizione
Repressilator: Negative feedback loop
M.B. Elowitz & S. Leibler Nature 403: 335-338(2000)
Il modo di funzionare del ciclo dipende da vari fattori:
• la velocità di trascrizione e la sua dipendenza dalla concentrazione di
repressore
• la velocità con cui le proteine vengono distrutte
• la velocità con cui viene distrutto l’mRNA.
Sono possibili due tipi di soluzioni:
• il sistema converge verso uno stato stazionario stabile
• lo stato stazionario diventa instabile e insorgono oscillazioni mantenute
dal ciclo limite
La cinetica del sistema è determinata da 6 equazioni differenziali
accoppiate del primo ordine:
dmi
mi
0
n
dt
(1 pi )
dpi
b ( pi mi )
dt
•Modelli matematici per i biosistemi - Benfatto - Triolo
Figure 7-72b Molecular Biology of the Cell (© Garland Science 2008)
3 tipi di RNA: t-RNA, r-RNA, m-RNA
Traduzione: t-RNA
Le molecole di RNA possono assumere
conferiscono loro particolari funzioni.
particolari
strutture
che
Singola catena → alta flessibilità → ripiegamento su stessa → legami
deboli con se stessa → complementarietà locale → forma sequenzadipendente → forma = riconoscimento specifico → catalizzatore di reazioni
chimiche → ruolo cetrale nei primi passi evolutivi
ulteriori legami H
che si formano nel
ripiegamento 3D
dihydrouridine
pseudouridine
IUPAC nucleotid code Base
A
Adenine
C
Cytosine
G
Guanine
T (or U)
Thymine (or
Uracil)
R
A or G
Y
C or T
S
G or C
W
A or T
K
G or T
M
A or C
B
C or G or T
D
A or G or T
H
A or C or T
V
A or C or G
N
any base
. or -
gap
Gli appaiamenti tra basi costanti sono 20, per un totale di 52 legami H
12 CG = 36 legami H; 8 UA = 16 legami H
Ci sono poi circa 40 ulteriori legami H
RNA Transfer
Molecola di tRNA
specifica per l’amino acido
triptofano.
Il tRNA ha attaccato da una parte
il triptofano e dall’altra
l’anticodone ACC complementare al
codone UGG sull’ mRNA
C
C A
basi conservate appaiamenti
struttura terziaria conservata
elevato impaccamento
la maggior parte delle basi
inaccessibili al solvente
fanno eccezione
• il terminale CCA accettore
• l’anticodone (mRNA)
Caratteristiche comuni a tutti i tRNA
1.
Singole catene di 7393 basi
2. Elevata percentuale di basi “insolite” (7 15)
Es: Metilazioni aumentata idrofobicità
3. Terminale 5’ fosforilato ( in genere pG)
4. Tripletta terminale 3’ CCA (legame con l’AA)
5. ~ metà dei nucleotidi appaiati
6. Ansa dell’anticodone composta di 7 basi con la seguente regolarità:
Pirimidina—Pirimidina-—x—y—z—-Purina--Base variabile
(C o U)
(C o U) anticodone (A o G)
Guanosina
Uridina
Uridina
Adenosina
Traduzione: ribosomi
l’mRNA esce dal nucleo
coefficiente di sedimentazione,
vt: velocità di sedimentazione, a: accelerazione applicata (es. gravità)
1S = 1 Svedberg = 10-13 sec
Sono mostrati solo tre dei molti fattori di
inizio richiesti: elF2, elF4G, elF4E
L’interazione tra la
coda poli-A e elF4G
serve ad accertarsi
che entrambe le
estremità dell’RNA
siano intatte
AUG=Met
Figure 6-72 Molecular Biology of the Cell (© Garland Science 2008)
E’ anche richiesta la coda di poli-A legata alla
relativa proteina
Figure 6-66 (part 1 of 4) Molecular Biology of the Cell (© Garland Science 2008)
Figure 6-66 (part 2 of 4) Molecular Biology of the Cell (© Garland Science 2008)
Figure 6-66 (part 3 of 4) Molecular Biology of the Cell (© Garland Science 2008)
Figure 6-66 (part 4 of 4) Molecular Biology of the Cell (© Garland Science 2008)
Figure 6-56 Molecular Biology of the Cell (© Garland Science 2008)
C
C A
Adenina, A
Figure 6-57 Molecular Biology of the Cell (© Garland Science 2008)
Figure 6-58 Molecular Biology of the Cell (© Garland Science 2008)
EF: elongation factor
EF-G: catalizza lo spostamento del ribosoma sull’mRNA
EF-Tu: media l’ingresso del t-RNA carico nel sito libero del ribosoma (è utilizzato
anche come controllo)
Figure 6-67 Molecular Biology of the Cell (© Garland Science 2008)
elongation
factor
Figure 6-67 (part 2 of 7) Molecular Biology of the Cell (© Garland Science 2008)
Figure 6-67 (part 4 of 7) Molecular Biology of the Cell (© Garland Science 2008)
Figure 6-74 (part 1 of 3) Molecular Biology of the Cell (© Garland Science 2008)
Figure 6-74 (part 2 of 3) Molecular Biology of the Cell (© Garland Science 2008)
Figure 6-74 (part 3 of 3) Molecular Biology of the Cell (© Garland Science 2008)
AA + AMP = AA adenilato
L’energia da idrolisi di un ATP
viene utilizzata per promuovere il
legame AA-tRNA
legame AA-tRNA e distacco di AMP
Traduzione 1
Traduzione 2