14/04/2015
A.A. 2014-15
Dipartimento di Scienze della Vita
CdS Biologia Molecolare e Cellulare (LM-6)
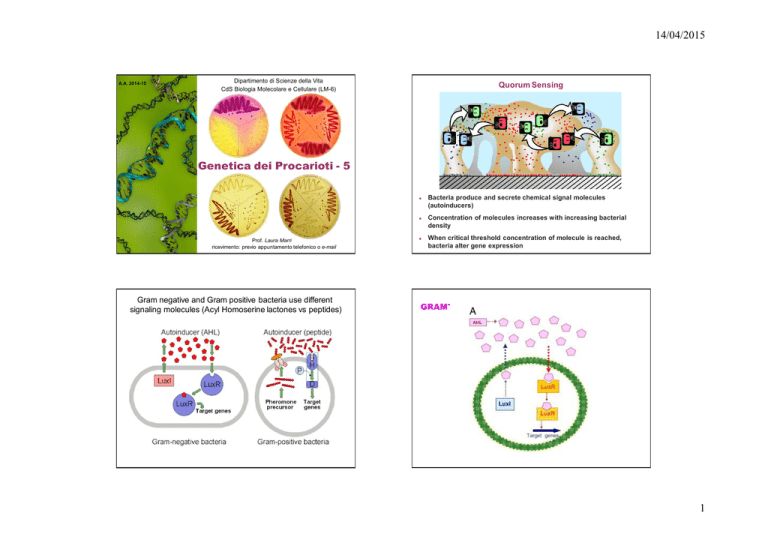
Quorum Sensing
Genetica dei Procarioti - 5
Prof. Laura Marri
ricevimento: previo appuntamento telefonico o e-mail
Gram negative and Gram positive bacteria use different
signaling molecules (Acyl Homoserine lactones vs peptides)
Bacteria produce and secrete chemical signal molecules
(autoinducers)
Concentration of molecules increases with increasing bacterial
density
When critical threshold concentration of molecule is reached,
bacteria alter gene expression
GRAM-
1
14/04/2015
GRAM+
Serratia liquefacens wild type
Ceppo mutante AHL
2
14/04/2015
AHL
Acyl Homoserine Lactone
autoinduttori
(b) Competence and sporulating stimulating factor (CSF)
autoinducer of Bacillus subtilis. (c) AI-2 autoinducer. (d) The Nbutyryl-homoserine lactone of Pseudomonas aeruginosa.
3
14/04/2015
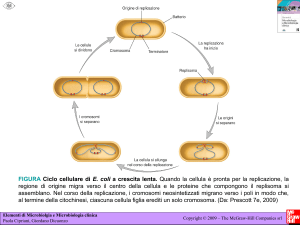
Una coltura di E. coli in terreno complesso (LB) entra
in fase di morte dopo tre giorni di incubazione.
In questa fase, il 90-99% della popolazione muore
ossidazione di proteine e DNA, programma simile
all’apoptosi eucariotica che determina la morte delle
cellule al raggiungimento di una densità elevata
EDF (extracellular death factor) attiva l’operone mazEF responsabile del modulo
tossina-antitossina che determina la morte delle cellule al raggiungimento di una
densità elevata
mazF encodes a stable toxin, MazF: a sequencespecific endoribonuclease that preferentially cleaves
single-stranded mRNAs at ACA sequences
mazE encodes a labile antitoxin, MazE, that
counteracts the action of MazF
Any stressful condition that prevents the expression
of mazEF will lead to a reduction of MazE levels in
the cell, permitting the MazF toxin to act
4
14/04/2015
Chromobacterium violaceum
Biosensori batterici per la rivelazione di AHLs
proteina della
famiglia LuxR
promotore della
famiglia LuxI
A mutant of C. violaceum ATCC
31532, designed strain CV026,
does not produce C6-HSL but does
produce violacein in response to
the presence of exogenous C6-HSL
(McClean et al., 1997)
gene reporter
the proper production of AHL molecules has been blocked by mutation of the AHL
synthase but the gene encoding the production of the purple pigment violacein
remains AHL-responsive.
In the presence of specific AHLs with acyl
chain lengths shorter than C10, this strain will
therefore produce purple pigment due to
violacein production
5
14/04/2015
Controllo post-traduzionale
Nei batteri Gram-positivi le proteine secrete o sono rilasciate nell’ambiente
esterno oppure sono legate covalentemente al petidoglicano da specifici enzimi
associati alla membrana e vanno a costituire le proteine di superficie
SECREZIONE PROTEINE
Proteine specifiche si trovano:
associate alla IM
nello spazio periplasmatico
OM
in alcuni casi secrete completamente dalla cellula
Le proteine legate alle membrane comprendono:
almeno 8 sistemi OM
Gram-
almeno 8 sistemi IM
Molte proteine sono secrete attraverso entrambe le membrane cellulari
(IM, OM) dei batteri GRAM-negativi
Alcune usano la via Sec per attraversare la IM ed un’altra via per la OM
Altre dipendono da altri sistemi per attraversare entrambe le membrane
Citocromi
F1/Fo ATP sintasi
Proteine di trasporto
Porine
CITOPLASMA
1
2
Sec-pathway
Il trasporto è realizzato attraverso un meccanismo dipendente da una
delivering protein
chaperone
translocation
competent state
SEQUENZA SEGNALE
Met–X – Lys/Arg – Z(10 -12x) – Gly/Ala –
Regione basica
5-6 residui
core idrofobico
forma un
canale
N-terminus proteina matura
20aa
translocation
complex
X = residuo idrofilico o idrofobico
Z= residuo prevalentemente idrofobico
PERIPLASMA
peptidase
6
14/04/2015
La proteina matura inizia il processo
di ripiegamento, se a destinazione
periplasmatica, oppure può essere
presa in consegna da un successivo
sistema di trasporto se destinata a
OM/ ambiente extracellulare
Phylogenetic tree of sequenced homologues of the SecY protein of E. coli
and Sec61 protein of Saccharomyces cerevisiae
two-step process
A+B
homologues follow the phylogenies of the
host organisms with few exceptions
7
14/04/2015
Type III – T3S
apparato che trasloca effettori in one step attraverso la
parete usando una struttura ancestralmente correlata al
flagello batterico
8
14/04/2015
nomenclatura di Yersinia
Yop: Yersinia outer protein
solo dopo il
contatto con la
cellula eucariotica
chaperonine specifiche
Shigella type III secretion system
Type IV – T4S
ancestralemente correlato all’apparato della coniugazione
che transloca DNA o substrati proteici attraverso la parete
(spesso in relazione alla patogenesi)
I batteri usano il sistema di secrezione di tipo IV (T4S) per due
motivi fondamentali:
•
scambio genico
•
rilascio di molecole effettrici (EFFETTORI) a
cellule
eucariotiche bersaglio
9
14/04/2015
(a) a model for T-pilus assembly in Agrobacterium tumefaciens
VirB4–VirB8–VirB5–VirB2
interaction sequence lead
to the formation of VirB2–
VirB5 complexes followed
by T-pilus assembly
(b)
transmission electron microscopic image showing
Secretion pathway and structure of the T4S system
T-pili and flagella on A. tumefaciens.
10
14/04/2015
secondary amine derivatives formed by condensation of an amino
acid, either with a ketoacid or a sugar
Crown gall tumors
galla del colletto
Ti è un esempio di plasmide batterico naturale
11
14/04/2015
MGEs: segmenti di DNA codificanti enzimi o altre proteine che
consentono lo spostamento di questi elementi nel genoma (mobilità
intracellulare) o tra cellule batteriche (mobilità intercellulare)
Also, virulence plasmids from Salmonella, Shigella, Yersinia, B.
anthracis, E.coli, and others.
PLASMIDI
PLASMIDI
Le forme molecolari circolari superavvolte durante le
nella maggior parte dei casi sono molecole di DNA circolari
manipolazioni sperimentali possono rilassarsi o linearizzarsi
(1 - 1000 kb) ma anche lineari o integrati nel cromosoma
in seguito a rotture a singolo o a doppio filamento.
batterico (episomi)
plasmide superavvolto
supercoiled
plasmide circolare
rilassato
nick
dal 1977
plasmide linearizzato
12
14/04/2015
In un gel di agarosio e bromuro di etidio le tre forme migrano a
velocità diverse e possono essere distinte
forma linerizzata ( tagliata con un
enzima di restrizione in un sito unico)
forma rilassata
forma lineare
forma superavvolata
Large-scale purification of plasmids
by cesium chloride density gradient centrifugation
“old school method of purifying plasmid”
since the 1950s
Plasmid replication
1. Plasmid replication requires host DNA replication machinery.
Three forms of plasmid DNA
2. Most wild plasmids carry genes needed for transfer and copy number
control.
3. All self replication plasmids have a oriV: origin of replication
4. Some plasmids carry and oriT: origin of transfer.
5. Plasmid segregation is maintained by a par locus-a partition locus
that ensures each daughter cells gets on plasmid. Not all plasmids
have such sequences.
6. There are 5 main “incompatibility” groups of plasmid replication. Not
all plasmids can live with each other.
72-96 hrs
during this time, the CsCl forms a gradient and the molecules migrate
according to their density until they float at their individual isopycnic points (the
point in the gradient that equals the buoyant density of the molecule).
7. Agents that disrupt DNA replication destabilize or cure plasmids from
cells.
13
14/04/2015
Replicazione plasmidica
Replicazione plasmidica
REPLICONI
I plasmidi si replicano
per replicazione (uni o bi-direzionale)
per circolo rotante
richiedono proteine plasmidiche e/o dell’ospite batterico
un plasmide deve contenere un’origine per la replicazione del DNA
Replicazione plasmidica
REPLICONI
circolo rotante
replicazione
I plasmidi si replicano
per replicazione (uni o bi-direzionale)
per circolo rotante
richiedono proteine plasmidiche e/o dell’ospite batterico
14
14/04/2015
Replicazione plasmidica
La capacità di replicarsi autonomamente è conferita loro dalla
Funzioni dell’origine di replicazione
presenza di una origine di replicazione, ori (oriV, “ori vector”)
Oltre ad essere essenziale per la replicazione, l’origine di
replicazione oriV controlla:
Il numero di copie
La specificità d’ospite
I gruppi di incompatibilità
host protein
P1
NUMERO DI COPIE e MODALITA' DI REPLICAZIONE
NUMERO DI COPIE e MODALITA' DI REPLICAZIONE
I plasmidi si replicano con due modalità diverse
Altri, in genere di piccole dimensioni, si replicano in maniera
Alcuni, generalmente quelli di grandi dimensioni, si replicano in
indipendente dalla replicazione batterica e si dicono sottoposti a
maniera coordinata con la replicazione del cromosoma batterico
e si dicono sottoposti a
controllo stringente
controllo rilassato
Sono presenti in molte copie - fino a 1000 - per batterio.
In genere sono presenti in una o poche copie per batterio.
15
14/04/2015
NUMERO DI COPIE e MODALITA' DI REPLICAZIONE
Plasmidi
ori
H
I
H
G
N. di copie
PUC e derivati
ColE1
500-700
pACYC e derivati
p15A
10-12
pSC101 e derivati
Psc10
1-5
C
O
P
Y
N
U
M
B
E
R
Sistema di ripartizione
L
O
W
C
O
P
Y
N
U
M
B
E
R
16
14/04/2015
Sistema di ripartizione
Bacterial plasmids encode partitioning
(par) loci that ensure ordered plasmid
segregation prior to cell division
ParM binds to DNA-binding proteins,
called ParR (orange proteins) around
which segments of genomic DNA are
coiled
parS-ParA-ParB
Sister plasmid segregation is achieved
through bidirectional insertional polymerization
of the ParM filaments.
Actina-omologo
17
14/04/2015
Controllo del numero di copie
I plasmidi possono controllare il numero di copie regolando l’inizio
della replicazione plasmidica
inibizione mediante RNA + proteina dell’inizio della
replicazione
Controllo del numero di copie
Rnasi H
RNA II
ori
la replicazione è innescata da un primer (RNA II), trascritto da un
promotore situato 550 bp a monte di ori.
Gli ibridi DNA:RNA formati dal filamento di DNA e dall’RNA II nascente,
La maggior parte dei vettori di clonaggio
derivano dal plasmide ColE1
costituiscono un substrato per la Rnasi H che taglia l’ibrido e fornisce
l’OH al 3’ per la replicazione del DNA.
18
14/04/2015
La maturazione di RNA II è controllata da RNA I, trascritto sul
filamento opposto della stessa regione di DNA e, quindi,
complementare a RNA II .
RNA I-RNA II compete con l’appaiamento tra RNA II e il filamento
stampo, riducendo la frequenza di inizio della replicazione.
Il prodotto del gene rop stabilizza il complesso RNA I-RNA II ,
riducendo ulteriormente la frequenza di inizio.
RNA II
ori
RNA I
rop
Replicazione del plasmide ColE1
RNA II is required for
priming DNA synthesis
at the origin. Positive
regulation.
-555
RNA II
P-II
P-I
RNA polimerasi
-20
origin
RNA I
RNA I (108 b)
P-I & P-II: promotori
RNA I è complementare alla regione terminale 5’ di RNA II
19
14/04/2015
SPETTRO D’OSPITE
Funzioni dell’origine di replicazione
Alcuni plasmidi sono in grado di replicare in un numero limitato di
specie batteriche e si dicono a specificità d'ospite limitata : narrow
Oltre ad essere essenziale per la replicazione, l’origine di
host range
replicazione oriV controlla:
Il numero di copie
Altri sono in grado di replicarsi in una vasta gamma di specie
batteriche e si dicono a largo spettro d'ospite: broad host range, BHR
Lo spettro d’ospite
I plasmidi BHR derivano questa loro proprietà dal possesso di
I gruppi di incompatibilità
funzioni per il riconoscimento dell' origine di replicazione, dipendono
meno, quindi, dall'apparato replicativo dell'ospite batterico
GRUPPI DI INCOMPATIBILITA’
Funzioni dell’origine di replicazione
Plasmidi con la stessa origine di replicazione sono incompatibili tra
loro.
Oltre ad essere essenziale per la replicazione, l’origine di
replicazione oriV controlla:
Il numero di copie
Se in uno stesso batterio entrano due plasmidi con la stessa origine
di replicazione, questa compete per componenti proteici comuni.
Come risultato nel giro di poche generazioni uno dei due plasmidi
La specificità d’ospite
verrà perso.
I gruppi di incompatibilità
Possono invece coesistere plasmidi con origine di replicazione
diverse che appartengono a diversi gruppi di incompatibilità
20
14/04/2015
INCOMPATIBILITA’ PLASMIDICA
21
14/04/2015
MARCATORI SELEZIONABILI
I plasmidi naturali a volte veicolano uno o più geni non
essenziali capaci di conferire loro un vantaggio selettivo in
alcune situazioni.
Per es. possono codificare per tossine o resistenza agli
antibiotici
Plasmidi coniugativi e non coniugativi
capacità di trasferire materiale genetico tra batteri
Plasmidi coniugativi e non coniugativi
plasmidi privi di queste funzioni geniche non sono trasmissibili
mediante coniugazione
Per essere coniugativo un plasmide deve possedere:
Una specifica regione di riconoscimento chiamata Ori T
(origine di trasferimento)
I prodotti genici, agenti in trans, specificati dal locus tra
(trasferimento)
una delle funzioni tra: sintesi del pilo coniugativo
Tuttavia . . .
22
14/04/2015
Plasmidi coniugativi e non coniugativi
Distribution of conjugative, mobilizable, and nonconjugative plasmids
according to plasmid size
se un plasmide possiede un Ori T può
essere trasferito per coniugazione
utilizzando funzioni helper che
forniscono, in trans, i prodotti genici
mancanti
Smillie C et al. Microbiol. Mol. Biol. Rev. 2010;74:434-452
Schematic view of the genetic constitution of transmissible plasmids.
Escherichia coli S17-1 λpir è in grado di mobilizzare plasmidi non coniugativi in
batteri Gram-negativi grazie all’integrazione stabile nel proprio cromosoma dei geni
codificanti per le funzioni di trasferimento derivate dal plasmide RP4 che agiscono
sulla sequenza oriT presente sui plasmidi mobilizzabili
oriT
geni tra
plasmide
coniugazione
MPF: mating pair formation
T4CP: Type IV coupling proteins
Ricevente
Smillie C et al. Microbiol. Mol. Biol. Rev. 2010;74:434-452
23