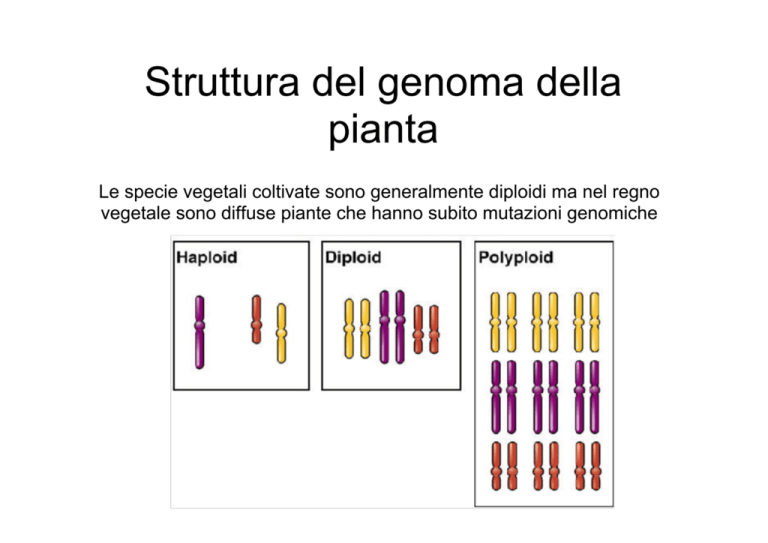
POLIPLOIDIA: moltiplicazione dei cromosomi al cui interno
troviamo
a) Autopoliploidi: piante poliploidi in cui due o più cromosomi
sono uguali tra loro
b) Allopoliploidi: piante poliploidi che derivano dall’incrocio
tra 2 specie affini (genomi simili ma non uguali)
Il caso del frumento:
Il T. urartu (diploide con genoma A) esiste esclusivamente in forma selvatica. Benché i
genomi di Triticum monococcum e Triticum urartu siano molto simili, le due specie
sono considerate distinte poiché non danno progenie fertile se interfecondate.
SNP discovery
• Confronto di sequenze disponibili in database pubblici per genotipi diversi
• Sequenziamento random di frammenti di DNA
• TARGET GENE approach, Esoni conservati: disegno di primer su sequenze conservate
in geni ortologhi di specie sinteniche (conserved primers, intron-flanking primers , or
exon-priming-intron-crossing (EPIC) primers)
Questa strategia si basa sul fatto che:
• la maggior parte degli esoni o regioni geniche sono conservate tra specie
sinteniche
• la posizione degli introni e la loro lunghezza sono conservate anche tra
specie evolutivamente distanti
• Gli introni evolvono più velocemente degli esoni quindi la probabilità di
identificare SNPs è superiore
PREREQUISITO: l’esistenza di database di EST
You et al., BMC Bioinformatics 2009
Ricerca di relazioni di sintenia tra Brachypodium, riso e frumento nella regione del QTL localizzato sul
cromosoma 7B di frumento
Chr. 6
Chr. 1
Chr. 7B
Oryza sativa
Brachypodium
distachyon
Xbarc340
Xgwm146
Xgwm344
Oryza sativa
Brachypodium
•The online tool MIPS (Comparative Genome Map Viewer) was used to search for orthologous genes within the
identified syntenic blocks.
T. aestivum
QLr14.ubo
Oryza sativa
Sviluppo di marcatori basati su EST di frumento e la sequenza
genomica di riso e Brachypodium :
Wheat
Wheat
cDNA ESTs
unigene
Rice or
Brachipodyum
Artificial wheat
PCR development
Gene sequence
Gene sequence
N Intron length prediction
N from rice/Brachipodyum
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
N
Cloning in pGemEasyVector
UBW14CTG1.1
C L
INTRON1
U
C
L
INTRON2
UBW19CTG2.4
U
C L U
INTRON3
Colony pcr
C L U
INTRON1
Subgenomic-SNP: 1 SNP / 50 bp
B gen.
A gen.
Co
Co
Co
LL
LL
LL
LL
Co
Co
LL
LL
U
U
Varietal-SNP (Colosseo vs. Lloyd): 1 SNP / 1640 bp
Colosseo
Colosseo
Colosseo
LLoyd
LLoyd
LLoyd
LLoyd
Colosseo
Colosseo
LLoyd
Lloyd
T. Urartu (AA)
T. Urartu (AA)
B gen.
C
Phylogenetic Tree
L
A gen.
Development of Codominant Markers on SNPs: ASO
M13
Pr Fw Co-specific
3’
Colosseo ATTgCATCTAtCTTAAGCATtAAGTAGGt............GTACGTACGTCTGTCAATTGCGT
5’
Lloyd
Pr Fw LL-specific
3’
ATTaCATCTAgCTAAGCATgAAGTAGGc............GTACGTACGTCTGTCAATTGCGT
3’
5’
Pr Rw B-genome-specific
0
Codominant marker
UBW14
Co LL
RIL CxL
M13
Co allele
142 bps
LL allele
123 bps
UBW18
RIL CxL
Co LL












