Seconda Università degli Studi di Napoli
DiSTABiF
Corso di Laurea in Scienze Biologiche
Insegnamento di
CHIMICA BIOLOGICA
Prof. Antimo Di Maro
Anno Accademico 2016-17
Lezione 11
“fattore trasformante”
L'esperimento di Avery dei suoi colleghi MacLeod e McCarty risale al 1943 e
rappresenta una delle esperienze fondamentali per l'avanzamento delle conoscenze nel
campo della genetica e della biologia molecolare.
separo
inietto
Gli acidi
nucleici
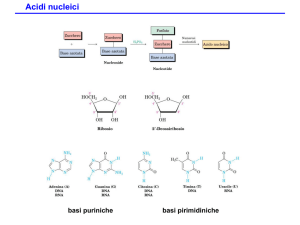
Acidi nucleici
Acido ribonucleico
• Ribosio
• Quattro basi azotate (A, U, G e C)
• il gruppo fosfato
Acido desossiribonucleico
•Nel ribosio un idrogeno sostituisce
l’ossidrile in 2’
•Quattro basi azotate (A, T, G e C)
•il gruppo fosfato
Organizzazione gerarchica
Anche per gli acidi nucleidi si parla di struttura primaria
(sequenza) struttura secondaria (doppia elica…) e struttura
terziaria (arrangiamento nello spazio)
Ribosio
Gli zuccheri presenti negli acidi nucleici sono
2-deossi-D-ribosio (DNA) e D-ribosio (RNA)
5’
1’
4’
3’
2’
5’
1’
4’
3’ 2’
Tautomerizzazione delle Basi
Nucleosidi
•
Il legame tra basi eterocicliche e ribosio forma i diversi
Nucleosidi
•
Il legame è di tipo N-β-glicosidico tra il Carbonio
anomerico C1’ dello zucchero e l’N9 delle purine o l’N1
delle pirimidine
•
Il legame può avere due orientazioni: Syn e Anti. Anti è
syn
predominante
•
Hanno numerosi gruppi polari e sono solubili in acqua.
Sono facilmente idrolizzabili da soluzione acquose di
acidi.
anti
Nucleosidi
Base
DNA
Adenina
Guanina
Citosina
Timina
Zucchero
Nucleoside
2-deossi-D-R.
2-deossi-D-R.
2-deossi-D-R
2-deossi-D-R.
2’-deossiadenosina
2’-deossiguanosina
2’-deossicitidina
2’-deossitimidina
RNA
Adenina
Guanina
Citosina
Uracile
D-R.
D-R.
D-R.
D-R.
Adenosina
Guanosina
Citidina
Uridina
Nucleotidi
•
•
•
•
I nucleotidi sono ESTERI FOSFATO dei nucleosidi.
Il sito di fosforilazione è l’ossidrile al 5’ dello zucchero
Possono essere mono- di- e tri-fosfati (Es.: TMP, TDP, TTP.)
I di- e tri-fosfati sono legati da legame anidridico, stabile e ad alta
energia
+ circa ‐30 kJoule/mol
(oppure ‐7,3 kcal/mol)
Da kcalorie a kJoule si
passa moltiplicando per
4,1868
+ circa ‐30 kJoule/mol
(oppure ‐7,3 kcal/mol)
Da kcalorie a kJoule si passa
moltiplicando per 4,1868
+ circa ‐30 kJoule/mol
(oppure ‐7,3 kcal/mol)
Da kcalorie a kJoule si passa
moltiplicando per 4,1868
Funzioni dei nucleotidi
• Unità strutturali degli acidi nucleici
• Deposito di energia delle reazioni di trasferimento di fosfato
(ATP - GTP)
• Mediatore di processi cellulari (cAMP)
• Parte di coenzimi (NADH, FAD, CoA )
• Intermedi di reazioni sintetiche (S-Adenosilmetionina)
ATP-ADP-AMP
L'adenosina è presente come fosfato in varie forme.
Il 5'-monofosfato, difosfato e trifosfato, così come il monofosfato 3',5'-ciclico, sono
intermedi chiave in numerosi processi biologici.
L'ATP contiene due legami di anidride fosforica ed ha la caratteristica di liberare
quantità notevoli di energia quando si idrolizza ad ADP e, successivamente, ad
AMP. Sono queste le reazioni che forniscono l'energia necessaria per altre
reazioni biologiche.
circa ‐30 kJoule/mol
(oppure ‐7,3 kcal/mol)
Da kcalorie a kJoule si passa
moltiplicando per 4,1868
Derivati dell’AMP
Il più comune è 3'-5'- adenosina monofosfato ciclico, cAMP, prodotto dall’enzima
adenilato ciclasi
È un secondo messaggero per la trasduzione del segnale dalla superficie cellulare
all’interno della cellula. E’ usato dalla protein-chinasi cAMP dipendente (PKA)
per fosforilare molte proteine.
La forma ciclica di GMP (cGMP), è un secondo messaggero analogo a cAMP, ma
con funzioni distinte
S-adenosilmetionina
La S-adenosil metionina è una forma attivata di metionina per la
donazione di metilici
CoA, NAD+, FAD
Coenzimi importanti contengono nucleotidi nella loro
struttura:
Coenzima A, nella cui struttura è presente l'ADP, un
agente biologico capace di trasferire i gruppi acilici,
che ha un ruolo chiave nel metabolismo dei grassi.
Il nicotinammide adenina dinucleotide (NAD) ed il
flavina adenina dinucleotide (FAD) sono coenzimi che
partecipano a numerose reazioni biologiche di ossidoriduzione.
Polinucleotidi
I polinucleotidi si formano dalla condensazione di due o più nucleotidi tra il
fosfato legato al 5’-OH del ribosio di un nucleotide ed il 3’-OH del ribosio
dell’altro con eliminazione di H2O
Ogni fosfato ha ancora un protone acido che, a pH 7, è ionizzato (carica
negativa sull'ossigeno). Se il gruppo fosfato fosse indissociato, la sostanza
sarebbe un acido; polinucleotidi biologici = acidi nucleici.
Il legame fosfodiesterico è direzionale, e la sequenza va da 5’ 3’
La sequenza delle basi definisce la molecola 5’-pGpApTpC-3’
I polinucleotidi biologici sono
l’ACIDO DEOSSIRIBONUCLEICO (DNA)
l’ACIDO RIBONUCLEICO (RNA)
Abbiamo visto i monomeri degli acidi
nucleici (PM circa 300/350 dalton) e le
differenze chimiche tra DNA e RNA a
adesso dobbiamo vedere come si
legano tra di loro per ottenere i
polideossiribonucleotidi e i
poliribonucleotidi
Legame fosfodiesterico
H
H
H
H
Legame fosfodiesterico
Definiti i monomeri, e la loro struttura, e il legame
che ne permette la polimerizzazione, come le
proteine anche gli acidi nucleici hanno:
Struttura primaria: la successione delle basi (singolo
filamento)
Struttura secondaria: a doppio filamento
Struttura terziaria: conformazione nello spazio
La struttura primaria degli acidi nucleici
Struttura primaria: la
successione
delle
basi
(singolo
filamento)
Basi rare .. bassa frequenza
La struttura secondaria del DNA
Regole di Chargaff:
Nel DNA la quantità di adenina (A) è sempre uguale alla
quantità di timina (T) e la quantità di citosina (C) è
sempre uguale alla quantità di guanina (G).
Di conseguenza il quantitativo di basi puriniche (A+G) è
sempre uguale al quantitativo di basi pirimidiniche (C+T).
A =1
T
G= 1
C
G= 1
C+5’me-Cit
Foto 51
Rosalind Franklin
1. è un’elica
2. probabilmente una doppia elica
antiparallela
3. i fosfati sono presenti all’esterno
1953.
Watson e Crick
propongono il
modello a doppia
elica del DNA
Franklin
Wilkins
89
Struttura secondaria del DNA,
modello Watson-Crick
Elica destrorsa costituita da due catene
polinucleotidiche antiparallele
Le basi sono all’interno dell’elica allineate ad
angolo retto con l’asse dell’elica (deossiribosio
e fosfato all’esterno)
Le due catene sono unite da ponti idrogeno
tra purine di una catena e pirimidine dell’altra
G-C formano 3 legami idrogeno
A-T formano 2 legami idrogeno
Le eliche sono a polarità invertita ma complementari!
3,4 Ǻ
34 Ǻ un giro completo, circa 10,5 basi
Il diametro è di 20 Ǻ
Le cariche dei fosfati sono
stabilizzate o da ioni (Mg2+)
o da interazione con proteine
basiche
La struttura secondaria degli acidi nucleici è
stabilizzata solo dai legami ad idrogeno tra le basi??
No
Vi sono anche le interazioni idrofobiche tra le coppie
di basi sovrapposte (impilate) sono la forza trainante
che stabilizza la struttura secondaria del DNA e degli
acidi nucleici con struttura secondaria
Conformazioni del DNA
Il modello della doppia elica di Watson-Crick è in realtà una
semplificazione.
Le conformazioni ad elica del DNA vengono attualmente classificate
in tre famiglie generali: forme A, B e Z.
B
A
Z
B-DNA
Forma predominante = regolare elica destrorsa di Watson e Crick,
con basi perpendicolari all’asse dell’elica
Si ottiene quando il DNA è completamente idratato (in vivo)
Diametro: circa 20 Ǻ
10 basi per giro completo dell’elica (10,5)
3,4 Ǻ = distanza tra due basi (3,4 Ǻ)
34 Ǻ = per ogni giro d’elica In soluzione
36°= angolo rotazione tra due basi
direzione di rotazione dell’elica
destrorsa
La superficie esterna mostra due solchi separati dalla catena fosfato-deoribosio: solco (scanalatura) maggiore (major groove) e solco minore (minor
groove).
Minor groove
Major groove
Solco maggiore
Solco minore
A-DNA
Ottenuto in condizioni di disidratazione non fisiologica (sembra non
esistere in vivo)
Elica destrorsa con catene antiparallele
Diametro
circa 26 Ǻ
11 coppie di basi per giro completo dell’elica
33,1° rotazione angolare tra due basi adiacenti
2,6 Ǻ = distanza tra due basi adiacenti
Z-DNA
Struttura trovata in sequenze caratterizzate da
alternanza di G e C.
Citosine metilate (isole metilate ricche in CG)
Probabile presente anche in vivo
Elica sinistrorsa e la dorsale zucchero-fosfato segue
un andamento a zig-zag.
Guanidine (purine) in posizione sin.
Major groove molto superficiale e minor molto stretto.
sin
anti
Aspetti chimico-fisici
degli acidi nucleici
Assorbanza a 260 nm
1.0
A=lc
0.5
0
220 nm
260nm
300 nm
Denaturazione al calore
Denaturazione termica del DNA
- Ipercromismo
- Ipocromismo
- Tm
Idrolisi alcalina dell’RNA
(in ambiente basico)
RNAsi, enzimi che degradano il DNA…
E. coli
Struttura terziaria
Topoisomerasi di tipo I e di tipo II ed
enzimi che ne regolano gli avvolgimenti
Topoisomerasi di tipo I tagliano una sola elica
Topoisomerasi di tipo II tagliano due eliche contemporaneamente
Girasi
Topoisomerasi di tipo II
Nucleosoma
H2A
H2B
H3
H4
(x 2, ottamero)
Che caratteristiche hanno gli istonici del nucleosoma
circa 150 deossiN
circa 60 deossiN
cariotipo di quale specie?
Oltre alle differenze chimiche che c’è
da sapere sul RNA?
RNA, classi
tRNA
L’RNA generalmente si dice che è a
singolo filamento, ma in realtà può
avere una struttura primaria,
secondaria (A=U, e G=C), e terziaria
Struttura secondaria
Legame in posizione 5 della pirimidina
Diidrouridina
Ribosio
Braccio accettore
3’OH
tRNA
Struttura
tipo DNA A
Per l’ossidrile del
Ribosio in 2’
Struttura terziaria
Base
Purinica
5’
A
C
C
Ansa D diidrouridina
Ansa T ΨC
Ansa dell’anticodone
rRNA
RNA ribosomale 16S,
struttura secondaria
mRNA
Tipo DNA A nella zona dell’ibrido
mRNA
hnRNA
RNA eterogeneo
nucleare
Splicing
Strutture palindromiche negli
acidi nucleici, sia DNA che RNA.
Sono importanti perché
rappresentano spesso zone di
interazione con fattori di
regolazione
Palindromo
Le-ta-le, Ne-ro-ne, … Radar, Anilina
Palindromo
Una sequenza palindromica è
una regione della struttura
primaria del DNA costituita
da due successioni di basi
ripetute ed invertite (in
figura).
Struttura a forcina
Sono sequenze autocomplementari lungo una
catena e pertanto sono in
grado dare appaiamenti
intracatena formando
strutture a forcina.
Struttura a croce
-Se sono coinvolte due
catene
Assodato che l’RNA si
polimerizza utilizzando come
stampo il DNA, come si replica il
DNA. In modo conservativo,
dispersivo o semiconservativo?
L'esperimento di Meselson e Stahl del 1958
Replicazione semiconservativa