Proteine: funzioni principali
1) La struttura (collagene e cheratina)
2) La catalisi (enzimi)
3) Il movimento (miosina ed actina)
4) Il trasporto (emoglobina e proteine di membrana)
5) La protezione (anticorpi)
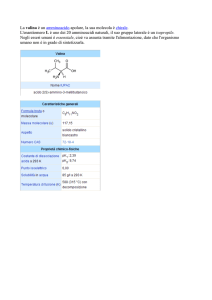
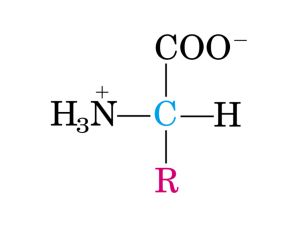
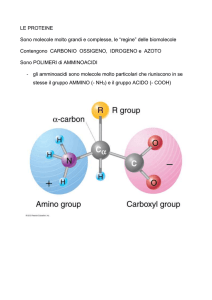
Amminoacidi
Forma non
ionizzata
Un α-amminoacido
Il carbonio α è asimmetrico (chirale)
Forma ionizzata
sale
interno
(zwitterione)
Gli α-amminoacidi sono le unità molecolari (monomeri)
che costituiscono le proteine (polimero)
Insulina umana: una piccola proteina
Gli amminoacidi proteici sono 20 ed hanno sempre chiralità L
Gli a.a. differiscono per la catena laterale R
Catene laterali non polari
Catene laterali polari
Catene laterali acide
Catene laterali basiche
Alcuni altri L-amminoacidi comuni
metaboliti
ormoni
neurotrasmettitore
Proprietà acido-basiche degli amminoacidi
Basicità del gruppo guanidinico dell’arginina
Basicità del gruppo imidazolico dell’istidina
è in grado di accettare protoni
Titolazione degli amminoacidi: curva di titolazione della glicina
OH
OH
H2NCH2COO
H3NCH2COO
H3NCH2COOH
H2O
H2O
PUNTO ISOELETTRICO
Valore di pH al quale un amminoacido, un polipeptide o una proteina
non posseggono una carica netta.
pI = 1/2(pKa α-COOH + pKa α-NH3+)
GLICINA
pI = ½(2.35 + 9.78) = 6.06
Elettroforesi
Tecnica analitica mediante la quale è
possibile separare composti in base alle
loro cariche elettriche
Carta
Campione
Soluzione
tampone
Il colorante più usato per rivelare
gli amminoacidi è la ninidrina
La reazione con un amminoacido porta ad un’adeide e ad un composto
colorato
Anione di colore porpora
Polipeptidi e proteine: il legame peptidico
Polipeptidi e proteine
Peptidi
2-10 amminoacidi (dipeptide, tripeptide, etc.)
Oligopeptidi
11-19 amminoacidi
Polipeptide
20-100 amminoacidi
Proteina
Più di 100 amminoacidi
Tripeptide Ser-Phe-Asp
legame peptidico
legame peptidico
Per convenzione una sequenza peptidica viene scritta iniziando da
sinistra con l’amminoacido N-terminale (gruppo –NH3+ libero) e
procedendo verso destra fino all’amminoacido C-terminale (gruppo
–COO- libero).
Struttura primaria di polipeptidi e proteine
Rappresenta la sequenza degli amminoacidi lungo la catena
polipeptidica (ovvero l’ordine con cui sono legati)
a.a N-terminale
a.a. C-terminale
Come si fa a stabilire la struttura primaria?
Analisi qualitativa-quantitativa degli a.a
Si procede in due tappe
Analisi della sequenza degli a.a.
Analisi quantitativa degli amminoacidi
Indica di che tipo sono gli amminoacidi ed in che quantità sono
presenti nel polipeptide:
Idrolisi acida
Miscela
di
amminoacidi:
si analizza per
cromatografia a
scambio ionico
HCl 6M, 110°, 24-72 ore
Colonna cromatografica
Miscela di amminoacidi
Analizzatore di
amminoacidi
L’analizzatore
fornisce un grafico
(cromatogramma)
Analisi di una miscela di amminoacidi
pH 3.25
treonina
Serina
Ac.
glutammico
pH 4.25
metionina
isoleucina
valina
leucina
alanina
fenilalanina
glicina
Ac.aspartico
prolina
cistina
tirosina
pH 5.28
fenilalanina
tirosina
lisina
istidina
arginina
Con questa analisi stabiliamo quali
sono gli amminoacidi e in che
quantità relative sono presenti nel
polipeptide
Analisi della sequenza di un polipeptide
Avviene in più fasi:
I
Idrolisi parziale
(specifica)
Agente enzimatico
o chimico
II
Separazione
Metodi cromatografici
A
III
sequenziamento
B
sequenziamento
C
sequenziamento
Il bromuro di cianogeno come agente chimico
A
sequenziamento
Il sequenziamento utilizza la
degradazione di Edman
Si basa sulla reazione selettiva e sul distacco del solo amminoacido Nterminale
Si isola e si
identifica
Si ripete la
degradazione
di Edman
Ripetendo degradazione di Edman sul peptide residuo (più
corto di un a.a.) si identifica l’a.a. successivo. Si procede così
per tutti gli a.a. (max 50-100)
A
B
C
Cosi facendo, si possono identificare le sequenze dei tre
frammenti peptidici A,B,C. Tuttavia non sappiamo in che
ordine sono legati tra loro.
A
B
C
?
A
C
B
?
B
A
C
?
Per conoscere l’ordine con cui i frammenti A, B, C, sono legati
tra loro si ripete il processo di sequenziamento sull’intero
peptide utilizzando un enzima o agente chimico diverso
I
Idrolisi parziale
(specifica)
Diverso agente
enzimatico o chimico
Separazione
Metodi cromatografici
II
A’
III
sequenziamento
B’
sequenziamento
Dal paragone dei risultati del primo sequenziamento con il
secondo è possibile risalire ai “punti di giunzione” tra i
frammenti peptidici A, B, C, (ed anche A’ e B’) e quindi
individuare l’esatta sequenza dell’intero peptide.
A
B
C
A’
B’
si deduce
sequenza completa
FORME TRIDIMENSIONALI DI POLI PEPTIDI E PROTEINE
Geometria del legame peptidico
Il legame ammidico ha un carattere parziale di doppio legame:
la rotazione intorno ad esso è impedita.
La rotazione impedita intorno al legame peptidico crea due
possibili configurazioni (cis e trans) – (Diastereoisomeria)
cis
Questo aspetto influenza molto
secondaria e terziaria delle proteine.
trans
le
strutture
Struttura secondaria di una proteina
Descrive le sistemazioni ordinate (conformazioni) assunte dagli
amminoacidi in particolari regioni di un polipeptide o di una
proteina
Legame
idrogeno
I legami idrogeno tra gruppi ammidici stabilizzano la conformazione
di un polipeptide o di una proteina.
Pauling propose due tipi di strutture secondarie:
Struttura ad
ad
Struttura
elica
αα elica
Conformazione
Conformazione
foglietto
ββ aa foglietto
ripiegato
ripiegato
Struttura ad
ad αα elica
elica
Struttura
Legami idrogeno
intracatena
N-H
O=C
Asse
dell’elica
-R
1) L’elica è destrorsa.
2) Vi sono 3.6 a.a. per
ogni giro d’elica.
3) I legami peptidici
sono planari e trans.
4) N-H punta verso il
basso, mentre C=O
verso l’alto.
5) Il legame H si
stabilisce tra due a.a.
che distano 4 unità.
6) I gruppi -R puntano
verso l’esterno.
Conformazione ββ aa foglietto
foglietto ripiegato
ripiegato
Conformazione
C-terminale
N-terminale
N-terminale
C-terminale
C-terminale
Ci sono legami idrogeno intercatena
-R
N-terminale
1) Le catene sono adiacenti e corrono in direzioni opposte (antipar.)
2) I legami peptidici sono planari e trans.
3) I gruppi N-H e C=O puntano ciascuno verso l’altro e giacciono
sullo stesso piano.
4) In ciascuna catena i gruppi –R si alternano verso l’alto e verso il
basso
Struttura terziaria di una proteina
Si riferisce al tipo di avvolgimento complessivo e alla sistemazione
nello spazio di tutti gli atomi di una singola catena polipeptidica.
Struttura terziaria della mioglobina. Si individuano alcuni tratti con
struttura α.
Oltre ai legami idrogeno la struttura terziaria di una proteina è
“tenuta insieme” anche da legami disolfuro:
Insulina umana
Catene laterali
di cisteine
Ox
Red
Legame
disolfuro
Struttura quaternaria di una proteina
La possiedono le proteine formate da più di una catena polipeptidica.
Indica la disposizione reciproca delle catene nello spazio: