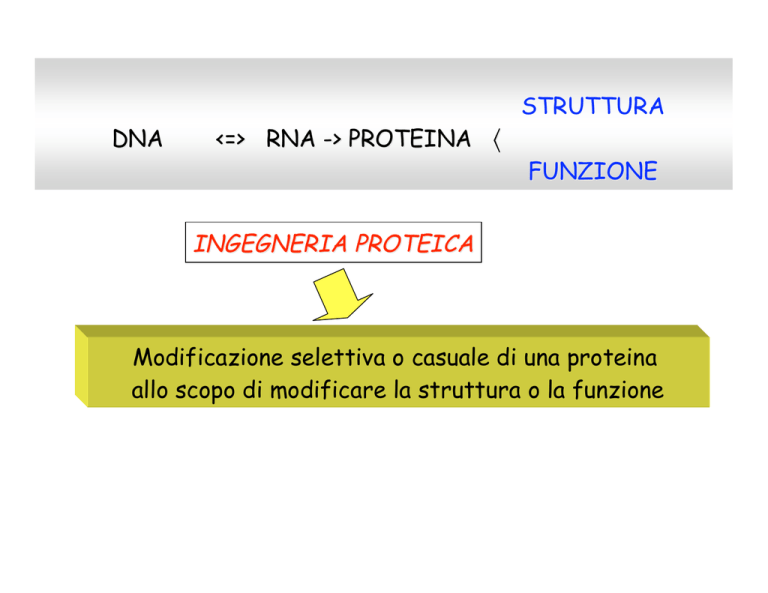
DNA
<=> RNA -> PROTEINA 〈
STRUTTURA
FUNZIONE
INGEGNERIA PROTEICA
Modificazione selettiva o casuale di una proteina
allo scopo di modificare la struttura o la funzione
PRODURRE
CARATTERIZZARE
MODULARE ATTIVITA’ O
INTRODURRE NUOVE FUNZIONI
PROTEINE NATURALI
Ingegneria proteica
PROTEINE ARTIFICIALI
PROGETTARE
APPLICAZIONI
INDUSTRIA
MOLECOLE STABILI ED ATTIVE IN
CONDIZIONI PARTICOLARI DI PH,
TEMPERATURA E CONDIZIONI DI
REAZIONE
BIOMEDICINA
MOLECOLE STABILI ED ATTIVE
IN
CONDIZIONI
FISIOLOGICHE,
IN
PRESENZA DI ENZIMI ED INIBITORI,
AD EMIVITA LUNGA E DA VEICOLARE
EFFICACEMENTE
AI
BERSAGLI
CELLULARI
IN UN ESPERIMENTO DI INGEGNERIA PROTEICA SI POSSONO MODULARE
MOLTE PROPRIETA’ DI UNA MOLECOLA
RESIDUI
IDROFOBICI
ESPOSTI
SOLUBILITA’
SITI DI LEGAME
AFFINITA’
SPECIFICITA’
TERMINALI
FUSIONI CON
PEPTIDI O PEG
Proprieta’ generali
LOOPS
RESISTENZA
PROTEOLISI
ALTRI PARAMETRI
BIOLOGICI E CLINICI
NUCLEO
STABILITA’
CONTROLLO
CONFORMAZIONALE
EPITOPI
IMMUNOGENICITA’
DESIGN RAZIONALE
EVOLUZIONE GUIDATA
L’INGEGNERIA PROTEICA RICHIEDE UN APPROCCIO MULTIDISCIPLINARE
Bioinformatica
Genomica strutturale
Genomica funzionale
Biochimica
strutturale
(NMR, X-ray, Massa)
IDENTIFICAZIONE
MOLECOLA
RACCOLTA INFORMAZIONI
Ingegneria genetica
Ingegneria proteica
Ingegneria metabolica
BIOMOLECOLE ATTIVE
DNA RICOMBINANTE ED INGEGNERIA PROTEICA
Di cosa ho bisogno per esprimere una proteina?
UN GENE
lamB
terminatore
promotore
UNA UNITA’ DI
TRASCRIZIONE
lamB
STOP
RBS
lamB
UNA UNITA’ DI
TRADUZIONE
Alcuni punti importanti da rispettare:
Qualità del prodotto proteico e Resa del sistema di produzione.
Rapporto qualità/costo vantaggioso.
Assenza di agenti infettivi per l’uomo.
FASI SPERIMENTALI PRINCIPALI
1 : Identificazione, isolamento
bersaglio
2:
e clonaggio gene codificante per proteina
espressione in sistema eterologo
3: purificazione e caratterizzazione proteina wild type
4: progettazione, produzione e caratterizzazione versioni mutate
FASE 1: isolamento gene codificante
-INFORMAZIONI SULLA PROTEINA DI INTERESSE
-INFORMAZIONI SU PROTEINE OMOLOGHE
(STRUTTURA/FUNZIONE)
-ANTICORPO SPECIFICO
⇓
- ISOLAMENTO (SINTESI) SEQUENZA CODIFICANTE
⇓
- ANALISI DI COLLEZIONI DI MOLECOLE (GENI O PRODOTTI)
- REAZIONE A CATENA DI POLIMERASI TERMOSTABILI (PCR)
- SINTESI CHIMICA EX NOVO
⇓
-DETERMINAZIONE SEQUENZA NUCLEOTIDICA E CLONAGGIO
Alcuni casi difficili
proteina e/o mRNA poco abbondante
proteina oligomerica
cofattori e/o gruppi prostetici
L’espressione eterologa di una proteina segue le regole della cellula ospite
eucarioti
procarioti
Controllo trascrizionale
Controllo traduzionale
Controllo post-traduzionale
Stabilità mRNA
Modificazioni
Velocità di traduzione
Ribosoma
mRNA
DNA
RNA polimerasi
Proteina
post-traduzionali
Compartimenti
(periplasma procarioti)
-Scelta coppia sistema di espressione/ospite dipende dalle caratteristiche
molecolari della proteina di interesse
( importanza di qualità e resa).
-Procarioti o eucarioti? Differenze (vantaggi e svantaggi):
- Compartimentalizzazione
- Uso del codice genetico (tRNA)
- Stabilità cloni ricombinanti
- Modificazioni post-traduzionali
- Resa in biomassa
- Purificazione
- Crescite terreni modificati (isotopi o metalli pesanti)
- Costo
- fattibilità su piccola scala e larga scala
(a) Sistemi di espressione procariotici (Rese: µg-mg/litro coltura)
-Plasmidi e virus batterici
- Ospite più utilizzato: Escherichia coli
Parametri principali nei procarioti
DNA
RNA
1. Caratteristiche vettore
2. Efficienza trascrizionale
2. Efficienza traduzionale
3. Stabilità della proteina
Proteina
VETTORE DI ESPRESSIONE PROCARIOTICO
NUMERO DI COPIE PER CELLULA:
-Derivati di pBR322: 15-20 copie per cellula
- Derivati di pUC: 150-200 copie per cellula
Il numero di copie influenza:
- positivamente la stabilità del plasmide, cioè la sua presenza nelle
generazioni successive.
-negativamente la velocità di crescita del ceppo ospite: crescono meglio le
cellule con meno copie.
I PLASMIDI AD ALTO NUMERO DI COPIE NON SEMBRANO
CONFERIRE UN VANTAGGIO PER LA PRODUZIONE DELLA PROTEINA
SELEZIONE: Ampicillina, tetraciclina, kanamicina
Sconsigliata ampicillina perché:
perdita di resistenza
potenzialmente allergenica
Fase stazionaria
Morte cellulare
Densità
cellulare
Fase logaritmica
Fase lag
Tempo di crescita
PROMOTORE: COSTITUTIVO O INDUCIBILE??
PROMOTORE (costitutivo o inducibile):
-costo dell’induttore
-efficacia dell’induzione e della repressione in condizioni di noninduzione per bilanciare produzione di biomassa e resa in proteina
-forza del promotore e resa in proteina (solubile)
Promotore
lac
tac
trc
T7
trp
araBAD
PL(l)
Induzione
IPTG
IPTG
IPTG
IPTG
Trp
mancanza
L-Arabinose
termica
PR(l)
lac(TS)
PSPA
termica
termica
Costitutivo
altro
lattosio
“
“
“
Analogo acido
indolacetico
Espr.basale/induzione
di hsp
“
Il sistema di espressione pET
E.coli lambda lisogeno (λDE3)
SEGNALI RELATIVI ALLA TRADUZIONE:
-SEQUENZA SHINE-DALGARNO OTTIMALE
5’-UAAGGAGG-3’
- POSIZIONE: 4-8 NUCLEOTIDI DA AUG (rimozione Nterminale?)
- SECONDO CODONE: di solito ricco in A nei geni molto
espressi. AAA(lys) è frequente (13%).
5’
RICCO
TRADUZIONE
IN
GC
DIMINUISCE
EFFICIENZA
CODON USAGE
USO DI CODONI RARI PUO’ PORTARE A PROBLEMI:
STABILITA’ mRNA DIMINUITA
TERMINAZIONE PREMATURA DI TRASCRIZIONE/TRADUZIONE
FRAMESHIFT, DELEZIONI, INCORPORAZIONI ERRATE
INIBIZIONE DELLA CRESCITA CELLULARE
CODONI USATI CON
FREQUENZA BASSA
NELLE VARIE SPECIE
(tRNA meno abbondanti)
FREQUENZA DI
USO DEI CODONI
RARI DI E.coli
IN ALTRE SPECIE
ESPRIMERE IN E.COLI UN GENE RICCO IN CODONI RARI
ABBASSA LA EFFICIENZA DI TRADUZIONE
QUALI SOLUZIONI?
-Geni mutati o costruiti (sintetici)
BL21 (DE3)
CodonPlus-RIL
BL21 (DE3)
CodonPlus-RP
Rosetta or
Rosetta (DE3)
AGG/AGA
(arginine),
AUA
(isoleucine)
and CUA
(leucine)
AGG/AGA
(arginine)
and CCC
(proline)
AGG/AGA
(arginine),
CGG
(arginine),
AUA
(isoleucine)
CUA
(leucine),
CCC
(proline),
and GGA
(glycine)
Stratagene
Stratagene
Novagen
CEPPI DI E.COLI
CHE ESPRIMONO
tRNA RARI
CEPPI CHE ESPRIMONO
MENO PROTEASI
MANCATA O BASSA ESPRESSIONE
LA PROTEINA VIENE ESPRESSA IN CONDIZIONI NON DI INDUZIONE
*
ESPRESSIONE COSTITUIVA DI UN REPRESSORE
lac repressor(the lacI or lacIq) PER PROMOTORE LAC
*
USARE UN PROMOTORE STRINGENTE, e.g. the arabinose promoter
(PBAD).
*
USARE PLASMIDE A BASSO NUMERO DI COPIE
*
VETTORI PET: USARE CEPPI CON T7 Lisozima from a compatible
pLysS or pLysE plasmid (Novagen). IL LISOZIMA SI LEGA ALLA POLIMERASI
ED INATTIVA L’ENZIMA IN ASSENZA DI INDUTTORE
•AGGIUNGERE 1% glucosio PER REPRIMERE IL PROMOTORE lac INDOTTO DA
LATTOSIO PRESENTE NEI MEZZI DI COLTURA MASSIMI ( LB, 2xYT).
LA STABILITA’ DEL PLASMIDE E’ BASSA
*AUMENTARE LA CONCENTRAZIONE DELL’ANTIBIOTICO DI SELEZIONE
LA PROTEINA E’ TOSSICA
*ESPRESSIONE NEL PERIPLASMA O IN CORPI INCLUSI
LA PROTEINA RICOMBINANTE DEVE ESSERE SOLUBILE
SPESSO SI HA LA FORMAZIONE
DI CORPI INCLUSI
(aggregati proteici insolubili in acqua)
STRATEGIE PER AUMENTARE LA SOLUBILITA’
RIDURRE LA VELOCITA’ DI TRADUZIONE
*
*
*
*
ABBASSARE LA TEMPERATURA DI CRESCITA
USARE UN PROMOTORE PIU’ DEBOLE
USARE UN PLASMIDE A BASSO NUMERO DI COPIE
ABBASSARE LA CONCENTRAZIONE DI INDUTTORE
CAMBIARE LE CONDIZIONI DI CRESCITA
*
AGGIUNGERE COFATTORI NECESSARI PER FOLDING
*
CONTROLLARE LE VARIAZIONI DI PH
*
AGGIUNGERE GLUCOSIO 1% PER REPRIMERE LA ESPRESSIONE
DEL PROMOTORE LAC INDOTTO DA LATTOSIO PRESENTE NEI
TERRENI MASSIMI ( LB, 2xYT).
*
AGGIUNGERE ETANOLO, TIOLI A BASSO PESO MOLECOLARE,
NaCl.
CO- ESPRIMERE CON IL GENE DI INTERESSE CON:
ALTRE SUBUNITA’ DELL’OLIGOMERO
CHAPERONES MOLECOLARI
*
*
*
GroES-GroEL
DnaK-DnaJ-GrpE
ClpB
FOLDASI
•
PEPTIDIL -PROLIL CIS/TRANS ISOMERASI (PPI's)
•
DISULFURO-OSSIDOREDUTTASI (DsbA)
•
DISULFURO ISOMERASI (DsbC)
•
PROTEINA DISOLFURO ISOMERASI (PDI) – EUCARIOTICACATALIZZA SIA OSSIDAZIONE DELLE CISTEINE CHE DSI: HA ANCHE
ATTIVITA’ CHAPERONE.
ESPRIMERE LA PROTEINA NEL PERIPLASMA MEDIANTE AGGIUNTA DI UNA
SEQUENZA SEGNALE (pelB/ompT)
•
L’AMBIENTE E’ PIU’ OSSIDANTE CHE NEL CITOPLASMA
•
SONO PRESENTI DsbA E DsbC
•
ATTIVITA’ PROTEOLITICA RIDOTTA
•
PERMETTE ACCUMULO DI PROTEINE TOSSICHE NEL
CITOPLASMA
•
N-TERMINALE VERO
Svantaggi: livelli di espressioni minori
USARE CELLULE OSPITI “SPECIALI” CON CITOPLASMA OSSIDANTE
CEPPI COMMERCIALI
*
*
AD494, mutato nella tioredossina reduttasi (trxB).
Origami, mutato nella tioredossina reduttasi (trxB) e glutatione reduttasi (gor).
PRODURRE UNA PROTEINA DI FUSIONE CON PROTEINA SOLUBILE
MALTOSE-BINDING PROTEIN
UBIQUITIN
DsbA
TIOREDOSSINA
IgG-BINDING DOMAIN
SVANTAGGI: RECUPERO DELLA PROTEINA MEDIANTE PROTEOLISI
ESPRIMERE FRAMMENTI SOLUBILI DELLA PROTEINA
IN ALCUNI CASI LA ESPRESSIONE NEI CORPI INCLUSI PUO’ ESSERE UN
VANTAGGIO:
•
•
•
LA PROTEINA RICOMBINANTE E’ FINO AL 50%
DELLE PROTEINE TOTALI
E’ PROTETTA DALLA DEGRADAZIONE
NON PUO’ ESSERE TOSSICA PER LA CELLULA
LA PROTEINA NATIVA DEVE ESSERE RECUPERATA MEDIANTE
DENATURAZIONE IN VITRO E REFOLDING
*ISOLAMENTO DEI CORPI INCLUSI. SHOCK OSMOTICO E
CENTRIFUGAZIONE
*DENATURAZIONE IN PRESENZA DI AGENTI DENATURANTI COME
GUANIDINA O UREA O CONDIZIONI RIDUCENTI
(e.g. DTT).
*REFOLDING DELLA PROTEINA MEDIANTE LENTA RIMOZIONE DEL
DENATURANTE CON DIALISI, DILUIZIONE O CROMATOGRAFIA
(IN PRESENZA DI AGENTI RIDUCENTI )












