© 88-08-07275-4
Capitolo 5 • Il paradigma dell’espressione differenziale dei geni 105
Scoperte recenti hanno dimostrato che numerosi fattori di trascrizione agiscono reclutando le istone acetiltransferasi. Quando le proteine istoniche sono acetilate,
il nucleosoma si disgrega, consentendo ad altri fattori di
trascrizione e alla RNA polimerasi di accedere al sito.
Alcuni TAF, come TAFII250, sono essi stessi istone acetiltransferasi, cosicché possono attivare la trascrizione sia
destabilizzando il nucleosoma, sia permettendo alla proteina TBP di legarsi al DNA e di istituire così un sito di
legame per la RNA polimerasi (Fig. 5.10; Mizzen et al.
1996).
Tipo selvatico
mitf–/–
Mitf
Tirosinasi
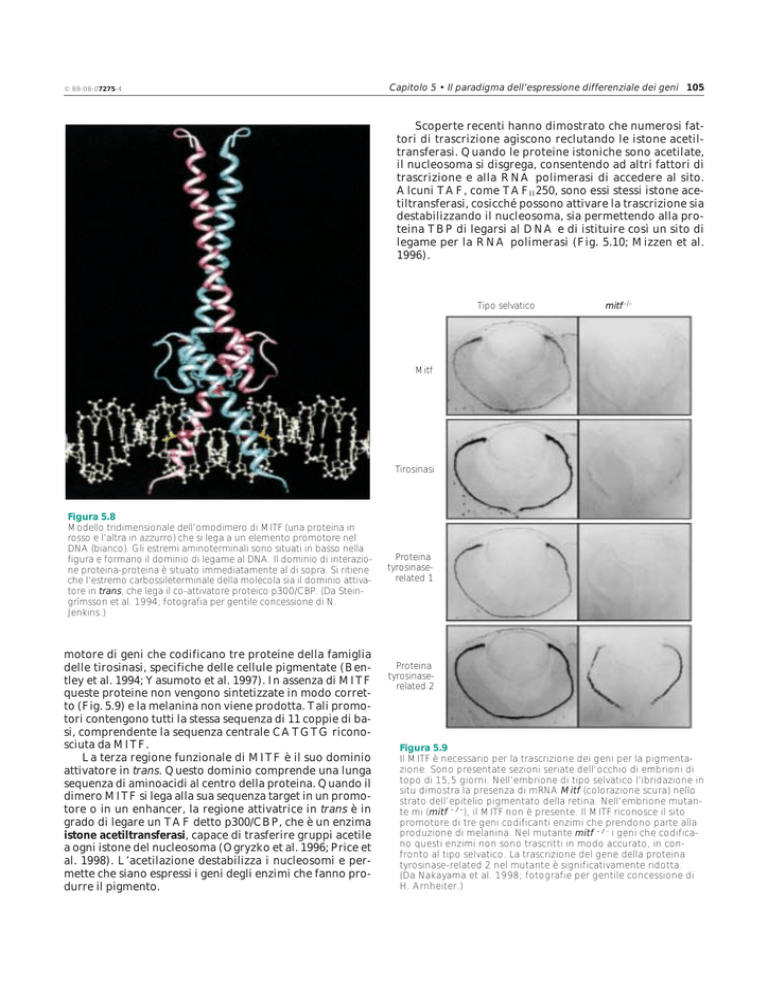
Figura 5.8
Modello tridimensionale dell’omodimero di MITF (una proteina in
rosso e l’altra in azzurro) che si lega a un elemento promotore nel
DNA (bianco). Gli estremi aminoterminali sono situati in basso nella
figura e formano il dominio di legame al DNA. Il dominio di interazione proteina-proteina è situato immediatamente al di sopra. Si ritiene
che l’estremo carbossileterminale della molecola sia il dominio attivatore in trans, che lega il co-attivatore proteico p300/CBP. (Da Steingrímsson et al. 1994; fotografia per gentile concessione di N.
Jenkins.)
motore di geni che codificano tre proteine della famiglia
delle tirosinasi, specifiche delle cellule pigmentate (Bentley et al. 1994; Yasumoto et al. 1997). In assenza di MITF
queste proteine non vengono sintetizzate in modo corretto (Fig. 5.9) e la melanina non viene prodotta. Tali promotori contengono tutti la stessa sequenza di 11 coppie di basi, comprendente la sequenza centrale CATGTG riconosciuta da MITF.
La terza regione funzionale di MITF è il suo dominio
attivatore in trans. Questo dominio comprende una lunga
sequenza di aminoacidi al centro della proteina. Quando il
dimero MITF si lega alla sua sequenza target in un promotore o in un enhancer, la regione attivatrice in trans è in
grado di legare un TAF detto p300/CBP, che è un enzima
istone acetiltransferasi, capace di trasferire gruppi acetile
a ogni istone del nucleosoma (Ogryzko et al. 1996; Price et
al. 1998). L’acetilazione destabilizza i nucleosomi e permette che siano espressi i geni degli enzimi che fanno produrre il pigmento.
Proteina
tyrosinaserelated 1
Proteina
tyrosinaserelated 2
Figura 5.9
Il MITF è necessario per la trascrizione dei geni per la pigmentazione. Sono presentate sezioni seriate dell’occhio di embrioni di
topo di 15,5 giorni. Nell’embrione di tipo selvatico l’ibridazione in
situ dimostra la presenza di mRNA Mitf (colorazione scura) nello
strato dell’epitelio pigmentato della retina. Nell’embrione mutante mi (mitf – / –), il MITF non è presente. Il MITF riconosce il sito
promotore di tre geni codificanti enzimi che prendono parte alla
produzione di melanina. Nel mutante mitf – / – i geni che codificano questi enzimi non sono trascritti in modo accurato, in confronto al tipo selvatico. La trascrizione del gene della proteina
tyrosinase-related 2 nel mutante è significativamente ridotta.
(Da Nakayama et al. 1998; fotografie per gentile concessione di
H. Arnheiter.)
Capitolo 5 • Il paradigma dell’espressione differenziale dei geni 111
© 88-08-07275-4
tre gli stessi promotori sono altamente metilati nelle cellule che non producono globine. Inoltre il modello di metilazione si modifica nel corso dello sviluppo (Fig. 5.18B). Le
cellule che producono emoglobina nell’embrione umano
hanno promotori non metilati nei geni che codificano le
globine ε («catene globiniche embrionali») dell’emoglobina embrionale. Questi promotori vengono metilati nel feto, quando vengono attivati i geni delle globine γ, specifiche dell’emoglobina fetale (al posto delle catene embrionali) (van der Ploeg e Flavell 1980; Groudine e Weintraub
1981; Mavilio et al. 1983). Analogamente, quando alle globine fetali subentrano quelle adulte (β), i promotori dei geni delle globine γ vengono metilati.
La correlazione tra citosine metilate e repressione della trascrizione è stata confermata sperimentalmente. Inserendo nelle cellule transgeni con differenti modelli di metilazione, Busslinger e collaboratori (1983) dimostrarono
che la metilazione del promotore o dell’enhancer di un gene è strettamente correlata con la repressione della trascrizione del gene. Nello sviluppo dei vertebrati l’assenza
di metilazione del DNA mostra una buona correlazione
con l’espressione tessuto-specifica di molti geni.
(A)
L1
promotore
NRSE
lacZ
(B)
L1
promotore
Assenza di NRSE
lacZ
Figura 5.17
Silencer. Analisi del modello di colorazione per la β-galattosidasi in
embrioni di topo di 11,5 giorni. (A) Embrione contenente un transgene costituito dal promotore L1, una parte del gene L1 e un gene lacZ
fuso nel secondo esone (che contiene la regione NRSE). (B) Embrione
contenente un transgene simile, ma privo della sequenza NRSE. Le regioni scure rivelano la presenza di β-galattosidasi (il prodotto del gene
lacZ). (Da Kallunki et al. 1997.)
principale di regolazione della trascrizione nei vertebrati;
invece nella Drosophila, nei nematodi e probabilmente in
molti invertebrati non si ha metilazione del DNA.
Nei vertebrati, la presenza di citosine metilate nel promotore di un gene è in correlazione con la repressione della trascrizione di quel gene. Nel differenziamento degli eritrociti dell’uomo e del pollo il DNA dei promotori dei geni delle globine è quasi completamente non metilato, menFigura 5.18
Metilazione dei geni della globina in cellule embrionali del sangue
nell’uomo. (A) Struttura della 5-metilcitosina. (B) L’attività dei geni
della globina β umana mostra una correlazione inversa con la metilazione dei loro promotori. (Da Mavilio et al. 1983.)
(A)
N
NH2
O
N
DNA
Citosina
Demetilazione
CH3
N
Metilazione
Come vengono metilati certi geni e in che modo la metilazione impedisce l’espressione dei geni? Una risposta possibile è che vi sia un’interazione reciproca tra gli istoni della
cromatina e il DNA che si avvolge attorno ad essi. Pare che
esista una competizione per la modificazione del residuo di
lisina in posizione nove dell’istone H3. Se questo sito viene
acetilato, l’istone viene destabilizzato e può causare la dispersione del nucleosoma. Se il sito non viene acetilato,
può essere metilato. La metilazione di questo istone aumenta la stabilità del nucleosoma, impedendone la dissociazione o lo spostamento. Così la modificazione della coda dell’istone H3 potrebbe agire come un «commutatore»
tra lo stato attivato (nucleosomi disgregati) e quello inattivato (nucleosomi stabili) di un gene. L’istone metilato potrebbe essere capace di reclutare nelle sue vicinanze gli enzimi che operano la metilazione del DNA (Rea et al. 2000;
Tamaru e Selker 2001). Una volta metilato, il DNA può
stabilizzare ancora di più i nucleosomi.
Una proteina detta MeCP2 si lega in modo selettivo alle
regioni metilate del DNA e si lega anche alle istone deaceti-
Promotore
non metilato
(B)
NH2
Modificazioni della cromatina
5′
Promotore
metilato
Gene della globina ε
Gene della globina γ
3′
DNA
O
N
6 settimane
Attivo
H
DNA
5-metilcitosina
Globina ε
Inattivo
5′
12 settimane
3′
Inattivo
Attivo
Globina γ












