BIOMOLECOLE – STRUTTURA E FUNZIONE
Lo studio delle relazioni tra struttura e funzione nelle biomolecole è uno degli aspetti più importanti per la
comprensione del funzionamento dei processi biologici
“La parola proteina, che ti propongo... vorrei
derivasse da proteios, perche sembra essere la
sostanza primitiva o principale della nutrizione
animale che le piante preparano per gli
erbivori,e che quest’ultimi forniscono ai
carnivori”.
J. J. Berzelius, lettera a G. J. Mulder, 1838
Tutti gli organismi viventi sono formati da cellule e, nonostante varino in termini di
forma, dimensione e aspetto, gran parte della materia che le costituisce è di natura
macromolecolare (biopolimeri) composta principalmente da carbonio, idrogeno, azoto
e ossigeno. Questi quattro elementi insieme costituiscono circa il 99% della massa
della maggioranza delle cellule. La chimica dei viventi è organizzata intorno all’atomo
di carbonio che da solo costituisce più della metà del peso secco della cellula. Il
carbonio, grazie alla sua capacità di formare quattro legami covalenti ed essere in
grado, per la sua dimensione, di formare doppi e tripli legami, ha la possibilità di
realizzare con altri atomi di carbonio catene più o meno lunghe, lineari o ramificate,
oppure chiuse ad anello. L’esistenza praticamente illimitata di molecole diverse
rendono questo elemento fondamentale per l’esistenza della vita. Le complesse
funzioni biologiche richieste di un organismo necessitano di strutture molecolari
altrettanto complesse diverse per dimensione, forma e composizione, che nessun altro
elemento chimico all’infuori del carbonio è in grado di realizzare. È quindi possibile
l’esistenza di macromolecole con le caratteristiche necessarie per sostenere, ad
esempio, l’azione meccanica della cellulosa nelle piante o la possibilità di
immagazzinare l’informazione genetica.
La conoscenza e la comprensione della chimica di queste biomolecole è quindi
necessaria per capire il funzionamento non solo della cellula ma di tutto l’organismo
vivente. Dal punto di vista strutturale e funzionale e della composizione chimica, i
biopolimeri vengono generalmente divisi in tre classi principali: proteine, polisaccaridi
e acidi nucleici. Queste macromolecole si formano a partire da precursori
relativamente semplici con peso molecolare inferiore o uguale a 500 e contengono un
gran numero di monomeri e presentando un peso molecolare elevato che va da 5000
fino a più di 1 milione per le proteine, di milioni per i polisaccaridi e fino ad alcuni
miliardi per gli acidi nucleici.
Un’altra classe di biomolecole di notevole importanza biologica è quella dei lipidi, i
quali svolgono diversi ruoli all’interno delle cellule: servono come molecole fornitrici e
di riserva di energia e come componenti strutturali delle membrane cellulari. Le
singole molecole lipidiche sono relativamente piccole (con pesi molecolari che vanno
da 750 a 1500), ma possono associarsi attraverso legami non covalenti per formare
strutture molto estese come le membrane cellulari.
1
Figura 1 - Struttura di un
generico
amminoacido.
Il
gruppo R legato al carbonio è
diverso in ogni amminoacido
Quello che tutte le macromolecole biologiche e gli
assemblaggi lipidici hanno in comune è che essi adottano
strutture tridimensionali definite che sono la chiave delle
loro
funzioni.
La
struttura
tridimensionale
della
macromolecola
può essere descritta in termini di
configurazione e conformazione. La prima corrisponde alla
disposizione spaziale permanente degli atomi ed è
determinata dalla presenza di doppi legami (isomeria
geometrica) o di centri chirali attorno ai quali sono disposti i
gruppi sostituenti (stereoisomeria). Due isomeri non
possono convertirsi tra
loro senza la rottura di
uno o più legami.
La
conformazione
rappresenta invece la
disposizione
spaziale
che
i
vari
gruppi
possono assumere per
rotazione
interna
ai
legami semplici senza la
rottura
di
alcun
1
legame .
Tutte le
macromolecole
biologiche
sono
caratterizzate
da
sequenze specifiche di
monomeri
(struttura
primaria)
che
determinano
la
loro
struttura e funzione.
La
relazione
tra
struttura e funzione
particolarmente
evidente nelle proteine
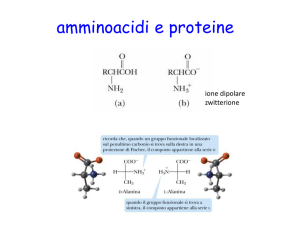
che sono molecole formate da amminoacidi, composti
bifunzionali contenenti un gruppo amminico (-NH2) e
un gruppo carbossilico (-COOH) legati allo stesso
atomo di carbonio e che differiscono l’uno dall’altro
per la catena laterale (fig.1). Sequenze diverse di
amminoacidi generano strutture che possono avere
compiti molto diverse come attività catalitica (enzimi),
protezione
immunologica
(anticorpi),
trasporto,
funzione
strutturale,
coordinamento
dell’attività
Figura 2 - Struttura dei 20 amminoacidi presenti nelle proteine. Nello
schema ad anello, che non rappresenta nessuna proteina reale, gli
amminoacidi sono posti l’uno accanto all’altro in base alle proprietà
chimiche delle catene laterali – (R. F. Doolittle - Le proteine - Le Scienze n° 208
(1985))
Figura
3
–
Strutture
dei
due
enantiomeri di un amminoacido con il
carbonio
asimmetrico.
Le
due
strutture
sono
l'una
l'immagine
speculare dell'altra
1
Vedi lezione Treccani: Dai gruppi funzionali ai polimeri
2
neuronale, controllo genetico ecc.. Le proteine sono le macromolecole biologiche più
abbondanti nelle cellule e sono sempre formate dalla stessa serie di 20 amminoacidi
sia che appartengano alle specie batteriche che agli organismi superiori (fig.2). A
differenza di polimero sintetico come il polietilene o il
nylon che presentano proprietà limitate, le proteine
sono molto più versatili perché presentano unità
monometriche con catene laterali diverse per
struttura, dimensione e carica. Queste possono
essere idrocarburiche, come nel caso della valina,
leucina ed isoleucina, possono presentare un gruppo
basico come nella lisina, arginina e istidina o
possono
contenere un gruppo carbossilico come
nell’acido aspartico e glutammico. A causa della
Figura 4 - Reazione di condensazione disposizione
tetraedrica del carbonio, tutti gli
tra due amminoacidi con la formazione
di un dipeptide e l'eliminazione di una amminoacidi, ad eccezione del più semplice, la
molecola d'acqua
glicina, posseggono un carbonio asimmetrico. Dei
due possibili stereoisomeri che si trovano nelle proteine idrolizzate solo uno due
enantiomeri, quello definito L, è presente e la configurazione è la stessa per tutti gli
amminoacidi (fig. 3).
Le molecole di amminoacidi nelle proteine sono
legate
insieme
attraverso
reazioni
di
condensazione con eliminazione di una molecola
d’acqua e la formazione del legame chiamato
peptididico (fig. 4).
Potendo un amminoacido legarsi ad altri 19, il
Figura 5 – Forme limiti di risonanza numero delle combinazioni cresce rapidamente
per il legame peptidico
all’aumentare delle dimensioni della catena.
Comunque, lo studio degli archivi di sequenze
proteiche mostra chiaramente che i residui
amminoacidici non sono presenti con uguale
frequenza
nelle
proteine
e
le
sequenze
rappresentano solo una piccola parte di tutte le
possibili combinazioni. Ad esempio, il triptofano e
la cisteina sono residui rari nelle proteine, meno
del 2% di tutti i residui, mentre l’alanina, glicina e
leucina sono presenti con una frequenza dal 7 al
9%.
Tra tutte le conformazioni che una proteina che
contiene centinaia di atomi può assumere, solo
una (o poche) sono quelle che predominano nelle condizioni biologiche. Quando una
proteina è in uno di questi stati conformazionali funzionali è detta nativa. Esistono una
serie di vincoli che determinano la conformazione delle proteine. Il primo deriva dal
fatto che il legame peptidico formato tra i gruppi carbonilico e amminico non può
ruotare liberamente.
Questo può essere giustificato assumendo che il legame peptidico possa essere
descritto come un ibrido di risonanza delle due formule limiti I e II mostrate in fig. 5.
3
Figura 6 - Legame peptidico. i due
carboni alfa sono separati da tre legami
che sono coplanari
Dalla teoria della risonanza deriva che la molecola reale non corrisponde né alla
formula limite I né a II, ma è una combinazione delle caratteristiche di entrambe. Così
il legame C-N avrà nella realtà un non trascurabile carattere di doppio legame e una
struttura più o meno planare, con angoli vicini a 120°, necessaria per permettere la
coniugazione del doppietto elettronico. Studi cristallografici ai raggi X confermano che
la lunghezza del legame peptidico è intermedio (1,33 Å) tra quello aspettato per un
semplice legame C-N (1.45 Å) e quello di un doppio legame C=N (1.25 Å).
L'accentuato carattere di doppio legame tra C e N impedisce la libera rotazione
intorno a questo legame e porta i sei atomi del gruppo peptidico ad essere coplanari
(fig.6).
Lo scheletro della catena polipetidica si può quindi considerare come una serie di
piani rigidi in cui i piani consecutivi hanno in comune un legame Cα–C o N–Cα in grado
esibire mobilità rotazionale. Come risultato della restrizione rotazionale sono possibili
due conformazioni, quella trans o quella cis. Per motivi sterici la configurazione trans è
preferita di un fattore 103, tranne quando l’ultimo amminoacido della catena è una
prolina. In tal caso, a causa della natura ciclica del residuo che determina una minore
repulsione sterica tra i lati della catena, la differenza energetica tra configurazione cis
e trans è solo di un fattore 4.
La
conformazione
della
proteina
è
determinata dai due angoli diedri, Φ e Ψ,
che riflettono le rotazioni intorno a
ciascuno dei due legami (N—C per Φ e
C —C per Ψ) che si ripetono lungo lo
scheletro della molecola e che possono
assumere solo alcuni valori (fig.7). Anche
se in linea di principio Φ e Ψ possono
assumere qualsiasi valore compreso tra 180° e + 180°, molti non sono permessi
per motivi di impedimento sterico tra gli atomi dello scheletro e le catene laterali. Le
conformazioni stericamente proibite sono quelle per cui la distanza interatomica di
un’interazione non covalente è inferiore alla corrispondente somma di raggi di van der
Waals.
In base ai raggi atomici degli
elementi della catena, sono
possibili soltanto valori di Φ
e Ψ che cadono in alcune
zone del cosiddetto grafico
di Ramachandran (fig.8), dal
nome del fisico indiano G.N.
Ramachandran che alla fine
anni ’60, per primo calcolò
le
regioni
stericamente
Figura 8 - Grafico di Rachamandran per due residue di L-alanina. Le permesse, e in cui gli angoli
zone verdi rappresentano conformazioni con valori di Φ e Ψ
Ψ sono riportati in funzione
permessi in cui non si hanno interazioni steriche
di Φ.
Figura 7 - La rotazioni dei legami N-C e C -C
descrivono
due
angoli
diedri,
chiamati
rispettivamente Φ e Ψ
4
Le zone in verde scuro sono quelle largamente permesse, mentre le zone in verde
chiaro indicano le conformazioni, comunque possibili, che hanno distanze di van der
Waals al limite inferiore dell’accettabilità. Solo il 25-30 % del diagramma è occupato
da zone stericamente permesse.
Nelle proteine i legami covalenti limitano le forme che queste possono assumere, ma
sono le interazioni non covalenti che determinano le conformazioni stabili e al tempo
stesso flessibili che permettono ad esse di svolgere le loro funzioni biologiche.
Sono le proprietà delle catene laterali le principali responsabili del ripiegamento di una
catena polipeptidica. Le interazioni di una
catena laterale con un'altra e con le
molecole presenti nel mezzo circostante
possono determinare il ripiegamento della
proteina in una conformazione stabile.
Queste interazioni sono molto più deboli dei
legami covalenti (l’energia è ad esempio
dell’ordine dei 350 kJ/mole per i legami CC) e possono variare da 0,1 a 30 kJ/mole. I
più importanti sono: il legame idrogeno, le
interazioni ioniche, le forze di van der Waals
e le interazioni idrofobiche2 (fig.9).
Analizzando il contributo delle interazioni
alla stabilità delle proteine, si osserva che
quelle predominanti sono le interazioni
idrofobiche. Le molecole di acqua formano
Figura 9 - Interazioni deboli presenti nella tra loro numerosi legami idrogeno. La
proteina
presenza di una molecola rompe i legami
idrogeno e le molecole d’acqua tendono a circondare la struttura idrofobica
riarrangiandosi in un guscio di molecole molto più ordinato unite da legami idrogeno
chiamato strato di solvatazione. Poiché il numero di modi con cui le molecole d’acqua
formano legami idrogeno sulla superficie di un gruppo non polare è inferiore a quello
che farebbero in sua assenza si ha una diminuzione sfavorevole di entropia del
sistema. La conseguenza è che la catena polipeptidica tende ad avvolgersi in modo
tale da esporre in superficie le catene laterali polari e posizionando all’interno quelle
non polari in modo da ridurre la dimensione dello strato di solvatazione, poiché meno
molecole d’acqua sono ordinate intorno alle molecole apolari. Questo determina un
aumento di entropia che costituisce la forza termodinamica principale alla
stabilizzazione della proteina.
Le strutture delle proteine sono estremamente complesse, ma alcuni elementi si
trovano frequentemente. Questi elementi rappresentano la cosiddetta struttura
secondaria delle proteine. Con il termine di struttura secondaria si intendono le
disposizioni regolari della catena polipeptidica principale, che vengono classificate
senza fare riferimento al tipo di catena laterale degli amminoacidi o con altri segmenti
della proteina.
2
Vedi lezione Treccani: Le forze intermolecolari
5
Esse sono stabilizzate da legami
idrogeno fra il gruppo amminico e il
gruppo
carbonilico
della
catena
principale. La regolarità di una
struttura si ha quando ogni angolo φ e
ψ rimane invariato all’interno del
segmento di proteina.
Si possono distinguere tre tipi
principali di strutture secondarie:
elica , foglietto β e reverse turn.
I segmenti di catena polipeptidica che
non sono in
elica, foglietto β o turn
Figura 10 - Struttura secondaria elica di una proteina
assumono la conformazione chiamata
loop o random coil, struttura non ripetitiva né regolare, spesso priva di legami
idrogeno tra gli aminoacidi che la compongono. Circa il 70-80 % degli aminoacidi delle
Struttura secondaria alfa elica di una proteina
proteine
globulari assume la conformazione regolare tipica di uno dei tre tipi di
struttura secondaria.
L’ elica è la struttura secondaria più adottata dalla catena polipeptidica delle
proteine: circa il 32-38 % degli aminoacidi delle proteine globulari di struttura
tridimensionale nota assume questa conformazione (fig. 10).
Essa fu descritta per la prima volta nel 1951 da Linus Pauling e Robert Corey
(California Institute of Technology), i quali proposero una struttura stabile basata su
gli angoli e sulle distanze di legame calcolate sperimentalmente sulla base dell’analisi
cristallografica per gli amminoacidi e i peptidi. La prova sperimentale dell’esistenza di
queste strutture si ebbe con la determinazione della struttura tridimensionale delle
mioglobina (John Kendrew, 1960) e dell’emoglobina (Max Perutz, 1961), due proteine
che presentano eliche come elementi di struttura secondaria.
L’α elica presenta un avvolgimento destrorso ed è caratterizzata da un angolo φ = 57° ed un angolo ψ = -47°, che corrispondono alla posizione nel quadrante in basso a
sinistra del plot di Ramachandran. Ogni giro d’elica contiene 3,6 residui ed ogni
amminoacido si estende per 1,5 Å lungo la catena determinando un passo dell’elica
pari a 5,4 Å (fig. 10B).
La struttura è stabilizzata da legami idrogeno che si
formano tra l’atomo di idrogeno legato all’atomo di azoto
di un legame peptidico e l’atomo di ossigeno carbonilico
del quarto amminoacido successivo (fig.10A) e ciascun
legame peptidico, tranne quelli vicino all’estremità
dell’elica, partecipa alla formazione di legami idrogeno. Le
catene laterali degli aminoacidi che appartengono ad un’α
Figura 11 - Struttura della elica destrorsa sono rivolte verso l’esterno e verso
prolina
l’estremità
N-terminale
dell’elica
così
da
evitare
interferenze steriche con la catena polipeptidica principale e tra le catene laterali
stesse. La sequenza amminoacidica influenza la capacità di formare delle eliche. Ciò
dipende dalle proprietà del residuo R dell’amminoacido che influenzano i valori
caratteristici degli angoli Φ e Ψ. Ad esempio, residui con gruppi carichi sono
fortemente influenzati dal pH dell’ambiente. A pH 7, i gruppi carbossilici del
6
glutammato adiacenti si respingono così come i residui carichi positivamente
dell’arginina, destabilizzando la formazione dell’elica. Altro esempio è la presenza della
prolina (fig.11) che presenta un atomo di azoto del gruppo amminico che fa parte di
un anello rigido e che non rende possibile la rotazione del legame N-Ca introducendo
un ripiegamento che destabilizza l’elica. Inoltre, non avendo l’idrogeno sul gruppo
amminico, non può formare il legame H con altri residui.
Nel 1951 Pauling e Corey ipotizzarono anche l’esistenza di un’altra struttura
secondaria: la conformazione . Essa è formata in media da 5 a 10 aminoacidi, con la
catena polipeptidica quasi completamente estesa. Le catene polipeptidiche sono
disposte l’una accanto all’altra in modo tale che si possano formare legami idrogeno
tra i gruppi CO di un filamento e i gruppi NH del filamento β adiacente e viceversa,
formando una struttura che presenta una serie di pieghettature e che è detta foglietto
β. Le catene laterali degli aminoacidi che compongono il foglietto β seguono lo stesso
andamento, per cui puntano alternativamente sopra e sotto il foglietto β.
Le catene polipeptidi che possono interagire
fra loro a formare i foglietti β in due modi
diversi:parallele o antiparallele a seconda che
abbiano lo stesso orientamento o un
orientamento opposto del legame NH-CO (fig.
12). Gli angoli diedri (φ, ψ) della catena
polipeptidica in conformazione foglietto β
cadono nella zona permessa del grafico di
Ramachandran corrispondente al quadrante in
alto a sinistra: φ=-139°, ψ=135° (parallelo)
e φ=-119°, ψ=113° (antiparallelo).
I reverse turn sono ripiegamenti che
collegano due filamenti β antiparalleli
Figura 12 - Struttura secondaria β elica. a) adiacenti. In questo caso sono due gli
foglietto ripiegato antiparallelo; b) foglietto
amminoacidi non coinvolti nei legami idrogeno
ripiegato parallelo
tra i filamenti β in quanto si forma un legame
idrogeno tra il gruppo CO dell’aminoacido 1 e il gruppo NH dell’amminoacido 4. Questo
tipo di ripiegamento si osserva nelle proteine globulari, che presentano una struttura
ripiegata e compatta.
Le regioni loop o random coil sono costituite da zone della catena polipeptidica che
non assumono alcun tipo di struttura secondaria nel senso che le coppie (φ, ψ) degli
amminoacidi che li compongono presentano valori diversi e non costanti ed hanno la
funzione di collegare elementi di struttura secondaria.
La disposizione spaziale degli atomi di una proteina è definita come struttura terziaria
e si distingue da quella secondaria in quanto questa riguarda la disposizione dei
residui amminoacidici adiacenti nella sequenza polipeptidica. La struttura terziaria
quindi, prende in considerazione delle interazioni a lungo raggio nella sequenza
primaria della proteina, che possono essere legami deboli e talvolta legami covalenti
come ponti disolfuro.
Per proteine globulari di 140 o meno residui, la struttura tridimensionale ha una forma
sferica compatta formata da motivi di struttura secondaria con poche irregolarità
strutturali. Per proteine più grandi la struttura terziaria può essere organizzata in più
7
unità strutturali chiamate domini. I diversi domini della proteina, interagiscono tra loro
con minori interazioni rispetto a quelli tra gli elementi strutturali secondari all’interno
del dominio. In molti casi un dominio di una proteina mantiene la sua struttura
terziaria anche se viene separato mediante una scissione idrolitica dal resto della
catena polipeptidica.
Molte proteine sono formate da più catene polipeptidiche e le interazioni tra queste
catene ne definiscono la struttura quaternaria. Queste strutture possono essere
dovute a proteine con diverse o uguali subunità. Ad esempio emoglobina è un
tetramero formato da due differenti subunità denominate alfa e beta, mentre la
proteasi
HIV
è
un
omodimero (fig.13).
Le
subunità
sono
mantenute insieme da un
gran numero interazioni
non
covalenti
che
nonostante
siano
individualmente
deboli
determinano
una
notevole
stabilità.
La
struttura quaternaria è
mostrata da proteine che
Figura 13 - Strutture quaternarie. A Proteasi HIV; B Emoglobina
svolgono
funzioni
catalitiche con la formazione di siti
catalitici o di legame con il
substrato all’interfaccia tra le
subunità. Un ulteriore vantaggio
di
queste
strutture
è
che
l’interazione della proteina con il
substrato o con i leganti causa
cambi conformazionali all’interno
della
struttura
ed
offre
la
possibilità di regolare l’attività
biologica. Questa è la base delle
cosiddetta regolazione allosterica
negli enzimi3.
Le proteine vengono sintetizzate
nelle cellule sui ribosomi come
Figura 14 – Nell’esperimento di Anfinsen, la ribonucleasi sequenza lineari di amminoacidi.
viene denaturata con urea e mercaptolo per ridurre e Successivamente
queste
si
scindere i ponti disolfuro. La rinaturazione ristabilisce i ponti
avvolgono (folding) per assumere
disolfuro e riporta la struttura nel suo stato nativo
la loro conformazione nativa che è
quella funzionale. La struttura tridimensionale della proteina è determinata dalle
interazioni tra i residui amminoacidici, quindi dalla sua struttura primaria. Queste
3
In una proteina allosterica il legame con un ligando a livello di uno specifico sito di legame modifica le proprietà di un
altro sito sulla stessa molecola proteica attraverso una modificazione conformazionale
8
interazioni sono distrutte dall’azione della temperature, in soluzioni alcaline o acide o
per addizione di sostanze come alcol o urea. Questo processo, che determina la
perdita della struttura terziaria e dell’attività della proteina è detto denaturazione.
Molte proteine, dopo essere state denaturate, sono in grado di ripiegarsi
spontaneamente nella propria conformazione nativa riacquistando la loro attività
biologica. L’esperimento di C. Anfinsen (1957) sulla ribonucleasi A (RNasi) ha
dimostrato che questa proteina se denaturata e ridotta riassume la sua conformazione
nativa e riforma i corretti 4 punti disolfuro quando viene rimosso il denaturante e il
riducente (fig.14). Il ruolo di tale proteina è quello di catalizzare l’idrolisi delle
molecole di RNA a livello dei legami fosfodiestere e pertanto la sua attività si misura in
base alla capacità di idrolizzare le molecole di RNA.
Questo risultato indica che la sequenza
peptidica
contiene
tutta
l’informazione
necessaria
perché
la
proteina
possa
avvolgersi nella sua struttura tridimensionale
dello stato nativo. Dal punto di vista
termodinamico
lo
stato
nativo
nelle
condizioni fisiologiche costituisce un minimo
dell’Energia Libera di Gibbs.
Ma una volta che la proteina è sintetizzata
come fa a ripiegarsi nella conformazione
nativa, dato che l’esplorazione casuale di
tutte le possibili conformazioni fino a
raggiungere quella corretta non è possibile?
Figura 15
- L’asse verticale rappresenta
l‟energia libera interna di ogni specifica Infatti,
ammettendo
soltanto
10
conformazione
mentre
l’asse
orizzontali
possibili per ogni residuo
rappresenta il numero di conformazioni (quindi conformazioni
l’entropia conformazionale). La forma ad imbuto amminoacidico,
una sequenza di 100
descrive la progressiva riduzione dello spazio
presenta
un
numero
di
conformazionale. Inizialmente sono presenti amminoacidi
100
molte strutture che diminuiscono fino ad
conformazioni possibili paria a 10
e se per
arrivare alla proteina nello stato nativo che è
caratterizzata
da
un
unico
sottostato ogni cambio di conformazione è necessario
conformazionale.
un tempo pari a circa 10-13 s, il tempo totale
per provare tutte le conformazioni sarebbe pari a 10 87 s (l’età dell’Universo è stimata
in 20 miliardi di anni che corrispondono a 6.1017 s)! Questo problema è noto come
paradosso di Levinthal (1968). Dato che i tempi di folding vanno da qualche secondo
ad alcuni minuti, è evidente che l’evoluzione ha trovato una soluzione efficace a
questo procedimento. Le cellule d’Escherichia coli, ad esempio, sintetizzano una
molecola proteica di 100 amminoacidi in circa 5 secondi alla temperatura di 37°C.
Le proteine si ripiegano seguendo vie dirette senza ricercare casualmente la
conformazione nativa tra le molte possibili. Sono stati proposti diversi modelli per
spiegare questo processo. Il primo, detto gerarchico, prevede che le proteine
inizialmente diano luogo a strutture secondarie (α eliche, foglietti β) seguite da
interazioni a lungo raggio tra queste che portano alla formazione di domini ed infine
alla conformazione nativa (fig. 15). Nel secondo modello, detto globulo fuso (molten
globule) il ripiegamento iniziale della proteina è favorito dalla formazione di uno stato
compatto dovuto a interazioni idrofobiche tra residui non polari (collasso idrofobico). Il
molten globule ha molte delle strutture secondarie dello stato maturo ma è meno
9
compatto e le interazioni all'interno della proteina non sono forti in quanto molte
catene amminoacidi non hanno ancora assunto una giusta conformazione. È probabile
che la maggior parte delle proteine si ripieghi attraverso un processo che segue i due
modelli, attraverso conformazioni intermedie diverse il cui numero tende a diminuire
via via che il processo di avvolgimento tende verso la struttura finale nativa.
L’andamento termodinamico del processo di ripiegamento può essere rappresentato
come un imbuto il cui restringimento
rappresenta una diminuzione delle specie a
diversa conformazione. Le depressioni lungo
le parti esterne dell’imbuto rappresentano
conformazioni transitorie che persistono fino
a che mediante attivazione termica casuale
viene superata la barriera energetica con
decadimento in conformazione ad energia
minore.
Figura 16 - I chaperoni si presentano forma di Molte proteine per avvolgersi hanno bisogno
capsule e sono in grado di fornire un ambiente
della presenza di altre proteine dette
sicuro per il giusto ripiegamento della proteina.
Una volta che la proteina ha assunto la corretta chaperoni molecolari, che interagiscono con
struttura viene rilasciata
la
proteina
parzialmente
ripiegata
facilitandone il corretto ripiegamento impedendo l’associazione scorretta di zone
idrofobiche (fig. 16).
La comprensione dei meccanismi molecolari, che sono
alla base del folding delle proteine rappresenta un
importantissimo obiettivo in quanto esso fornirebbe la
possibilità di curare molte patologie associate ai
processi di avvolgimento in una forma sbagliata della
proteina (misfolding).
Ad esempio nella fibrosi cistica è presente un difetto
nella proteina transmembrana che agisce come un
canale degli ioni cloro nelle cellule epiteliali (1480
amminoacidi) che non si avvolge correttamente a
causa di una mutazione con la delezione di un
amminoacido
(Phe
508).
Essa
determina
principalmente la produzione di secrezioni molto ricche
in sodio e cloro; il muco è denso e vischioso e tende ad
ostruire i dotti nei quali viene a trovarsi.
In questi ultimi anni, grazie all’utilizzo di potenti
Figura 17 - Alcuni esempi di strutture computer, si sono sviluppati dei metodi computazionali
di proteine analizzate mediante il aventi lo scopo di predire la struttura tridimensionale di
metodo Rosetta.
una proteina partendo dalla sola conoscenza della sua
A: fattore di trascrizione MarA legato
al DNA;
sequenza amminoacidica e di comprendere come
B: batteriocina AS-48;
avviene il ripiegamento nelle strutture native.
C: Secondo dominio del MutS.
A sinistra sono riportate le struttura Tra questi, c’è ad esempio l’algoritmo sviluppato dal
determinate mediante i raggi x, gruppo di David Baker chiamato Rosetta, con grande
mentre a destra le strutture previste
capacità predittiva. Esso divide la sequenza primaria
teoricamente
in gruppi di alcuni residui (da 3 a 9) ed effettua una
10
ricerca su frammenti di strutture proteiche note presenti in un database e provenienti
da strutture cristalline, generando così per ogni frammento una serie di strutture
possibili. Vengono poi analizzate tutte le combinazioni e le varie conformazioni
valutate con varie funzioni di scoring in modo da trovare quella a più bassa energia.
Questo metodo offre un valido aiuto nella ricerca sulle possibili strutture locali
restringendo notevolmente il numero di conformazioni da analizzare. Questo metodo
di previsione ha partecipato con grande successo ad una competizione chiamata CASP
(Critical Assessment of Methods for Protein Structure Prediction), nella quale team di
ricercatori da tutto il mondo cercano di prevedere le strutture di proteine la cui
struttura è stata recentemente determinata sperimentalmente, ma che non è ancora
stata resa pubblica. In fig. 17 sono riportate alcune strutture previste da Rosetta
confrontate con quelle risolte sperimentalmente mediante analisi ai raggi X.
11