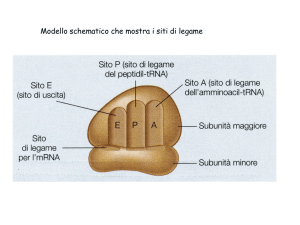
Ogni passaggio della procedura di purificazione determina la separazione delle
proteine totali presenti nel campione in una serie di frazioni (frazionamento).
Per ciascuna frazione è determinata la quantità di proteine totali e le unità di
attività enzimatica della proteina di interesse.
Una unità enzimatica (U) è definita come la quantità di enzima richiesta per convertire
1 µmol di substrato in prodotto in 1 min in condizioni definite (generalmente 25 o
30 °C, ad un valore di pH ottimale).
mg o U di proteina di interesse nella frazione
RESA
mg o U di proteina di interesse nella preparazione originale
Il grado di purezza di un enzima in una particolare frazione è espresso tramite
l’attività specifica, la quale pone in relazione l’attività enzimatica totale con il
contenuto totale di proteine presenti nella preparazione.
ATTIVITA’
SPECIFICA
U di enzima di interesse nella frazione
mg di proteine totali nella frazione
Nel corso di una purificazione ottimale
la resa diminuisce e
l’attività specifica aumenta
proteine Enzima
totali
(mg)
(mg)
Resa
%
Purezza
(%)
U totali
As
(U/mg)
Omogenato
1000
100
100%
10%
10000
10
1° stadio di
purificazione
100
50
50%
50%
5000
50
2° stadio di
purificazione
27
25
25%
92%
2500
92
3° stadio di
purificazione
20
19.8
19.8%
99%
1980
99
Saggio
colorimetrico
Stima da
SDS-PAGE
Saggio di
attività
enzimatica
Valore ipotetico
proteina pura =
100 U/mg
La messa a punto di un procedimento sperimentale di purificazione di una proteina
richiede la possibilità di:
1. Identificare e quantizzare la proteina di interesse fra tutte le altre proteine
del campione di partenza (gli ENZIMI possono essere identificati sulla
base della reazione che essi catalizzano tramite un opportuno SAGGIO
ENZIMATICO)
2. Misurare la quantità di proteine totali nel campione
3. Valutare la presenza di proteine contaminanti nel campione di interesse
Identificare e quantizzare la proteina di interesse fra
tutte le altre proteine del campione di partenza
E’ necessario disporre di un opportuno saggio che consenta di
seguire la proteina durante i vari passaggi del procedimento
di purificazione
E’ importante che il saggio disponibile sia eseguibile
rapidamente su molti campioni
Il saggio deve indicare in maniera affidabile la quantità della
proteina desiderata presente ai vari stadi di purificazione
Inoltre il saggio deve essere eseguibile con una piccola quantità
di campione proteico
Specifico
Quantitativo
Gruppo di enzimi che
catalizzano l’
l’idrolisi
di monoesteri del fosfato
FOSFATASI
R-O-PO32- + H2O
R-O-H + HO-PO32-
NO2
NO2
FOSFATASI
O -PO32- + H2O
O-H + HO-PO32-
p-nitrofenil-fosfato
p-nitrofenolo
INCOLORE
GIALLO
Le reazioni che richiedono nucleotidi piridinici sono molto
usate nei metodi enzimatici di analisi.
Tali coenzimi (NAD, NADH, NADP, NADPH) sono ideali
perché sono usati stechiometricamente in un gran numero
di reazioni di ossido-riduzione.
Composto ridotto + NAD+
NAD+
(non assorbe a 340 nm)
Composto ossidato + NADH
NADH
(assorbe a 340 nm)
Saggio enzimatico non specifico, dal momento che evidenzia
tutti gli enzimi che usano come coenzima il NAD+.
proteine Enzima
totali
(mg)
(mg)
Omogenato
1000
100
Resa
%
Purezza
(%)
U totali
As
(U/mg)
100%
10%
10000
10
1° stadio
purificazione
5000
2° stadio
purificazione
2500
3° stadio
purificazione
1980
Saggio
colorimetrico
Stima da
SDS-PAGE
Valore ipotetico
Saggio di
proteina pura = 100
attività
U/mg
enzimatica
La complementarità geometrica e chimica fra piccole
molecole biologiche (LIGANDI) e le strutture dei
loro bersagli macromolecolari (RECETTORI) gioca un
ruolo molto importante all’interno dei processi
biologici.
Concetti basilari per lo studio del binding
recettoriale
I recettori sono presenti in concentrazioni molto piccole nei tessuti.
Incubando il tessuto contenente i recettori R ed il ligando
radiomarcato D in opportune condizioni sperimentali si formerà il
complesso RD secondo l’equazione
R+D
RD
Quando il sistema raggiunge l’equilibrio in ogni provetta avremo:
Radioligando
Tessuto
Tampone
di incubazione
Incubazione
Ligando radiomarcato D
Equilibrio
Recettore RD
Recettore libero R
Il recettore libero R non potrà essere misurata ma possiamo
misurare la quantità di complesso ligando-recettore RD.
Per poter calcolare la quantità del complesso RD è necessaria
un’operazione di separazione del legato (Bound) dalla quantità
di ligando radiomarcato D libero in soluzione non legato al
tessuto (Free).
Equilibrio
Separazione
Metodi di separazione del RD dal mezzo di reazione
Filtrazione
Il metodo più semplice e classico è quello della
filtrazione su membrane in fibra di vetro
Whatman GF/A, GF/B, GF/C dove varia la porosità 0.7 µm-2.7 µm
Il tessuto contenente il recettore legato al radioligando
aderisce al filtro mentre il radioligando libero passa
attraverso la membrana.
[R] [L] ([Rtot] - [RL]) [L]
[RL] =
=
Kd
Kd
[RL] Kd = [Rtot] [L] - [RL] [L]
[RL] Kd + [RL] [L] = [Rtot] [L]
[RL] (Kd + [L] ) = [Rtot] [L]
[RL] =
[Rtot] [L]
Kd + [L]
Bound
(pmol/mg protein)
0.15
Binding totale
Binding non specifico
Binding specifico
0.10
Le tre curve corrispondenti
sono riportate in figura
0.05
0.00
0.1
0.2
0.3
0.4
0.5
0.6
[3H]spiroperidolo, (nM)
Visualizzando solo la
curva
del
binding
specifico
possiamo
individuare la Kd e la
Bmax
Bound
(pmol/mg protein)
0.100
0.075
Bmax
0.050
0.025
0.000
0.00
Kd
0.25
0.50
0.75
[3H]spiroperidolo, (nM)
1.00
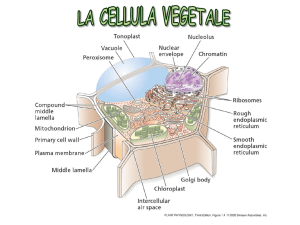
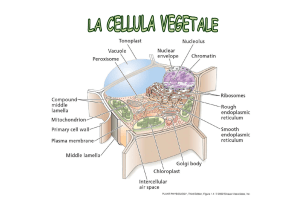
METODI DI Estrazione delle proteine
Compartimenti circoscritti da membrana
Nucleo
Reticolo
Endoplasmatico
Perossisoma
Lisosoma
Vescicola
Mitocondrio
Apparato
Del Golgi
Membrana
plasmatica
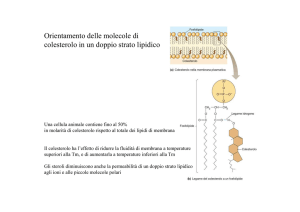
Doppio strato (bilayer) lipidico
Bilayer
lipidico
(5 nm)
Molecole
lipidiche
Molecole
proteiche
Le membrane biologiche sono composte di…
1. Lipidi – il doppio strato lipidico crea una barriera
idrofobica
Per la maggior parte fosfolipidi ma anche glicolipidi e
colesterolo
2. Proteine – conferiscono specificità alle funzioni svolte dalla
membrana
a. Proteine di membrana periferiche legate alla superficie
della membrana
b. Proteine di membrana integrali – contengono domini
idrofobici e idrofilici anfipatiche
c. Glicoproteine (integrali) – contengono molecole glucidiche
recettori di superficie
Struttura dei Fosfolipidi
gruppo di testa
polare
(idrofilico)
testa
code
code non
polari
(idrofobiche)
doppio
legame cis
Ricostruzione di un bilayer lipidico
+ H2O
Tutte le membrane biologiche sono bilayers lipidici
Le proteine conferiscono proprietà uniche a ciascun tipo
di membrana
Classi di Proteine di Membrana
ancorate
periferiche
superfice
extracell.
integrali
Superfice
citosolica
ancorate
Come può un legame peptidico polare essere inserito
nel core idrofobico di un bilayer fosfolipidico?
estremità
amino (N-)
terminale
estremità
carbossi (C-)
terminale
Le - eliche transmembrana tipicamente
sono costituite da 20-25 aminoacidi la
maggior parte dei quali idrofobici.
triptofano
fenilalanina
prolina
isoleucina
In una -elica i legami peptidici
polari si trovano all’interno e i
gruppi R delle catene laterali
protrudono all’esterno
3.6 residui/giro
Glicoforina: tipica
proteina che attraversa
la membrana
plasmatica una volta
Batteriorodopsina,
tipica proteina che attraversa la membrana plasmatica sette volte
Associazione di proteine di membrana
con un bilayer lipidico
Periferche
Integrali
Transmembrana
-elica
foglietto-
attaccate
a proteine
legate
a lipidi
SPAZIO
EXTRACELLULARE
Bilayer
lipidico
CITOSOL
Legame covalente
a molecola lipidica
Legame debole,
non-covalente, ad
un’altra proteina
di membrana
Proteine di membrana
• Nelle cellule animali, il 50% della massa del
plasmalemma sono proteine
• Le proteine di membrane hanno molte funzioni:
Trasportatori
Collegamento
Recettori
Enzimi
SPAZIO
EXTRACELLULARE
CITOSOL
Membrane differenti esprimono proteine differenti funzioni differenti
Tutte le membrane biologiche sono bilayers
di fosfolipidi
Le proteine in ciascun tipo di membrana le
conferiscono proprietà uniche
DETERGENTI
Si usano per solubilizzare
proteine intrinseche di
membrana
Ionici:
SDS, sodio deossicolato,
CTAB e CHAPS
Non – ionici:
Triton X-100 e Nonoident P-40
C o n d i z i o n i
p e r
l ’ e s t r a z i o n e
• L ’ e s t r a z i o n e s e p o s s i b i l e v a f a tta a freddo, specialmente per i
metodi meno delicati; talvolta puo’ essere necessario farla sotto
a z o t o p e r r i d u r r e l ’ o s s i g e n o i n so l u z i o n e .
•L’estrazione non va mai fatta a secco, usualmente si fa in
tampone bassa G, pH neutro, con inibitori di proteasi, EDTA.
•Per proteine di membrana, aggiungere un detergente non ionico e
non denaturante (es. Triton X100, Octyl-glucoside, Tween).
•Per proteine facilmente ossidabili, aggiungere DTT o b-ME
•Per proteine molto delicate, aggiungere glicerolo fino al 50%
Attenzione alle letture a 280 nm in caso di queste aggiunte.