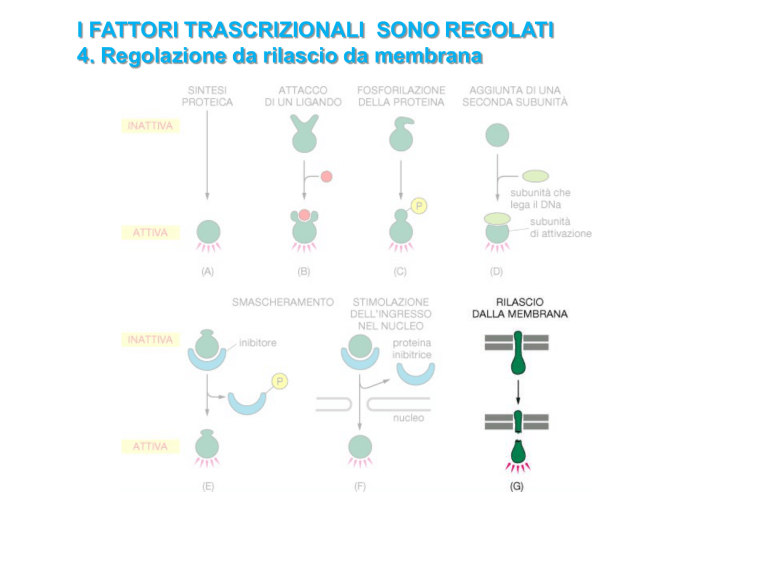
I FATTORI TRASCRIZIONALI SONO REGOLATI
4. Regolazione da rilascio da membrana
REGOLAZIONE DA RILASCIO DA MEMBRANA: il fattore SREBP
Nell’uomo sono state
identificate tre proteine nella
famiglia, denominate SREBP1a, 1c e SREBP-2.
Tutte le proteine della
famiglia SREBP hanno una
struttura tripartita simile che
consiste di:
1) un dominio di legame al
DNA N-terminale di circa 400
aminoacidi che ha una
struttura HLH
2) una regione centrale di
circa 80 aa idrofobica che
contiene due segmenti
transmembrana
3) una regione regolatoria
C-terminale
I GENI REGOLATI DALLE PROTEINE SREBP
SREBP 2 e SREBP 1c regolano l’espressione di geni diversi.
SREBP-2 attiva preferenzialmente i geni del metabolismo del colesterolo
SREBP-1c attiva preferenzialmente i geni della biosintesi degli acidi grassi, dei
trigliceridi e fosfolipidi.
REGOLAZIONE DA RILASCIO DA MEMBRANA: il fattore SREBP
Attivazione mediante proteolisi controllata
L’attivazione avviene in 2 steps
che sono regolati dagli steroli:
S1P: una prima proteasi taglia SREBP
nel sito 1 che si trova nel mezzo del
loop che si affaccia nel lume del Golgi
rompendo il legame covalente tra le
due regioni transmembrana
S2P:una seconda proteasi taglia il
frammento N-Terminale all’interno
del dominio transmembrrana
rilasciando il dominio N-terminale
maturo nel citosol
S1P è sotto lo stretto controllo degli
steroli
LA PROTEINA SCAP (SREBP-cleavage-activating protein)
L’attivazione del taglio proteolico
richiede l’intervento della proteina
SCAP che agisce come sensore del
contenuto di colesterolo della
membrana del RE.
SCAP è una proteina integrale di
membrana di 1,276-aminoacidi
formata da due domini distinti:
il dominio N-Term ha 8 eliche
transmembrana 5 delle quali (2-6)
comprendono il dominio SSD
(STEROL-SENSING DOMAIN).
Il dominio C-term (WD) media
interazioni proteina-proteina e
interagisce con la regione
regolatoria C-term di SREBP.
In presenza di steroli, SREB e SCAP
si trovano nelle membrane del RE
mentre S1P e S2P risiedono nelle
membrane del Golgi.
LA PROTEINA INSIG
La ritenzione nel RE richiede
l’interazione tra il SSD di SCAP e la
proteina INSIG.
Quando è presente nelle membrane
del RE, il colesterolo si lega al
dominio SSD di SCAP e induce un
cambio conformazionale nel suo
loop citosolico localizzato tra le
eliche 6 e 7.
Il cambio conformazionale aumenta
l’affinità per la proteina INSIG e
distrugge l’interazione di SCAP con
il rivestimento COPII e ciò risulta
nella ritenzione del complesso nel
RE.
In assenza di steroli SCAP e INSIG
non interagiscono e il complesso è
libero di essere incorporato nelle
vescicole rivestite da COPII e di
andare all’apparato del Golgi per
essere processato.
CONTROLLO TRASCRIZIONALE DEL
METABOLISMO
CONTROLLO TRASCRIZIONALE DELL’OMEOSTASI DEL
GLUCOSIO
Glucosio
X
Pancreas
endocrino
Insulina
Glucagone
Glucosio
Glucosio
Glicogeno
Trigliceridi
Fegato
Tessuto adiposo
REGOLAZIONE DELL’ESPRESSIONE GENICA
Alti livelli di glucosio
Promotore del gene dell’ insulina
Una regione conservata di ∼340 bp a monte del sito di
inizio della trascrizione è responsabile sia della tessutospecificità sia della regolazione metabolica del gene
dell’insulina
Inibizione della trascrizione del gene dell’insulina da prolungata
esposizione al glucosio
Glucotossicità:
stress ossidativo
induzione di JNK e attivazione di cjun
dedifferenziazione delle cellule beta
attraverso upregolazione di –cmyc
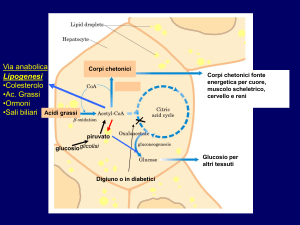
Lipotossicità (diabete di tipo 2)
produzione di ceramide
azione indiretta attraverso
attivazione di JNK
azione diretta attraverso inibizione
di PKB e traslocazione di FOXO1 nel
nucleo che compete con PDX1 per il
legame al promotore
REGOLAZIONE DELL’ESPRESSIONE GENICA
Bassi livelli di glucosio
AZIONE DELL’INSULINA SUI TESSUTI PERIFERICI
OMEOSTASI DEL COLESTEROLO
FXR (farnesol X
receptors)
FXR KO -> alti
livelli di acidi
biliari nel sangue
FXR inibisce
CYP7A
(colesterolo 7
idrolasi che serve
per la sintesi degli
acidi biliari)
FXR attiva la
trascrizione del
gene BSEP
(pompa di
escrezione degli
acidi biliari)
Il ligando di LXR è
oxysterol
LXR (liver X
receptors)
LXRa KO ->
sensibili a diete
ricche di
colesterolo
LXR attiva ABC
genes (aumentata
escrezione di
colesterolo)
LXR attiva la
trascrizione del
gene ApoE
LXR favorisce la
formazione di HDL
OMEOSTASI della GLUTAMINA
•Lungs
•Muscles
Digiuno
•Digiuno
•Diabete
•Diete
ricche di
proteine
Nei tessuti periferici la deaminazione degli AA forma il glutamato che è convetito in
glutamin attraverso la glutamina-sintasi. In questi tessuti l’espressione dell’enzima è
attivata da glucorticoidi. la glutamina è il principale trasportatore si ammonio e viene
portata al fegato dove viene convertita in urea o degradata o utilizzata nella sintesi di AA.
Nel fegato l’attività della glutaminasi è sotto il controllo del cAMP
CICLO DELL’UREA
HNF4a regola il gene della OTC
(HNF4a KO -> iperammoniemia)
C/EBPa regola il gene della CPS-1
(C/EBPa KO -> iperammoniemia)
Il catabolismo degli AA porta alla formazione di NH3 il cui accumulo tossico è prevenuto dalla
trasformazione in urea nel fegato. 5 enzimi coordinano il ciclo dell’urea :
(CPS-1), carbamoyl phosphate synthetase, (OTC) ornithine transcarbamoylase
(ASS) argininosuccinate synthase, (ASL) argininosuccinate lyase e argininase.
La regolazione trascrizionale dell’espressione di questi enzimi è sotto il controllo di CEBP, PPAR e
HNF4