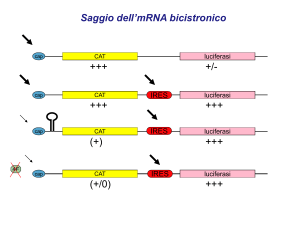
Sequenze IRES virali
IRES negli mRNA cellulari
Strutture secondarie delle IRES
Fattori trans-agenti delle IRES
Ruolo delle IRES
Meccanismi di traduzione
negli eucarioti
Regolazione della traduzione
•generale
•specifica
Initiation Factors
prokaryotes
Activity
eukaryotes
IF3
eIF-1
Fidelity of AUG codon recognition
IF1
eIF-1A
Facilitate Met-tRNAiMet binding to small
subunit
eIF-2
Ternary complex formation
eIF-2B (GEF)
GTP/GDP exchange during eIF-2 recycling
eIF-3 (12 subunits)
Ribosome antiassociation, binding to 40S
eIF-4F (4E, 4A, 4G)
mRNA binding to 40S, RNA helicase activity
IF2
eIF-4A
ATPase-dependent RNA helicase
eIF-4E
5' cap recognition
eIF-4G
Scaffold for of eIF-4E and -4A
eIF-4B
Stimulates helicase, binds with eIF-4F
eIF-4H
Similar to eIF4B
eIF-5
Release of eIF-2 and eIF-3, GTPase
eIF5B
Subunit joining
eIF-6
Ribosome subunit antiassociation
Via delle MAP chinasi
stress
Chinasi di eIF2a
Azione delle chinasi di eIF2a
Risposta UPR (Unfolded Protein Response)
Risposta UPR (Unfolded Protein Response)
Interferenza dei virus nella traduzione negli eucarioti
Meccanismi virali d'inibizione della fosforilazione di eIF2a
Regolazione di eIF2a
Fosforilazione su ser 51 in risposta a stress da
parte di 4 chinasi: PKR, GCN2, PERK, HRI
La fosforilazione impedisce il riciclo da parte di
eIF2B
La traduzione di alcuni mRNA è stimolata da bassi
livelli di eIF2a attivo
Molti virus hanno sistemi per impedire la
fosforilazione di eIF2a
eIF4F
Fosforilazione di 4E-BP
Regolazione di eIF4E
Regolazione eIF4E
Fosforilazione su ser 209 da parte di Mnk1 (e Mnk2) correlata
con attivazione
Mnk interagiscono con eIF4G e sono attivate dalla via delle
MAP chinasi
Doppio KO per Mnk1 e 2 è normale
eIF4E è inibito dall’interazione con 4E-BP (1, 2 e 3)
4E-BP sono inibite da fosforilazione dipendente dalla via di
mTOR
Alterazione della traduzione da parte dei virus
mTOR è un regolatore centrale
Struttura di mTOR
Inibizione della rapamicina
mTOR è un regolatore centrale
Complessi formati da mTOR
Bersagli di mTOR
mTOR regola l'inizio e l'allungamento
Translation and oncogenesis
• Hypertrophic nucleoli were a prominent feature of malignant cell
• Ribosome alterations are associated with malignancies
• Common cancer-related mutations are found in pathways feeding
into the translation machinery
• Translation initiation factors are frequently amplified or dysregulated
in tumours
Alterazione della traduzione e tumori
Role of ribosome in tumorigenesis
defective ribosome
QUANTITATIVE/QUALITATIVE
ALTERATIONOF TRANSLATION
Translation
initiation
factors and
cancers
Traduzione e cancro
Regolazione della traduzione
•generale
•specifica
Transcriptional control
Translational control
gene
mRNA
protein
•Slow
•Lasting
•Transcription-dependent
•Rapid
•Short-lived
•Transcription-independent
Indicazione di controllo traduzionale
northern
-Fe
mRNA
ferritina
western
+Fe
-Fe
ferritina
actina
mRNA
actina
+Fe
METODI PER STUDIARE IL CONTROLLO
TRADUZIONALE
• Confronto tra la variazione della quantità di
mRNA e quella del prodotto proteico
gel poliacrilammide 2D (DIGE)
western blot
• Analisi della stabilità delle proteine mediante
l'uso di inibitori della traduzione
• Analisi della quantità di mRNA sui polisomi
(distribuzione)
Polysome analysis
TOP mRNA translational control in cultured cells
Meccanismi di regolazione traduzionale
Meccanismi di
regolazione
traduzionale