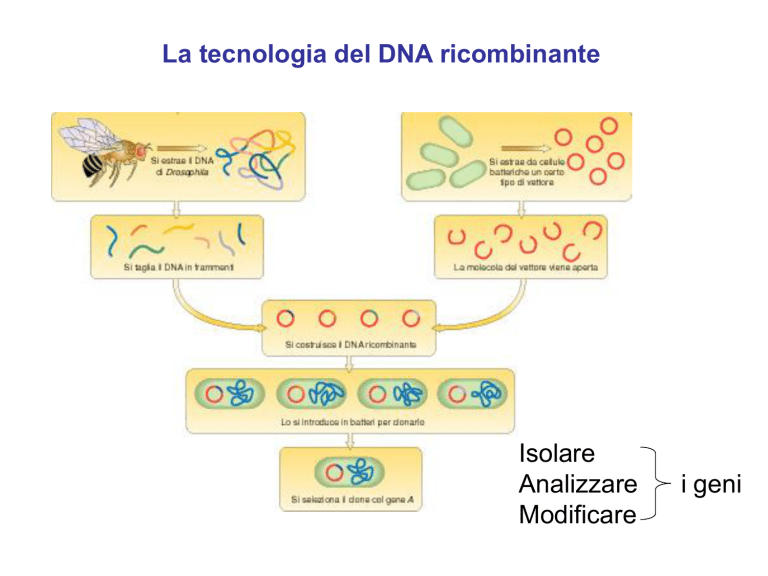
La tecnologia del DNA ricombinante
Isolare
Analizzare
Modificare
i geni
2 metodi per isolare geni di interesse
In vivo
(1973)
Clonaggio
genico
In vitro
(1983)
PCR
(polymerase
chain reaction)
Clonaggio genico. Fasi
• 1. Isolamento del frammento di DNA di interesse
(DNA genomico, di sintesi, cDNA)
• 2. Unione del frammento di DNA al vettore di
clonaggio
• 3. Trasferimento del vettore ricombinante alla
cellula ospite
• 4. Identificazione delle cellule che contengono la
molecola ricombinante
1. Isolamento del frammento di DNA di interesse.
Gli enzimi di restrizione
FENOMENO DELLA RESTRIZIONE
Alcuni
ceppi
batterici
sono
immuni
dall’infezione da parte di batteriofagi in quanto
contengono enzimi capaci di degradare il DNA
fagico (enzimi di restrizione) senza che venga
danneggiato il DNAdell’ospite batterico.
I batteri modificano il proprio DNA attraverso la
metilazione rendendolo così insensibile
all’azione degli enzimi di restrizione.
Digestione del DNA con gli enzimi di
restrizione
Estremità
adesive
complementari
Unione del
frammento di
DNA al vettore
Il vettore ricombinante
2. Vettori di clonaggio. Proprietà.
Molti vettori utilizzabili per il clonaggio sono stati ottenuti
modificando plasmidi presenti in natura
Plasmidi: “repliconi” : molecole capaci di replicazione
autonoma
Uno o più siti di
restrizione unici
Origine della
replicazione
POLYLINKER
Marcatore
selezionabile
UNO O PIU’ GENI CHE
CONFERISCONO LA RESISTENZA
AD ANTIBIOTICI
polylinker
sequenze uniche di riconoscimento
per diverse endonucleasi di restrizione
che permettono l’inserzione del DNA
eterologo
questo elemento facilita il lavoro di
clonaggio permettendo di utilizzare
l'enzima di restrizione più
conveniente.
Il batteriofago l
come vettore
COSMIDI
PLASMIDI RICOMBINANTI CHE UNISCONO
LE CARATTERISTICHE DEI PLASMIDI
TRADIZIONALI E QUELLE DEL
BATTERIOFAGO l
SONO PICCOLE MOLECOLE DI DNA
CIRCOLARE (5000-7000 bp) CHE
CONTENGONO:
UN ORIGINE DI REPLICAZIONE
UNO O PIU’ MARCATORI DI SELEZIONE
UN POLYLINKER
UN SITO COS
CONSENTONO IL CLONAGGIO DI
FRAMMENTI DI DNA MOLTO LUNGHI
(FINO A 45000 bp)
NELLA CELLULA INFETTATA IL
COSMIDE TORNA A MOLTIPLICARSI
COME UN NORMALE PLASMIDE
NON PUO’ FORMARE PARTICELLE
FAGICHE IN QUANTO NON POSSIEDE I
GENI STRUTTURALI DI l
I BATTERIOFAGI ED I COSMIDI
SONO UTILIZZATI COME VETTORI
PER LA COSTRUZIONE DI LIBRERIE
GENOMICHE
PROCEDURA di CLONAGGIO
ISOLAMENTO DEL GENE
Idrolisi con enzimi di restrizione
INSERZIONE DEL GENE IN UN VETTORE
PLASMIDICO
Ligasi
INTRODUZIONE DEL VETTORE
PLASMIDICO IN CELLULE VIVENTI PER
PROPAGARLO
Trasformazione
DNA LIGASI
DOPO AVER TAGLIATO (E ISOLATO) SPECIFICI FRAMMENTI
DI DNA, IL PASSO SUCCESSIVO DI UN CLONAGGIO
CONSISTE NEL “CUCIRLI” TRA LORO IN MODO
COVALENTE.
DNA LIGASI: PERMETTE L’UNIONE DI DUE MOLECOLE O
FRAMMENTI DI DNA
TALE ENZIMA LEGA COVALENTEMENTE L’ESTREMITA’ 5’
FOSFATO DI UNA BASE
CON
L’ ESTREMITA’ 3’ FOSFATO DELLA BASE SUCCESSIVA
QUESTA PROPRIETA’ E’ SFRUTTATA PER PRODURRE
MOLECOLE DI DNA RICOMBINANTE
DUE TRATTI DI DNA CHE IN NATURA NON SONO
ADIACENTI, VENGONO UNITI IN PROVETTA.
LA DNA LIGASI E’ UN ENZIMA CHE LEGA
COVALENTEMENTE DUE MOLECOLE DI DNA
OH
OH
P
5’
G
AATTC
CTTAA
G
5’
3’
P
3’
OH
G
AATTC
CTTAA
G
P
OH P
5’
3’
P
GAATTC
CTTAAG
P
OH
GAATTC
CTTAAG
OH
3’
5’
FOSFATASI ALCALINA
ENZIMA CHE RIMUOVE IL GRUPPO FOSFATO AL 5’
IN MOLTI CASI, PUR OTTIMIZZANDO LA REAZIONE DI LIGASI, NON SI
RIESCE AD EVITARE UNA ELEVATA FREQUENZA DI
RICIRCOLARIZZAZIONE DEL VETTORE
(QUANDO IL VETTORE E L’INSERTO SONO TAGLIATI CON UN SOLO
ENZIMA DI RESTRIZIONE).
P-5’
P-5’
AATTC
HO-3’
vettore
AATTC
vettore
3’OH HO-3’
inserto
TTAAG
-5’P
3’OH
TTAAG
-5’P
LA DEFOSFORILAZIONE DEL VETTORE CON LA FOSFATASI ALCALINA
IMPEDISCE LA RICIRCOLARIZZAZIONE DEL VETTORE
SENSIBILE ABBASSAMENTO DEL BACKGROUND POICHÉ IL VETTORE
DEFOSFORILATO NON PUÒ ESSERE LIGATO.
FILLING IN
NEL CORSO DEI CLONAGGI PUÒ CAPITARE DI DOVER
TRASFORMARE ESTREMITÀ COESIVE SPORGENTI AL 5’
IN ESTREMITÀ PIATTE, SINTETIZZANDO LE BASI
MANCANTI NEL FILAMENTO INCOMPLETO AL 5’
ESEMPIO ESTREMITÀ “STICKY” 5’ PROTUDING
(ES.ECORI)
LA TECNICA DEL “ FILLING IN” CONSISTE NEL
“RIEMPIRE”
l’ ESTREMITÀ SPORGENTE AL 5’ CON IL FRAMMENTO DI
KLENOW, UNA DNA POLIMERASI I MODIFICATA PRIVA
DELL’ATTIVITÀ 5’-3’ ESONUCLEASICA
dTTP
dATP
dGTP
3’OH
5’P
3’OH
G
C AATTC
5’P
5’P
3’OH
G TTAAG
C AATTC
3’OH
5’P
3. Introduzione del vettore nelle cellule ospiti
una volta avvenuta la ligasi è
necessario trasferire il plasmide
ricombinante nella cellula
batterica dove può replicarsi
per far questo è necessario
rendere le cellule ospiti
competenti cioè capaci di
acquisire DNA estraneo.
TRASFORMAZIONE BATTERICA
LE CELLULE POSSONO ESSERE RESE COMPETENTI
UTILIZZANDO VARI METODI FISICI E CHIMICI:
ELETTROPORAZIONE un breve impulso elettrico favorisce la
formazione di pori nella membrana cellulare e il dna esogeno in tali
condizioni è capace di entrare nella cellula
TRATTAMENTO CON CACL2 il cloruro di calcio, agisce da chelante e
“scherma” le cariche del dna. in queste condizioni il dna esogeno
può entrare attraverso microfratture a livello di membrana formatesi a
causa della bassa temperatura (0°c) a cui avviene il trattamento
4. Identificazione delle cellule che contengono la
molecola ricombinante.
IL PROBLEMA DELLA SELEZIONE
La trasformazione produce tre tipi di batteri, che bisogna
riuscire a discriminare, nel modo più rapido e semplice
possibile.
BATTERI VUOTI
BATTERI CONTENENTI IL SOLO VETTORE
BATTERI CONTENENTI IL VETTORE LIGATO ALL'INSERTO
l'uso di marcatori selezionabili, per esempio resistenze ad
antibiotici, permette facilmente di distinguere
batteri vuoti – sensibili all'antibiotico – da
batteri contenti il vettore, con o senza l'inserto, resistenti
all'antibiotico.
IL PROBLEMA DELLA SELEZIONE
PER SELEZIONARE LE COLONIE BATTERICHE CHE OSPITANO IL
PLASMIDE RICOMBINANTE SI UTILIZZANO VARIA STRATEGIE:
METODI DI INATTIVAZIONE GENICA
SI TRATTA DI UN GENE CHE VIENE INATTIVATO QUANDO UN TRATTO
DI DNA ESOGENO VIENE CLONATO NEL PLASMIDE.
ESEMPIO 1: DOPPIA RESISTENZA AGLI ANTIBIOTICI
ESEMPIO 2: SAGGIO CROMOGENICO
METODI DI SCREENING
ESEMPIO 1: COLONY HYBRIDISATION
ESEMPIO 2: ANALISI PER DIGESTIONE CON ENZIMI DI RESTRIZIONE
ESEMPIO 3: PCR
ESEMPIO 4: SEQUENZIAMENTO
IL PROBLEMA DELLA SELEZIONE
ESEMPIO 1 : DOPPIA RESISTENZA AGLI ANTIBIOTICI
ESISTONO PLASMIDI CHE PRESENTANO UNA DOPPIA
RESISTENZA AGLI ANTIBIOTICI, AD ESEMPIO ALL’AMPICILLINA
E ALLA TETRACICLINA.
UN TRATTO DEL DNA DEL GENE CHE CODIFICA PER LA
RESISTENZA ALLA TETRACICLINA COINCIDE CON IL
POLYLINKER
QUINDI IL VETTORE NON DIGERITO O RILIGATO SU SE STESSO,
CONSENTE AI BATTERI DI CRESCERE SIA IN PRESENZA DI
AMPICILLINA CHE DI TETRACICLINA.
SE INVECE UN TRATTO DI DNA VIENE INSERITO IN UN SITO DEL
POLYLINKER, IL GENE TCR
VIENE INATTIVATO.
IL PROBLEMA DELLA SELEZIONE
ESEMPIO 2 : SAGGIO CROMOGENICO
IN ALCUNI PLASMIDI IL POLYLINKER E’
SITUATO IN UN SEGMENTO DEL GENE LAC-Z DI
E.coli, CHE CODIFICA L’ENZIMA
-GALATTOSIDASI
IL POLYLINKER NON INTERROMPE LA FASE
DI LETTURA DEL GENE LAC-Z E
L’ESPRESSIONE DI QUESTO GENE
NELL’OSPITE PORTA ALLA SINTESI DI GALATTOSIDASI DOTATA DI ATTIVITA’
ENZIMATICA
L’ INSERIMENTO DI DNA ESOGENO, NEL
POLYLINKER DEL VETTORE DI CLONAGGIO,
INATTIVA LA SINTESI DI
-GALATTOSIDASI
(INTERRUZIONE DELLA FASE DI LETTURA )
SAGGIO CROMOGENICO
SUBSTRATO CROMOGENO DELL’ ENZIMA:
5bromo-4-cloro-3-indolil--D-galattoside X-GAL
INDUTTORE DELLA SINTESI DELL’ ENZIMA
isopropil--D-galattosidasi IPTG
BATTERI CRESCIUTI SU UNA PIASTRA DI
AGAR CONTENENTE X-GAL ED IPTG
PRODUCONO
-GALATTOSIDASI
LA DEMOLIZIONE DI X-GAL PRODUCE UN
COMPOSTO BLU INSOLUBILE CHE RENDE LE
COLONIE COLORATE
Utilizzo di LacZ per identificare i batteri
ricombinanti
COLONY HYBRIDISATION
Analisi DNA clonato. Separazione di frammenti di DNA
per elettroforesi su gel
Analisi di frammenti di DNA clonati in
plasmide per digestione ed
elettroforesi
La reazione a catena
della polimerasi
(PCR)
Amplificazione
esponenziale in vitro
di un frammento di
DNA a sequenza nota
Copie di DNA prodotte= 2n
Da una singola molecola
dopo 30 cicli >109 molecole
Analisi DNA
clonato.
Sequenziamento
(F. Sanger)
Chromosome walking
Clonaggio di
frammenti di DNA
parzialmente
sovrapposti



![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
![ESTRAZIONE DNA DI BANANA [modalità compatibilità]](http://s1.studylibit.com/store/data/004790261_1-44f24ac2746d75210371d06017fe0828-300x300.png)




![(Microsoft PowerPoint - PCR.ppt [modalit\340 compatibilit\340])](http://s1.studylibit.com/store/data/001402582_1-53c8daabdc15032b8943ee23f0a14a13-300x300.png)


