Farmacogenetica e Farmacogenomica
Definizione dei termini:
Genetica: studio dei singoli geni di
un organismo e delle caratteristiche
individuali ereditabili legate ad essi
Farmacogenetica
Genomica: determinazione e studio
dell’intera sequenza del DNA di un
organismo, identificazione e studio
del livello di espressione dei geni
per comprenderne le interazioni e
l’influenza sui meccanismi biologici e
fisiologici in situazioni normali e
patologiche
Farmacogenomica
Molto spesso i termini Farmacogenetica e
Farmacogenomica vengono utilizzati come sinonimi
Farmacogenetica e Farmacogenomica
Definizione dei termini:
Farmacogenetica: studio delle differenze ereditarie nella risposta
ad un farmaco, dovute a polimorfismi di particolari geni
studio del rapporto tra mutazioni nella sequenza del DNA e
differente efficacia e sicurezza di un farmaco
Farmacogenomica: applicazione delle tecniche usate nella genomica
per la determinazione e lo studio di più geni associati a malattie e
alla risposta ai farmaci per individuare sia nuovi geni bersaglio per
nuove terapie, sia profili genetici che influenzano l’effetto dei
farmaci
disegno di studi clinici che prevedano un arruolamento genotipospecifico (genostratificazione)
creazione, sviluppo e validazione di test diagnostici per la
genotipizzazione dei pazienti
Variabilità della risposta ai farmaci
Nonostante i farmaci vengano prescritti come validi per ogni paziente
affetto dalle condizioni cliniche verso cui i farmaci hanno mostrato
sperimentalmente di essere efficaci, c’è una notevole variabilità di
risposta alle molecole che vengono somministrate. Tale variabilità si può
manifestare in termini di mancata o solo parziale efficacia, oppure con la
comparsa di più o meno gravi effetti collaterali, per cui, mentre un
determinato principio attivo può essere benefico e innocuo per alcuni
soggetti, per altri può rivelarsi non solo inutile ma addirittura letale.
Nel corso del 1994, negli Stati Uniti, quasi due milioni di pazienti sono
stati ospedalizzati a causa di reazioni avverse a farmaci (ADR =
Adverse Drug Reaction), pur se appropriatamente prescritti e
somministrati, con più di 100.000 pazienti deceduti.
Variabilità della risposta ai farmaci
Percentuale di efficacia nelle risposte dei pazienti ai
principali farmaci in alcune aree terapeutiche
Area terapeutica
Alzheimer
Analgesici (inibitori Cox-2)
Asma
Aritmia cardiaca
Artrite reumatoide
Depressione
Diabete
HCV
Incontinenza
Oncologia
Osteoporosi
Schizofrenia
Efficacia (%)
30
80
60
60
50
62
57
47
40
25
48
60
Variabilità della risposta ai farmaci
Possibili cause dell’inefficacia o della reazione avversa ai
farmaci
Cause non genetiche
patogenesi e severità della
malattia
età del paziente
funzione epatica e renale
co-somministrazione di altri
farmaci
stile di vita e abitudini
alimentari
Cause genetiche
polimorfismo di geni
codificanti proteine che
influenzano il metabolismo,
la biodisponibilità o
l’effetto dei farmaci
si calcola che circa il 2550% dei casi di terapia
inefficace o ADR siano
dovuti a cause genetiche
Polimorfismo genico e SNPs
Variazione della sequenza all’interno di un gene con successiva
creazione di uno o più alleli presenti con frequenza di almeno l’1%
all’interno di una popolazione
GATTTAGATCGCGATAGAG
GATTTAGATCTCGATAGAG
Nel 90% dei casi si tratta di polimorfismi che riguardano un singolo
nucleotide (SNP = Single Nucleotide Polimorphism) all’interno della
sequenza codificante la proteina o della regione di controllo
dell’espressione genica, il rimanente 10% è rappresentato da inserzioni,
delezioni, ripetizioni in tandem e microsatelliti
I polimorfismi sono alla base della variabilità genica tra un individuo ed
un altro e possono influenzare l’attività, l’espressione, la degradazione e
le caratteristiche delle proteine interessate
La scoperta degli SNPs e’ stata possibile grazie al completamento del
Progetto Genoma Umano avvenuto nel 2001
Polimorfismo genico e Farmacogenetica
E’ stato calcolato che nel genoma umano sia presente uno
SNP ogni 1.000 paia di basi circa, per un totale di almeno 1,42
milioni di SNPs complessivi nell’intero genoma, almeno 60.000
dei quali situato in regioni codificanti
I geni fondamentali per la produzione di proteine nell’uomo
sono 30-40 mila e tra questi da 3.000 a 10.000 sono importanti
per l’attività dei farmaci
Il numero totale di patologie per le quali è stata identificata
una mutazione genetica determinante è circa 1.500
Importanza della farmacogenomica per lo studio di malattie poligeniche
(ad es. cardiovascolari ed oncologiche) e per l’individuazione dei
farmaci più efficaci per la cura dei pazienti in base ai profili genici
individuali
Banche dati delle SNPs
Identificazione di nuovi SNPs nella popolazione
Raccolta ed elaborazione dei dati tramite la bioinformatica
Creazione di banche dati di pubblico dominio
Nascita della biologia in silicio per identificare polimorfismi in regioni
codificanti mediante algoritmi al computer che allineano EST (Expressed
Sequences Tag) da sequenze sovrapposte
Databases
SNP
CGAP-GAI
LEELAB
dbSNP
JSNP
Compilation
HOWDY
HGVBase
GeneSNP
PharmGKB
Website
Data source
http://lpgw s.nci.nih.gov:82/perl/snpbr
EST
http://www .bioinformatics.ucla.edu/snp/
http://www .ncbi.nlm.nih.gov/SNP/
http://snp.ims.u-tokyo.ac.jp/
EST
Genomic clones
Genomic clones
http://gdb.jst.go.jp/HOWDY/
http://hgvbase.cgb.ki.se/
http://www .genome.utah.edu/genesnps/
http://pharmgkb.org
JSNP/dbSNP
Whitehead-MIT/ CGAP-GAI/literature
dbSNP/egSNP/UUGC/UWGC
Direct submission w ith links to target
genes
Effetti farmacocinetici e farmacodinamici di mutazioni
Polimorfismi di geni codificanti
proteine coinvolte in:
metabolismo dei farmaci (es.
attivazione di prodrugs o
inattivazione di molecole attive)
assorbimento, trasporto ed
eliminazione dei farmaci (es.
proteine trasportatrici di membrana)
EFFETTI FARMACOCINETICI
Polimorfismi di geni codificanti
proteine
recettori dei farmaci
bersaglio dei farmaci
EFFETTI FARMACODINAMICI
Esempi di mutazioni che influenzano l’attività di enzimi
Esempi di polimorfismo di enzimi metabolizzanti farmaci
OSSIDAZIONE
CONIUGAZIONE
ADH, alcohol deidrogenasi; ALDH, aldeide deidrogenasi; CYP, citochrome P450; DPD,
diidropirimidina deidrogenasi; NQO1, ADPH:chinone ossidoriduttasi o DT diaforasi; COMT, catecol
O-metiltransferasi; GST, glutatione S-transferasi; HMT, istamina metiltransferasi; NAT, Nacetiltransferasi; STs, sulfotransferasi; TPMT, tiopurina metiltransferasi; UGTs, uridina 5’-trifosfato
glucuronosiltransferasi
Esempi di polimorfismo che influenzano gli effetti dei farmaci
Protein or gene
Medications
Examples of altered drug effects
Drug-metabolizing enzymes
CYP2C19
Non-steroidal anti-inflammatories, warfarin,
tolbutamide, phenytoin
Omeprazole, mephenytoin
CYP2D6
Antidepressants, codeine, -blockers
Peptic ulcer response to omeprazole
Increased antidepressant toxicity, decreased
codeine analgesia
CYP3A4/3A5/3A7
Cyclosporin, tacrolimus, calcium channel
blockers, midazolam, terfenadine, etoposide,
lovastatin, tamoxifen, steroids
Decreased efficacy of tacrolimus in organ
transplantation
Dihydropyrimidine dehydrogenase
DPD
5-Fluorouracil
Increased neurotoxicity
Glutathione transferases GSTM1,
M3, T1
Several anticancer agents
Thiopurine methyltransferase
TPMT
Mercaptopurine, thioguanine, azathioprine
uridine diphosphate
glucuronosyltransferase UGT1A1
Irinotecan
Increased gastrointestinal toxicity
Digoxin, HIV protease inhibitors, natural
product anticancer drugs
2-agonists (for example, albuterol,
terbutaline)
Decreased CD4 response in HIV-infected
patients, decreased digoxin bioavailability
CYP2C9
Increased anticoagulant effects of warfarin
Increased response in breast cancer, more
toxicity and poorer outcome in acute myeloid
leukaemia
Increased haematopoietic toxicity, increased
risk of secondary cancers
Drug transporters and targets
ABCB1 (MDR-1, P-glycoprotein)
2-adrenergic receptor
1-adrenergic receptor
1-antagonists
Thymidylate Synthase (TS)
5-Fluorouracil
ALOX5
Leukotriene receptor antagonists
Serotonin transporter (5-HTT)
Antidepressants (for example, fluoxetine)
Decreased bronchodilation
Decreased cardiovascular response to 1antagonists
Decreased antitumoral effect
Lower changes in FEV1 (forced expiratory
volume)
Decreased clozapine effects, antidepressant
response
Variabilità individuale nell’efficacia terapeutica dei farmaci
Individuo normale
Efficacia ridotta
collaterale
Effetto
Effetto
collaterale
terapeutico
terapeutico
Dose
Accettabile
Dose
Non accettabile
Variabilità individuale negli effetti collaterali dei farmaci
Individuo normale
Aumento di effetti collaterali
collaterale
terapeutico
Effetto
Effetto
collaterale
terapeutico
Dose
Accettabile
Dose
Non accettabile
Determinanti poligenici dell’effetto dei farmaci
Polimorfismo enzima
metabolizzante
Polimorfismo proteina
bersaglio
Omozigote wt
Eterozigote
Omozigote mut
Storia della Farmacogenetica
Pharmacogenetic phenotype
Sulfonal porphyria
Suxamethonium hypersensitivity
Primaquine hypersensitivity; favism
Long QT syndrome
Isoniazid slow/fast acetylation
Malignant hyperthermia
Fructose intolerance
Vasopressin insensitivity
Alcohol susceptibility
Debrisoquin hypersensitivity
Retinoic acid resistance
6-Mercaptopurin toxicity
Mephenytoin resistance
Insulin insensitivity
Described
ca. 1890
1957–1960
1958
1957–1960
1959–1960
1960–1962
1963
1969
1969
1977
1970
1980
1984
1988
Underlying gene/mutation
Porphobilinogen deaminase?
Oseudocholinesterase
Glucose 6 phosphate dehydrogenase
Herg etc.
N-Acetyltranferase
Ryanodine receptor
Aldoalase B
Vasopressin receptor 2
Aldehyde dehydrogenase
CYP2D6
PML-RARA fusion gene
Thiopurine methyltransferase
CYP2C19
Insulin receptor
Identified
1985
1990–1992
1988
1991–1997
1989–1993
1991–1997
1988–1995
1992
1988
1988–1993
1991–1993
1995
1993–1994
1988–1993
Pre-genomica e Post-genomica
Applicazioni della Farmacogenetica
Identificazione di
pazienti che non
rispondono (nonresponders) o a
rischio di ADR
(toxic
responders) per
un determinato
farmaco: verranno
trattati solo i
pazienti risultati
responders, gli
altri riceveranno
una terapia
alternativa o dosi
ridotte del farmaco
“Il farmaco giusto alla dose giusta per il paziente giusto”
Le promesse della Farmacogenomica
Farmaci più potenti: scoperta di nuovi geni bersaglio associati alle malattie
farmaci più specifici e meno tossici
Prescrizione di farmaci migliori e più sicuri: profilo genetico del paziente
giusto farmaco ad ogni paziente anziché metodo “tentativo ed errore” (es
cura dell’ipertensione)
Determinazione più accurata dei dosaggi: profilo genetico del paziente
calcolo del dosaggio in base alle caratteristiche genetiche e metaboliche
individuali anziché peso ed età
Screening preventivo e predittivo delle malattie: determinazione del
codice genetico del paziente possibilità di prevedere l’insorgenza di malattie
genetiche ed adottare cure preventive o adeguati stili di vita
Migliori vaccini: vaccini di DNA o RNA attivazione sistema immunitario
senza rischio di infezione, economici, stabili, più ceppi nello stesso vaccino
Maggior efficienza nella scoperta di nuovi farmaci: caratterizzazione
bersagli genomici trials specifici per particolari gruppi genetici
Diminuzione dei costi delle cure: diminuzione reazioni avverse o farmaci
inefficaci, diminuzione trials falliti diminuzione complessiva dei costi
Ostacoli allo sviluppo della Farmacogenomica
Difficoltà nell’individuare i geni le cui variazioni influenzano l’effetto dei
farmaci: spesso numerosi geni sono coinvolti nel metabolismo di un farmaco
e gli effetti del loro polimorfismo possono sommarsi o annullarsi
reciprocamente
Limitata scelta di farmaci alternativi: per alcune patologie sono disponibili
solo pochi farmaci con il rischio di non avere alternative terapeutiche per i
pazienti risultati non trattabili con i farmaci convenzionali
Scarso interesse delle società farmaceutiche nello sviluppo di farmaci
alternativi: potrebbe non essere economicamente vantaggiosa la ricerca e la
commercializzazione di farmaci alternativi per pazienti con polimorfismi rari
Necessità di corsi di formazione per il personale medico: per
l’applicazione delle tecniche di farmacogenomica e la corretta interpretazione
dei risultati sono necessarie specifiche competenze soprattutto in campo
genetico
Problemi di carattere etico e di difesa della privacy: discriminazioni,
problemi psicologici, conseguenze in campo lavorativo ed assicurativo
Test genetici: scopi, benefici e rischi
Tecniche utilizzate in Farmacogenetica
Technology
Typical genetic markers
Characteristics
DNA sequencing
Sequences, mutations, SNPs,
VNTRs, deletions, insertions
Broad utility for characterization of genetic
mutations; Not quantitative
Hybridization based
correlated methods:
target amplification
Sequences, mutations, SNPs,
mRNA
Sequential copying of target sequence followed by
signal generation event to presence of initial target.
Highly sensitive and specific. Can be used for
quantification. PCR is most widely used method.
Hybridization based
methods: signal
amplification
Sequences, mutations, SNPs,
mRNA
Signal amplification event triggered by an initial
binding event. Particularly useful for quantification.
Sensitivity less than target amplification. Amenable
to high throughput screening applications.
Microarray
SNPs, SBH analysis, mRNA
expression level profiling
Amenable to high levels of multiplexing.
Quantitative and qualitative analysis. Useful for
screening broad patterns of sequences. Less
well established in diagnostics than
sequencing or target amplification
Restriction and
conformational analysis
Polymorphism detection and
confirmation
Primarily used for mutation detection and analysis.
Widely used examples are RFLP and SSCP.
Single base primer
extension
SNP detection and confirmation
Adaptable to generic formats. Amenable to high
throughput screening
Abbreviations: RFLP, restriction fragment length polymorphism; SBH, sequencing by hybridization; SNP, single
nucleotide polymorphism; SSCP, single stranded conformation polymorphism; VNTR, variable number tandem
repeat.
Individuazione di pazienti a rischio ADR
Profilo SNP dei soggetti del
clinical trial
Nessun ADR durante i
clinical trials
Manifestazione di ADR
durante i clinical trials
Profilo SNP dei pazienti
Basso rischio ADR
Alto rischio ADR
SNP-LD: SNP Linkage Disequilibrium
Approcci per l’analisi delle variazioni farmacogenetiche
Approach
Candidate polymorphism
analysis
Pathway approach
Genomics or proteomics
(microarray technology)
Advantages
Limitations
• Quick and relatively inexpensive
• Uses knowledge regarding drug
pharmacodynamics and pharmacokinetics
• Hypothesis driven
• Only helpful in the clinical setting if the polymorphic
genotype corresponds to a distinct phenotype (high
sensitivity/specificity)
• Assumes extensive previous knowledge regarding
what gene is likely to be important — such as one
that determines the excreted proportion of a drug
• Very limited approach
• Accounts for associations between proteins
in a metabolic pathway
• Uses knowledge regarding drug metabolism
and other meaningful biological
associations (such as regulatory function
and homeostasis)
• More likely to explain inter-individual
variation in drug response
• Hypothesis driven
• Requires a solid biochemical understanding —
pharmacological or biological — of the respective
pathway
• Need to understand how the drug is metabolized and
its mechanisms of action
• Data analysis is more complex than evaluation
• Requires large study sizes
• Does not require a hypothesis
• Provides a complete gene- or proteinexpression profile (tumour or individual)
• Provides information on associations that
have not previously been suspected
• Provides a large amount of data
• Probably useful for prediction of tumour
response
• Does not use information on known biological or
pharmacological associations
• Optimal data management and data analysis
techniques are not well defined at this point
• Expensive (expression arrays), not fully developed
(proteomics) or not currently practical in a clinical
setting
• Not clear how useful for prediction of toxicity
Identificazione di un gene che influenza la risposta poligenica ad
un farmaco
Analisi di un
gene candidato
Approcci di tipo genomico
Esempio di progettazione di test diagnostico con microarray
Sovraespresso
Sottoespresso
La farmacogenetica e le terapie antitumorali
Il dosaggio preciso del farmaco è particolarmente importante
per gli antitumorali a causa del loro ristretto indice terapeutico
Aumentando la dose di farmaco aumenta
l’efficacia ma anche la probabilità di avere
effetti tossici, è quindi importante valutare
la dose che garantisce il massimo di
efficacia con la minima tossicità: con la
farmacogenetica si possono individuare i
pazienti a rischio ADR e quelli che non
rispondono al trattamento
Polimorfismi ed effetto sulle terapie antitumorali
Drugs
Clinical
phenotype
6-Mercaptopurine
Toxicity
Pharmacological
phenotype
Decreased TPMT
activity (Increased
TPMT degradation)
Genes
TPMT
(Drug-metabolizing
enzyme)
DPD
(Drug-metabolizing
enzyme)
Mutated alleles
TPMT*2, TPMT*3A,
TPMT*3Ca
Toxicity
Decreased DPD
activity
Downstaging
Response
Toxicity
Increased TS
expression
TS
(Drug target)
TSER*3
Irinotecan
Toxicity
Decreased UGT1A1
expression and
activity
UGT1A1
(Drug-metabolizing
enzyme)
UGT1A1*28
CMF regimen
(methotrexate)
Raltitrexed
Toxicity
Decreased MTHFR
activity
MTHFR
(Drug target)
C677T
Cyclophosphamide,
Oxalyplatin
Toxicity
Response
Decreased GSTP1
activity
GSTP1
(Drug-metabolizing
enzyme)
GSTP1 I105V
5-Fluorouracil
DPD*2A
CMF, cyclophosphamide, methotrexate, 5-fluorouracil; DPD, dihydropyrimidine dehydrogenase; GSTP1, glutathione Stransferase P1; MTHFR, 5,10-methylenetetrahydrofolate reductase; TPMT, thiopurine methyltransferase; TS, thymidylate synthase;
UGT1A1, uridine diphosphate glucuronosyltransferase.
Clinical phenotypes include response, downstaging, survival and toxicity. Pharmacological phenotypes include in vivo and ex vivo
indexes like blood cell enzyme activity, plasma metabolic ratios and use of phenotypic probes.
a TPMT*2,
TPMT*3A, TPMT*3C are associated with up to 95% of the TPMT deficiency.
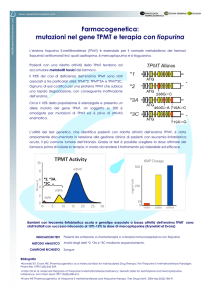
Tiopurina metiltransferasi e terapia con mercaptopurina
Test genetici o enzimatici su TPMT sono usati per determinare la dose corretta di tiopurine
Thiopurine methyltransferase
Hypoxanthine phosphoribosyl transferase
Thioguanine nucleotides
Diidropirimidina deidrogenasi, timidilato sintasi e terapia con 5-fluorouracile
(TS inhibitor)
Uridindifosfato glucuronosiltransferasi e terapia con irinotecan
Nel 2005 l’FDA ha approvato l’uso del test clinico per l’allele UGT1A1*28
(inactive)
(carboxylesterase)
UGT1A1
Wild-type
UGT1A1*28
UGT1A1 = uridine diphosphate
glucuronosyltransferase 1A1
Metabolismo dei folati e vie collegate, terapia con metotrexate
5,10-methylenetetrahydrofolate reductase
dihydrofolate reductase
Altri polimorfismi con effetto sulle terapie antitumorali
GSTP1 (glutatione S-transferasi P1)
Reagisce con vari farmaci antitumorali (es. ciclofosfamide, oxaliplatino) inattivandoli
Polimorfismo GSTP1 I105V in circa 33% della popolazione caucasica
GSTP1 I105V ha attività ridotta rispetto al wt: gli omozigoti presentano maggiore
rischio di tossicità e aumentata risposta alla terapia rispetto ai wt, gli eterozigoti
presentano tossicità e risposta intermedia
Enzimi citocromo P450 dipendenti
Reagisce con vari farmaci antitumorali attivandoli o inattivandoli
Vari tipi di polimorfismo per gli enzimi di questa classe
MDR1 (multidrug resistance-associated protein 1)
o Glicoproteina-P
E’ coinvolta nel trasporto transmembrana e nell’eliminazione di metaboliti endogeni e
farmaci antitumorali
Quando è overespressa può diminuire la risposta ai farmaci aumentandone la loro
escrezione
Trastuzumab (Herceptin) ed oncogene ERBB2 (o HER2)
ERBB2 = erythroblastic leukemia viral oncogene homolog 2
(o NEU2 = neuro/glioblastoma derived oncogene homolog)
Appartiene alla famiglia dei recettori per l’EGF (epidermal growth factor) ed è presente
sulla membrana cellulare, quando viene attivato stimola la crescita cellulare
ERBB2 è associato ad alcuni tumori alla mammella: errori di replicazione del DNA
portano alla formazione di copie multiple del gene (amplificazione genica) con aumento
del numero di recettori sulla membrana e conseguente replicazione cellulare
incontrollata.
In circa il 25% dei tumori alla mammella si riscontra overespressione del gene ERBB2
con basso tempo di sopravvivenza libera da malattia e resistenza ad alcuni farmaci
antitumorali.
Nel 1998 l’FDA ha approvato l’utilizzo, per il trattamento del tumore metastatico alla
mammella, di un anticorpo umanizzato ricombinante rivolto verso la proteina ERBB2: il
Trastuzumab (o Herceptin della Genentech)
Trastuzumab (Herceptin) ed oncogene ERBB2 (o HER2)
ERBB2 è un ottimo bersaglio per la terapia con Trastuzumab:
Livelli strettamente correlati alla patogenesi e alla prognosi del tumore
Livelli nelle cellule tumorali molto più elevati che nelle cellule sane (ridotta
tossicità ed elevata specificità del trattamento)
Espresso in un’altissima percentuale di cellule tumorali (elevata efficacia della
terapia)
Espresso sia nel tumore primario che nelle metastasi (trattamento efficace in tutte
le sedi tumorali)
Importanza dell’accurata determinazione dei livelli di espressione di ERBB2
Informazioni sulla
prognosi ed
evoluzione del tumore
Individuazione dei
pazienti trattabili con
Trastuzumab
Metodi d’analisi dell’espressione di ERBB2
Method
Advantages
Western blot
Widely available;
relatively inexpensive
PCR
Rapid; specific;
(polymerase chain
sensitive; small amount
reaction)
of starting material
IHC
(immunohistochemistry:
Widely available;
staining paraffinrelatively inexpensive
embedded tissue with
an ERBB2-specific Ab)
Specific; quantitative;
FISH
strong correlation with
(fluorescence in situ
response to
hybridization)
trastuzumab
ECD ELISA
(extracellular domain
ELISA)
Serum easily obtained
Disadvantages
Semiquantitative; Ab
variability; tumor extract
is required
Semiquantitative
Clinical use
Not in clinical use
Not in clinical use
Semiquantitative (scale
from 0 to 3+); Ab
FDA-approved; most
variability; subjective
frequently used clinically
interpretation
Expensive; requires
specialized equipment
not widely available
ECD levels do not
always correlate with
tumor load
FDA-approved; valuable
for confirmation of
HER-2 status if IHC
score is 2+
FDA-approved to
monitor response to
chemotherapy;
multicenter prospective
study ongoing in
patients on trastuzumab
Terapia con Trastuzumab, associazioni e nuovi anti-ERBB2
Il Trastuzumab viene utilizzato quando ERBB2 è overespresso (IHC 3+)
I trials clinici indicano una risposta del 12-26% dopo trattamento con Trastuzumab in
monoterapia, spesso i pazienti che inizialmente rispondono alla terapia manifestano però
una progressione entro 1 anno
Studi di fase II per l’associazione con altri antitumorali (es. doxorubicina+ciclofosfamide,
docetaxel, carboplatino, paclitaxel, vinorelbina, gemcitabina) indicano il 24-80% di risposte.
Risultati promettenti per l’associazione trastuzumab-inibitori specifici (es. inibitore della
tirosina chinasi anti-EGFR o inibitore della farnesil transferasi)
Progettazione di nuovi farmaci anti-ERBB2 (es. Trastuzumab legato ad una tossina, inibitori
specifici, vaccini a DNA)
Farmaco
Trastuzumab - DM1
Pertuzumab (2C4)
Canertinib (CI-1033)
Lapatinib
(GW572016)
2B1
AutoVac
Tipo di farmaco
Coniugati MAb-tossina
MAb
TKI (inibitore di tirosin
chinasi)
TKI (inibitore di tirosin
chinasi)
Ab bispecifico verso
HER-2 e Fc RIII
Vaccino a DNA
Fase di sviluppo
Commercializzato
III
Ditta
Genentech
Genentech
II
Pfizer
III
Glaxo Smithkline
II
Chiron
II
Pharmexa
K-RAS Mutation Assay: terapia con Cetuximab o
Panitumumab in tumori iper-esprimenti EGFR
EGFR (HER1 o ErbB1) = recettore della superficie cellulare stimolato da fattori di
crescita e coinvolto nella crescita, replicazione e differenziazione cellulare.
Alcuni tipi di cancro – in particolare polmone, prostata, colon-retto, ovaio, distretto
cervico-facciale - iper-esprimono il recettore EGFR e questa iper-espressione è
correlata alla metastatizzazione del tumore e ad una prognosi negativa.
Per il trattamento di questi tumori sono attualmente utilizzati anticorpi monoclonali (ad
es. cetuximab e panitumumab) che riconoscono la parte di recettore esposta verso
l’esterno della cellula. Questi farmaci sono però molto costosi e causano importanti effetti
collaterali.
Studi recenti hanno inoltre dimostrato che i tumori colon-rettali iper-esprimenti il recettore
EGFR che presentano mutazioni del gene K-RAS (circa il 40% del totale) sono resistenti
al trattamento con anticorpi anti-EGFR. La proteina espressa da K-RAS è coinvolta nel
meccanismo di trasduzione dei segnali di crescita cellulari mediato dal recettore EGFR: la
mutazione rende K-RAS costitutivamente attivata e quindi in grado di stimolare la
proliferazione cellulare indipendentemente dall’attività di inibizione degli anticorpi antiEGFR.
Recentemente è stato messo a punto il test “K-RAS Mutation Assay”, che consente di
rilevare, nei pazienti affetti da tumore colon-rettale, la presenza di mutazioni del recettore
EGFR e prevedere quindi quali pazienti potranno trarre beneficio dal trattamento con gli
anticorpi.
Esempio di applicazione nello screening dei pazienti per
l’individuazione di terapie mirate: lo studio ITACA
International TAilored Chemotherapy Adjuvant (ITACA) trial su pazienti
affetti da NSCLC (carcinoma polmonare non a piccole cellule) stadio II-IIIA
Osservazioni sperimentali:
L’Excision Repair Cross-Complementation 1 (ERCC1) è un gene coinvolto nella riparazione dei danni al DNA
causati dal cisplatino o composti simili, è overespresso in molte cellule tumorali, il suo livello di espressione nel
tumore di pazienti affetti da NSCLC è correlato con la risposta al trattamento con cisplatino più gemcitabina: i
pazienti con bassi livelli di mRNA di ERCC1 rispondono meglio alla terapia ed hanno sopravvivenza mediana
maggiore rispetto ai pazienti con alti livelli di mRNA.
Il livello relativo di mRNA di ERCC1 espresso nel tessuto tumorale può essere inversamente associato con la
risposta e la sopravvivenza, come un indicatore indipendente dell’efficacia del cisplatino.
L’espressione della Timidilato Sintasi (TS), enzima bersaglio del 5-fluorouracile, è correlata alla risposta a
questo farmaco: livelli elevati di TS in tumori gastrointestinali, mammari e in NSCLC sono associati a prognosi più
infauste.
I poliglutamil derivati del Pemetrexed inibiscono la TS, quindi pazienti che esprimono bassi livelli di TS dovrebbero
rispondere meglio alla terapia con questo farmaco.
Disegno dello studio:
•Pazienti affetti da NSCLC e sottoposti a resezione completa
•Analisi del livello di espressione dell'ERCC-1 e della TS tramite qRT-PCR su di un frammento del tessuto tumorale
asportato
•Randomizzazione in un gruppo di controllo, trattato con terapie adiuvanti standard, o in un gruppo sperimentale
trattato con terapia adiuvante personalizzata secondo l’appartenenza del paziente ad uno dei 4 diversi profili
genetici
Confronto dell’efficacia della terapia personalizzata rispetto alla standard
CONCLUSIONI
Come la farmacogenomica influenzerà…
…la ricerca per lo sviluppo
di nuovi farmaci
…il rapporto del paziente
con medico e farmacista