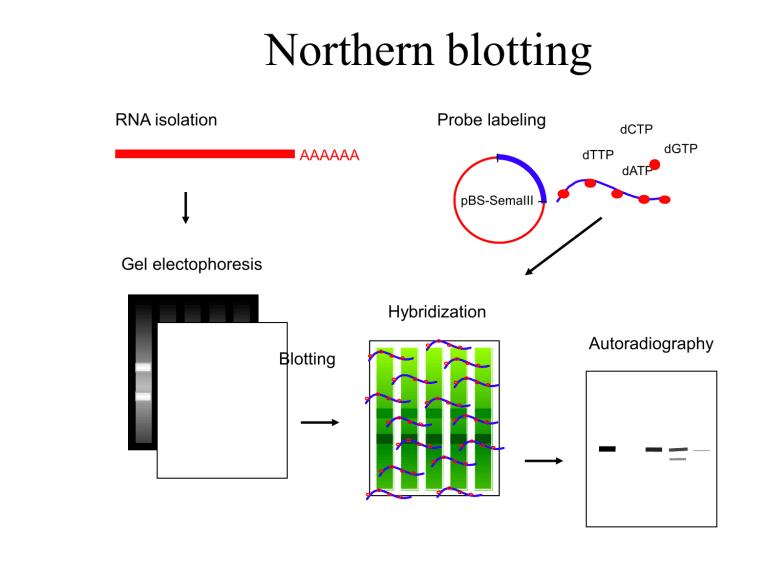
Northern blotting
RNA isolation
Probe labeling
AAAAAA
dCTP
dGTP
dTTP
dATP
pBS-SemaIII
Gel electophoresis
Hybridization
Blotting
Autoradiography
reverse Northern blotting
Northern
reverse Northern
specific probes
*
*
labeled target
cDNA arrays (continued)
• macro-arrays
– low-density
– filter-supported
– radioactive probes
(duplicates)
– low sensitivity
– only cDNA clones
spotted
– cheap
cDNA arrays (continued)
• micro-arrays
– high-density
– glass-supported
– fluorescent probes
(single slide)
– high sensitivity
– ability to spot
oligonucleotides
– very expensive
Tessuto della prostata,
normale
Tessuto della prostata,
tumorale
In questo esempio i cDNA
sono stati posizionati su di un
vetrino, simile ai normali
vetrini usati per l’istologia.
I microarrays
Un microarray è un supporto solido
sul quale sono stati posizionati
diverse migliaia di cDNA in spot
separati. Ciascuno spot rappresenta
un gene, in quanto contiene
numerose copie di un cDNA
corrispondente a tale gene.
Microarray technology
Probes
Microarray synthetized by photolithography or EST/oligo “linking”
http://www.ym.edu.tw/excellence/HBP/HBP_CP4/procedure.htm
Tessuto della prostata,
normale
Tessuto della prostata,
tumorale
utilizzo dei microarrays
si confrontano i profili di
espressione genica di due campioni
differenti.
E’ necessario estrarre le molecole di
mRNA dai due campioni.
Arrays
Probe DNAs are “spotted”
onto a glass slide
Each spot corresponds to a
particular gene e.g. β-actin
Each array will have
thousands of spots (typically
3000 to 30000)
reference
test
RNA is extracted from tissues
Targets
mRNA
cDNA
or cells
RNA is copied to DNA
labelled
DNA is fluorescently labelled
pooled
Samples are pooled
Hybridisation
Labelled target DNA
hybridised to array
Fluorescence of each spot
indicates how much of
particular RNA was present in
both samples
Hybridised
Array
Data Processing
Further
analysis
Scanner
Images
Normalised
Ratios
Normalisation
Spot-finding
Ratios
Cy3
Cy5
test
reference
Cy3
Cy5
Spot-finding (1)
Software identifies grid of
spots and maps individual spot
locations
Estrazione
dell’mRNA dai
2 campioni di
cellule che si
vogliono
confrontare
Conversione in
cDNA
Marcatura con
2 fluorocromi
diversi
Confronto dei profili di espressione genica in
due campioni cellulari diversi
(1)
(2)
(3)
(6)
Immagine a colori
raffigurante il
microarray
Eccitazione della
fluorescenza
tramite laser
Riconoscimento (4)
tra i cDNA
provenienti dai 2 campioni e
quelli già presenti sul microarray
(5)
STUDIO DEI PROMOTORI: siti di legame dei fattori
di trascrizione
EMSA
Trans Factor Methods: EMSA
Trans Factor Methods: EMSA
Electromobility
“Shift”
Trans Factor Methods: Consensus Sequence Capture
Il saggio EMSA identifica
interazioni tra DNA e proteine
anticorpo
Il saggio EMSA identifica
interazioni tra DNA e proteine
• Competizione con stesso sito, sito analogo, sito mutato
• Identificazione dei fattori coinvolti tramite “supershift” con anticorpi
Lodish
Figure 10-7
Il footprint con DNasi I permette
di localizzare il sito di interazione
tra proteina e DNA
Lodish
Figure 10-6







![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)




