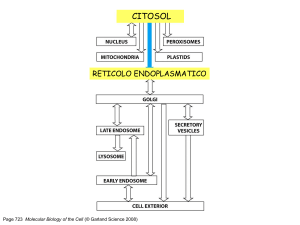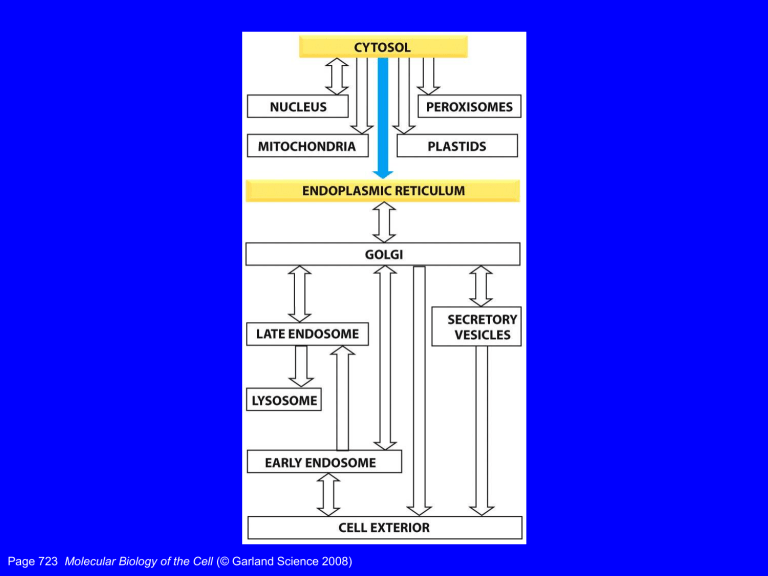
Page 723 Molecular Biology of the Cell (© Garland Science 2008)
IL POOL DI RIBOSOMI E’ UNICO
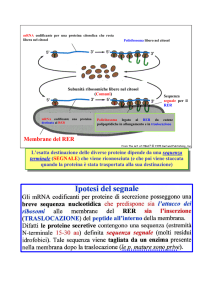
LE PROTEINE ATTRAVERSANO LE MEMBRANE CON DUE
DIFFERENTI MODALITA’
TRASLOCAZIONE
CO-TRADUZIONALE
TRASLOCAZIONE
POST-TRADUZIONALE
Signal sequence is both necessary and sufficient for
import into ER
Necessary
Sufficient
Translocation into the ER
Sec61 is a hetero-trimeric
complex composed of a, b, g
subunits related to SecYEG
SRP is a ribonucleoprotein
complex composed of 7S
RNA and numerous proteins
binding of signal sequence
is modulated by NAC
a post-translational translocation pathway
that makes use of Sec61 also exists;
preproteins are maintained in a translocationcompetent form by Hsp70/Hsp40
SRP pathway is cotranslational; SRP mediates
arrest of elongation until it
docks with SRP receptor;
translocation then proceeds
through Sec61
SRP is the major pathway
used for import into ER
10-7
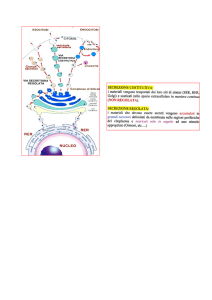
NUMEROSI ELEMENTI SONO COINVOLTI NELLA
TRASLOCAZIONE CO-TRADUZIONALE
1. PEPTIDE SEGNALE
2. PARTICELLA SRP
3. RECETTORE DI SRP
4. TRASLOCONE
5. PEPTIDASI DEL SEGNALE
IL PEPTIDE SEGNALE E’ UNA SEQUENZA IDROFOBICA
1.
15-45 AMMINOACIDI
2.
DI REGOLA AMMINOTERMINALE
3.
IDROFOBICA
4.
IN GENERE RIMOSSA
5.
SEQUENZA VARIABILE
ESEMPIO:
NH2-MMSFVSLLLVGILFWATEAEQLTKCEVFQ
MOVIE
SRP
SRP
E’ COSTITUITA DA 1 RNA E 6 PROTEINE
B) La particella SRP
Figure 12-39a Molecular Biology of the Cell (© Garland Science 2008)
COME SRP INTERAGISCE COL RIBOSOMA
QuickTime™ and a
TIFF (U ncompressed) decompressor
are needed to see this pi cture.
IL RECETTORE DI SRP E’ LOCALIZZATO SUL RER
1. PROTEINA
TRANSMEMBRANA
DEL RER
2. COSTITUITO DA
2 SUBUNITA’ (SRa ed
SRb)
3. SI DISTACCA
DA SRP CON
IDROLISI DI GTP
SRP SVOLGE 3 FUNZIONI
1
2
3
riconoscimento
del segnale
blocco della
traduzione
legame al
recettore
IL TRASLOCONE E’ UN CANALE COSTITUITO DA
+ puromicina
TRAM+SEC61
IL TRASLOCONE SEC61 PUO’ TROVARSI IN UNA CONFIGURAZIONE
CHIUSA O APERTA
solco
APERTO
IL RIBOSOMA INTERAGISCE CON LE SUBUNITA’ DEL SEC61
LA PEPTIDASI DEL SEGNALE E’ UN ENZIMA CRITICO
1. ENZIMA LUMINALE
DEL RER
2.
VARIE SUBUNITA’
3. RICONOSCE UN
SITO ADIACENTE AL
PEPTIDE SEGNALE
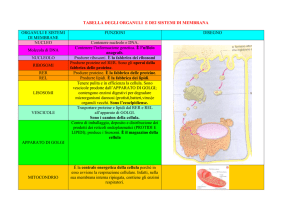
RER E SER POSSONO ESSERE PURIFICATI
Traslocazione di proteine “solubili”
Figure 12-45 Molecular Biology of the Cell (© Garland Science 2008)
Glicoforina
LDL (R)
Insulina (R)
HA
Proteine integrali di membrana sintetizzate
nel RER hanno diverse topologie
Cit. p450
CITOSOL
Cit. b5
Asialo (R)
Transferrina (R)
Galattosil transf.
Sialil transferasi
Rodopsina
b-adr. (R)
Movie 12.6
L’orientamento degli aa carichi del peptide segnale
determina la topologia della proteina: TIPO 2
La disposizione degli aa carichi nel peptide segnale
determina la topologia della proteina: TIPO 1
LA TRASLOCAZIONE NEL RER DI ALCUNE
PROTEINE PUO’ ESSERE POST-TRADUZIONALE
LE PROTEINE NEL RER SUBISCONO MODIFICAZIONI
1.
GLICOSILAZIONE (asparagine)
2.
FORMAZIONE DI PONTI DISOLFURO
3.
OLIGOMERIZZAZIONE
4.
RIMODELLAMENTO DELLE CATENE
OLIGOSACCARIDICHE
5.
ARCHITECTURAL EDITING
QUALITY CONTROL
1.
GLICOSILAZIONE (asparagine)
OT
OLIGOSACCARIL TRANSFERASI
RIBOFORINA I
RIBOFORINA II
PROTEINA 48kDa
ASN-X-SER
ASN-X-THR
OLIGOSACCARIDE
RER
Glu
CITOSOL
DOLICOLO
P
DOLICOLO
P
P GlcNAc
DOLICOLO
P
P GlcNAc Man
Man GlcNAc
5
2
P
P
DOLICOLO
Man GlcNAc
9
2
P
P
DOLICOLO
Man GlcNAc
3
9
2
P
P DOLICOLO
2
2
5
Ser/Thr
X
Asn
= N-Acetilglucosamina
= Mannnosio
Man
= Galattosio
Gal
= Acido sialico
= Glucosio
Glu
GlcNAc
NANA
Ser/Thr
RER
X
Asn
Ser/Thr
X
Asn
GOLGI
Elaborazione degli oligosaccaridi N-Linked nel RER
2.
FORMAZIONE DI PONTI DISOLFURO
PDI (PROTEINA DISOLFURO ISOMERASI)
AMBIENTE NON RIDUCENTE
IDROLISI DI ATP
3.
OLIGOMERIZZAZIONE
ALCUNE PROTEINE SONO COSTITUITE DI PIU’
SUBUNITA’.
LE SUBUNITA’ SI ASSOCIANO NEL RER.
L’OLIGOMERIZZAZIONE E’ SPESSO
INDISPENSABILE PER USCIRE DAL RER.
4.
RIMODELLAMENTO DELLE CATENE
OLIGOSACCARIDICHE
LE 3 MOLECOLE DI GLUCOSIO VENGONO
RIMOSSE DALL’OLIGOSACCARIDE PRIMA
CHE LA PROTEINA ESCA DAL RER.
5.
ARCHITECTURAL EDITING
QUALITY CONTROL
NEL RETICOLO ENDOPLASMATICO SONO
PRESENTI NUMEROSE PROTEINE CHE
SVOLGONO FUNZIONI DI CHAPERONI O
CHAPERONINE. QUESTE PROTEINE:
1. INTERVENGONO NEL RIPIEGAMENTO
DELLE PROTEINE IN TRANSITO.
2. IMPEDISCONO CHE PROTEINE NON
CORRETTAMENTE RIPIEGATE ESCANO
DAL RER.
ESEMPI: BiP - CALNEXINA - CALRETICULINA - HSP70
DURANTE LA TRASLOCAZIONE NEL RER LE PROTEINE SI
LEGANO A CHAPERONI E SI RIPIEGANO
2001
L’ESEMPIO DELL’EMOAGGLUTININA (HA)
C’E’ UNA RELAZIONE TRA GLICOSILAZIONE E RIPIEGAMENTO
The Unfolded Protein Response (UPR)
Nucleus
UPRE
UPR genes
S1P/S2P
– BiP,PDI,SCJ1
XBP-1
– BiP, CHOP
– ERAD (ER-ass. degradation)
– XBP1
–XBP1
Phospholipid
Synthesis
mRNA
– Lipogenesis
ATF6
IRE
Golgi
ER
Unfolded
protein
© Sabine Schnyder 2005
BiP
Figure 12-55a Molecular Biology of the Cell (© Garland Science 2008)
PROTEINE DEL RER RIPIEGATE MALE POSSONO
ESSERE RETROTRASPORTATE E DEGRADATE
LA SINTESI DI LIPIDI AVVIENE SUL RE
Per quanto riguarda i lipidi delle membrane dei mitocondri,
essi sono in parte sintetizzati in loco e in parte derivano
da siti di assemblaggio a livello del reticolo
endoplasmatico, con passaggio mediato da proteine di
trasferimento (o proteine di scambio dei lipidi).
La membrana esterna ha composizione in lipidi del
50% e presenta colesterolo; pertanto è simile a quella
del RE.
Composizione della membrana interna e delle creste
La membrana interna ha un alto contenuto in proteine
(circa il 70%).Ha similitudini con la membrana dei
batteri: è priva di colesterolo, presenta cardiolipidi.
La cardiolipina è un lipide che rappresenta il 20%
circa delle molecole della membrana interna
mitocondriale. La cardiolipina è responsabile della
forte impermeabilità della membrana interna ai
protoni.