Interferenza e vettori
27-X-06
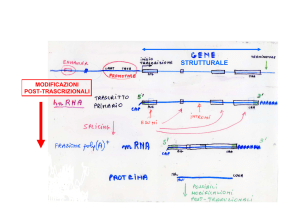
Per reprimere l’attività genica : metodo definitivo = knock out
di un gene,
metodo temporaneo : prima antimessaggeri, ora interferenza
L’antimessaggero ha un’azione meccanica
(appaia il messaggero)
L’interferenza è un sistema enzimatico complesso
È un sistema fisiologico
Come si può fare interferenza
Vediamo come si può dimostrare che esiste
l’interferenza e che se si inibisce ricomincia
l’attività enzimatica su cui abbiamo interferito
Si tratta di fare degli esperimenti che osservano il
fenomeno e poi di dimostrare il meccanismo con
cui si verifica
Per dimostrare che il meccanismo identificato è il
responsabile effettivo dell’interferenza devo avere un
modello in cui funziona l’interferenza e poi devo
bloccare l’interferenza (in un punto del pathway) e
ripristinare l’espressione bloccata dalla interferenza.
Espressione di emerald GFP
B) Cells transfected with Lipofectamine™
2000 (efficiency 50%)
- 100% tracking of EmGFP and miRNA
expression.
-1° Hoechst nuclear stain After 48 hours,
- cells stained with a red lamin stain, and
monitored for GFP expression.
-2° half of the cells express lamin protein
-3° cells expressing EmGFP and lamin
A/C stained are combined,
- cells expressing GFP do not appear to
have lamin A/C present,
- cells stained red for lamin A/C do not
appear to have any GFP expression.
- This demonstrates that cells expressing
EmGFP are also all greatly reduced in
lamin expression due to the presence of
the miRNA that is co-cistronically
expressed.
Come è il costrutto ?
vettore GFP+lamina siRNA
espressione di un micro RNA
(emerald green fluorescent protein)
Sequenze di integrazione retrovirale
Vettore per interferenza
L’interferenza avviene per effeto di un enzima “dicer”
che serve come difesa da RNA virali (forse anche altro)
Come sarà il costrutto ?
se dobbiamo esprimere la GFP e inibire l’espressione di
Lamin A/C
(emerald green fluorescent protein)
lamin A/C siRNA
small interfereing
mi = micro
Sequenze di integrazione retrovirale
Vettore per interferenza
L’interferenza = meccanismo enzimatico specifico di difesa da
infezione di dsRNA, presente in quasi tutti gli org. superiori
Interference come difesa
A brief history of RNAi: the silence of the genes.
Sen GL, Blau HM.
Department of Molecular Pharmacology, Baxter Laboratory in Genetic Pharmacology, 269
Campus Dr., CCSR 4225A, Stanford University School of Medicine, Stanford, California 94305,
USA. [email protected]
The use of the RNA interference (RNAi) pathway to eliminate
gene products has greatly facilitated the understanding of gene
function. Behind this remarkable pathway is an intricate network
of proteins that ensures the degradation of the target mRNA. In
this review, we explore the history of RNAi as well as highlighting
recent discoveries.
FASEB J. 2006 Jul;20(9):1293-9.
PMID: 16816104 [PubMed - indexed for MEDLINE
1° paper plant cell (petunia) 1990; 2° Neurospora 1992;
Sistema enzimatico Dicer 1998-2000
Dimostrazione tramite
esperimenti di controllo
Dimostrazione: soppressione con antisenso RNA
l’interferenza è dal blocco con antimessaggero ?
Come lo dimostrereste ?
Un antimessaggero può legare il messaggero e bloccarne
il funzionamento (va trasfettato nella cellula)
Piccoli RNA possono bloccare l’espressione di un gene
osservazione in piante (x cambiar colore alle petunie)
osservazione di silenziamento (quelling) in Neurospora crassa
Esperimenti tramite vettori
Per ogni esperimento un vettore con caratteristiche specifiche
tramite tecniche di DNA ricombinante e trasfezioni
vettori di controllo con e senza inserto
I exp. overexpression in petunia del gene “chalcone syntase”
biosintesi antocianine - colore viola dei fiori
fiori binchi = repressione dell’espressione (‘90)
II exp. Neurospora crassa (Romano e Macino) sequenze
omologhe di geni inibivano temporaneamente
specificamente l’attività di molti geni (‘92)
III exp. Caenorabditis elegans sequenze senso ed antisenso
inibiscono il gene par 1 (gene della partizione) (‘98)
IV exp. Verifica che è il dsRNA a innescare l’interferenza
Altre evidenze di RNAi
Gli esperimenti a singola elica di RNA erano
contaminati da dsRNA
Mancavano i controlli giusti di purificazione dell’ RNA
Exp con singolo filamento senso o antisenso
10-100 volte minor efficienza rispetto a dsRNA
Ipotesi di un prodotto intermedio stabile
Exp in C.elegans mostrava interferenza nella II generazione
Isolamento di un piccolo RNA da 25 nt antisenso
Exp in cellule di Drosofila con firefly luciferase: interferenza
con dsRNA 22-23nt con sporgenza libera di 2-3 nt. Si
inibisce sia il gene esogeno che endogeno
Interferenza nei mammiferi
Nelle cellule di mammifero dsRNA lunghi inducono
interferone e blocco della sintesi proteica (traduz.)
- alternativa: prova con dsRNA corti
Ipotesi di funzionamento in 2 passaggi:
1° taglio del dsRNA in piccoli frgm 22-23nt
2° taglio del mRNA specifico
Exp. di centrifugazione per separare le due attività
nel surnatante resta solo attività di taglio del dsRNA
Ipotesi di attività ribonucleasica della famiglia delle Rnase-III
Exp. costrutti con epitopo tag T7 di fusione a RNAse (tipo 1-2-3) che funge
da carrier, trasfezione in cellule S2 di Drosofila, immunoprecipitazione, 1
solo enzima che taglia dsRNA in frammenti di 21-23nt. L’ enzima viene
chiamato DICER legato ad Rnase di tipo III.
Mutante KO perde attività di interference.
Omologhi di DICER in tutte le specie
Cosa è l’immunoprecipitazione
La tecnica di immunoprecipitazione la dovreste conoscere,
però in poche parole:
Molti enzimi formano dei complessi oppure
interagiscono con altri enzimi, proteine o DNA
immunoprecipitazione con uno degli enzimi del complesso
da studiare
Si deve avere un buon anticorpo : non deve interagire col
sito attivo che lega l’altro enzima
Viene fatto l’estratto cellulare e si fa reagire con l’anticorpo
contro la proteina, la precipitazione dell’anticorpo con la
proteina fa da carrier per le altre proteine legate
Immunoprecipitazione e artefatti
in breve I
La tecnica ha molti rischi di carry-over
Come si può fare per non precipitare artefatti
Evidenze incrociate con altre tecniche
Se la proteina che coprecipita è già nota è più facile
dimostrare la sua identità con un anticorpo specifico
Inversione dell’esperimento = col 2° ab si ripesca il
1°enzima
Controlli con altri tipi di estratti e con condizioni diverse
Stressare le condizioni di legame per eliminare legami
aspecifici
Per proteine nuove il rischio di vedere artefatti è più difficile
da controllare
Imm.precipitazione con DNA* o RNA
in breve II
Immunoprecipitazione del DNA legato a fattori di trascrizione
Anticorpo specifico (monoclonale, dovete sapere cosa è)
Non deve alterare il sito di legame al DNA
homeo box
zip domain
zinc finger
Viene fissata la prot. al DNA precipitato e amplificato
Si deve conoscere proteina (Ab) e sito di legame (PCR)
Verifica del legame in vitro o in vivo
*CHIP = chromosome immune-precipit.
Iterferenza exp in cellule Hela
Cellule epiteliali di carcinoma umano della cervice
Exp. trasfezione con siRNAs biotinilato
co-immunoprecipitazione: coprecipita compl. enzimatico
purificazione di 2 enzimi ≈100 kDa
famiglia geni Argonauta (sviluppo Drosofila Ago1 e Ago2
interaz. con metab. RNA)
Solo Ago 2 ha la capacità di taglio, gli altri legano piccoli RNA
Topo transgenico per Ago2 non chiude il tubo neurale
Attività Rnase, sito attivo con motivo DDE (aspart. glutam.)
Identico ad Ago2 - due domini PIWI e PAZ
sito PIWI taglia e PAZ lega mRNA (slicer activity)
Mutazioni al sito DDE non tagliano
Più attività e funzioni di un sistema
complesso
Come viene scelta l’elica del dsRNA da processare ?
quale elica lega RISC/Ago2 ? l’antisenso è meno stabile
RISC avrebbe 2 attività come si libera la siRNA dal dsRNA ?
Il complesso formato da Dicer-Ago2- siRNA con TRBP
Nuova evidenza che RISC è complessato ad rRNA 80S
(controlla i messaggeri per la traduzione)
Negli oociti di Drosofila non c’è traduzione e nemmeno
l’interferenza funziona
Necessità di altro exp di conferma
prossima diapo
RISC = RNA interference (gene)-silencing complex
TRBP = HIV transactivating response RNA binding protein
Exp traduzione - interference
Sistema Iron = ferro
Le proteine regolatrici del ferro IRPs in assenza di ferro
legano siti specifici di risposta al ferro che stanno nei mRNA
dei geni di controllo Iron e bloccano la traduzione
impedendo ai fattori di inizio di legare l’mRNA
Esperimento con reporter gene regolato con siti Iron
L’attività RISC non richiede traduzione di mRNA
RNA-induced gene-silencing complex (RISC)
siRNAs contro il gene reporter: era attivo sia in presenza
che assenza di ferro, indipendentemente che l’mRNA fosse
tradotto o meno
Vettore per dsRNA palindrome
In alternativa al primo vettore con GFP e lamina A/C costitutivo
Vettori per il silenziamento di geni espressi con metodo piu’ efficace
di quello dell’RNA antisenso. Paddison et al. PNAS 5 Feb. 2002
Clonaggio con inserimento di elementi fiancheggianti LoxP del fago
P1 che tramite l’enzima CRE (ricombinasi) se gli elementi sono in
palindrome produce una inversione dell’inserto, se gli elementi sono in
tandem fa excidere ed integrare se c’e’ un solo elemento
pcDNA3
P
GFP
ZEO
r
L
GFP
L
ZEO r
Gene per la resistenza alla
zeocina;
L Lox P;
GFP
Le prime 500 bp codificanti di
EGFP;
P
Promotore di
citomegalovirus;
funzione del vettore per interferenza
P
CMV
GFP
ZEO r
L
GFP
L
Ricombinasi CRE
P
CMV
GFP
ZEO r
L
GFP
L
ZEO r
dsGFP
Exp DICER + GFP
pGFP
+
500ng dsRNA
+
1000ng dsRNA
Control P19
pGFP
+
no dsRNA
ds FF
siRNA marker
(~ 500 bp)
P19 GFP Harpin
Enzima Dicer
dsRNA
(due negazioni affermano !)
L’interferenza contro la GFP
siRNA
viene bloccata bloccando
(~ 21 - 20 nts)
DICER con interferenza.
Exp. very elegant
pGFP
siRNA
ds Dicer
In vivo
Cellule trasfettate con pGFP e dsRNA di DICER o Firefly GFP
Dimostrazione che è DICER a mediare l’interferenza
P19 EC cells: cellule con il vettore stabilmente integrato.
2 modelli di interferenza: Modello 1
dicer TRBP
Dicer taglia il ds RNA
Ago2
lungo in siRNA
Ago2
TRBP
Formazione del
complesso
Ago2 + TRBP
Ago2
5’ degradato da exonucleasi
Corpi P di
degradazione
di mRNA
3’ finisce nei corpi P e Ago2
viene rilasciato di nuovo
Il lungo RNA a doppio filamento è degradato da
DICER che recluta Ago2 e TRBP. Ago2 taglia il
piccolo siRNA che poi si associa al mRNA, viene
tagliato in due frammenti, l’exisoma digerisce il
frgm 5’, il frgm 3’ entra nel corpo P e la
degradazione con Xrn1* rilascia Ago2 che nel
citoplasma ricomincia il ciclo e si riassocia ad un
altro complesso mRNA
*Xrn1 =
esonucleasi
modello di interferenza n.2
mRNA si associa nei corpi P
tramite RCK (elicase) che
permettono ad AGO2 e RISC di
trovare il bersaglio di ibridazione
con il siRNA, dopo l’ibridazione
con il bersaglio l’ mRNA è tagliato
ed i frammenti degradati all’interno
del corpo P
Inibizione della traduzione tramite RCK
ribosoma
Il ribosoma si
disassembla
Ago2
DICER e TRBP
Digeriscono il
dsRNA in siRNA
approfondimento
PLoS Biol. 2006 Jun 13;4(7):e210 [Epub ahead of print] Links
Translation Repression in Human Cells by MicroRNA-Induced Gene Silencing
Requires RCK/p54. Articolo disponibile o-l sulla biblioteca bio-medica di TV
Chu CY, Rana TM.
Department of Biochemistry and Molecular Pharmacology, University of Massachusetts Medical School, Worcester,
Massachusetts, United States of America.
RNA interference is triggered by double-stranded RNA that is processed into small interfering RNAs
(siRNAs) by Dicer enzyme. Endogenously, RNA interference triggers are created from small noncoding RNAs
called microRNAs (miRNAs). RNA-induced silencing complexes (RISC) in human cells can be programmed
by exogenously introduced siRNA or endogenously expressed miRNA. siRNA-programmed RISC (siRISC)
silences expression by cleaving a perfectly complementary target mRNA, whereas miRNA-induced silencing
complexes (miRISC) inhibits translation by binding imperfectly matched sequences in the 3' UTR of target
mRNA. Both RISCs contain Argonaute2 (Ago2), which catalyzes target mRNA cleavage by siRISC and
localizes to cytoplasmic mRNA processing bodies (P-bodies). Here, we show that RCK/p54, a DEAD box
helicase, interacts with argonaute proteins, Ago1 and Ago2, in affinity-purified active siRISC or miRISC from
human cells; directly interacts with Ago1 and Ago2 in vivo, facilitates formation of P-bodies, and is a general
repressor of translation. Disrupting P-bodies by depleting Lsm1 did not affect RCK/p54 interactions with
argonaute proteins and its function in miRNA-mediated translation repression. Depletion of RCK/p54
disrupted P-bodies and dispersed Ago2 throughout the cytoplasm but did not significantly affect siRNAmediated RNA functions of RISC. Depleting RCK/p54 released general, miRNA-induced, and let-7-mediated
translational repression. Therefore, we propose that translation repression is mediated by miRISC via
RCK/p54 and its specificity is dictated by the miRNA sequence binding multiple copies of miRISC to
complementary 3' UTR sites in the target mRNA. These studies also suggest that translation suppression by
miRISC does not require P-body structures, and location of miRISC to P-bodies is the consequence of
translation repression.
conclusioni
Resta da capire se è valido il modello n.1 o n.2
Potrebbe venir proposto un altro modello alla luce di
nuove evidenze che sconfessa quelli precedenti
“ del doman non v’è certezza ” L. da Vinci
L. il Magnifico
A. Poliziano
L. Pulci
?