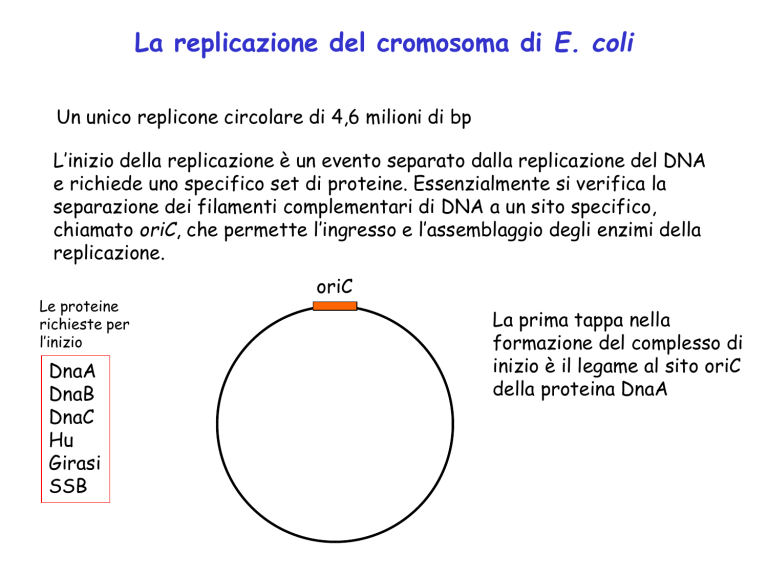
La replicazione del cromosoma di E. coli
Un unico replicone circolare di 4,6 milioni di bp
L’inizio della replicazione è un evento separato dalla replicazione del DNA
e richiede uno specifico set di proteine. Essenzialmente si verifica la
separazione dei filamenti complementari di DNA a un sito specifico,
chiamato oriC, che permette l’ingresso e l’assemblaggio degli enzimi della
replicazione.
Le proteine
richieste per
l’inizio
DnaA
DnaB
DnaC
Hu
Girasi
SSB
oriC
La prima tappa nella
formazione del complesso di
inizio è il legame al sito oriC
della proteina DnaA
Il sito oriC presenta sequenze ripetute
di 9 e 13 basi all’interno di una regione di
245 bp che costituisce la sequenza
minima necessaria per l’inizio della
replicazione. Le due sequenze sono
diverse ma comunque ricche in AT.
Si ritiene che DnaA riconosca inizialmente
le sequenze di 9 bp e vi si leghi in maniera
cooperativa fino a che 20-40 monomeri
formino un nucleo centrale attorno al quale
si avvolge il DNA di oriC
Il risultato è l’apertura delle sequenze
ripetute di 13 bp ricche in AT. A questo
punto entrano DnaB, C ed altre proteine
che formano ul complesso di 480.000
daltons.
DnaB è l’elicasi necessaria per separare la
doppia elica. Altre proteine sono necessarie
per l’inizio come pure per il prosieguo della
replicazione e sono la DNA girasi che
rimuove superavvolgimenti positivi e SSB
che stabilizza il DNA a singola elica.
Si è venuta a formare così una forca replicativa da dove la sintesi di
DNA potrà procedere bidirezionalmente non senza che sia prima
stato sintetizzato un innesco di RNA che funge da primer per la DNA
polimerasi III
Le varie fasi della replicazione possono essere seguite al ME. All’inizio si
ha la struttura a “occhio” che si trasforma nella struttura intermedia a
“teta” con l’avanzamento delle forche replicative
forma
terC
23 bp
La replicazione termina al sito di terminazione indicato come terC dove si
incontrano le due forche replicative. La regione contiene due sequenze di 23
bp che delimitano il punto di incontro.
La completa replicazione del cromosoma richiede circa 40 minuti
La segregazione del nucleoide
La replicazione del cromosoma è seguita dalla segregazione: processo dove
i cromosomi duplicati vengono disposti ai lati opposti della cellula in
divisione. I batteri quali E. coli riescono a portare a termine questo
processo in modo efficiente nonostante l’assenza di un apparato mitotico
I geni responsabili di questo processo non sono ancora del tutto noti ma
sicuramente iplicato è l’operone mukFEB
Il gene B codifica per una proteina di 177 kDa che è la più grande proteina
conosciuta in coli e ha una struttura simile alla miosina eucariotica. Ha una
attività ATPasica e GTPasica e la parte globulare ha affinità per il DNA.
Mutanti in mukB portano alla formazione di cellule anucleate
Problematiche poste dalla replicazione
Quale è il segnale che dà inizio alla replicazione?
Quale meccanismo mette in relazione la fine della replicazione con la
successiva divisione?
L’inizio della replicazione è determinato dal raggiungimento di una massa
critica, quindi la frequenza di inizio è legata alla massa cellulare. Più un
batterio cresce velocemente, cioè più velocemente può aumentare la sua
massa, più frequentemente inizierà la replicazione.
Deve pertanto esistere un meccanismo di controllo che correli la massa
all’inizio della replicazione
Un modello per spiegare il fenomeno è che esista un iniziatore che venga
sintetizzato costitutivamente e si accumuli proporzionalmente
all’aumento della massa cellulare fino a raggiungere una concentrazione
critica che avvii l’inizio della replicazione. Il modello non è stato ancora
provato
Il ciclo cellulare di Escherichia coli
I
variabile
C
40 min
D
20 min
Fase I: periodo di interinizio. Osservabile in cellule che si dividono lentamente
(T>60’); la cellula si prepara alla replicazione del DNA
Fase C: periodo necessario alla replicazione del DNA. Il limite è dovuto alla
velocità di sintesi del DNA da parte della DNA polimerasi
Fase D: periodo della divisione. E’ il tempo che intercorre tra il completamento
della replicazione del cromosoma e la divisione cellulare.
Il paradosso della divisione in E. coli
Come può un batterio dividersi ogni 20 minuti se il DNA impiega 40 minuti
per duplicarsi?
forca replicativa
T = 60’
0
10
20
30
40
50
C
D
40 min
20 min
60
T = 40’
replicazione iniziata 20’ prima
0
10
20
30
D
40
50
60
T = 20’
replicazione che inizia
replicazione iniziata
20’ prima
replicazione iniziata 40’ prima
la distanza tra il centro dei
due nuceoidi deve essere pari
a una lunghezza cellulare per
permettere la formazione del
setto
1L
La divisione in E. coli
La divisione nei batteri gram-negativi comporta la cresita interna e
coordinata dei tre strati che delimitano il citoplasma sotto forma di un setto
La formazione del setto richiede una regolazione:
1) TEMPORALE
2) SPAZIALE
La regolazione temporale fa in modo che la divisione avverrà sempre dopo la
duplicazione del DNA (un blocco nella sintesi del DNA non sarà seguito dalla
divisione).
Un ruolo importante è svolto dalla proteina SulA (sistema sos) che inibisce
in modo selettivo la principale proteina responsabile della formazione del
setto (FtsZ).
Altri fattori che attivano la formazione del setto devono ancora essere
individuati.
La regolazione spaziale della divisione in E. coli
In E. coli come in altri batteri di forma bastoncellare, il setto si localizza
esattamente nella posizione centrale della cellula.
Come viene scelta la corretta posizione del setto?
SI
NO
Si ritiene che siano 2 i fattori coinvolti nel corretto posizionamento del
setto:
1) OCCLUSIONE DEL NUCLEOIDE
2) SISTEMA Min
Occlusione del nucleoide
Il nucleoide esercita un effetto negativo sul posizionamento del setto: in
una regione occupata dal nucleoide non si formerà mai il setto.
Il volume cellulare è occupato dal
nucleoide. Non ci sono siti liberi
per la formazione del setto
In seguito alla segregazione del
DNA, la regione mediana rimane
libera dal nucleoide. Il setto può
formarsi
Non è richiesta la completa separazione dei nucleoidi ma è sufficiente una
diminuizione della concentrazione del DNA
Il sistema Min
Il sistema Min di E. coli consiste di 3 proteine:
MinC
MinD
MinE
MinC è un inibitore della divisione ed è attivato da MinD. (complesso MinCD)
L’azione inibitrice di MinC è esercitata direttamente sulla proteina FtsZ
MinD è una proteina associata alla memrana
MinE agisce su MinD ed è responsabile della localizzazione spaziale del
complesso MinCD: la sua funzione è quella di far localizzare MinCD ai
poli della cellula e lontano dal piano di divisione mediano.
Ma come agisce MinE?
MinE provoca una rapida oscillazione di MinD tra un polo e l’altro della
cellula nel giro di pochi secondi. In questo movimento è coinvolto anche
MinC in quanto associato a MinD.
Comportamento dinamico di GFP-MinD
tratto da:
The EMBO Journal, Vol. 20, No. 7 pp. 1563-1572, 2001
La rapida oscillazione fa in modo che l’inibitore del setto MinCD sia
statisticamente più presente ai poli che al centro impedendo, quindi, la
formazione del setto ai poli
I poli sono siti potenziali di divisione non occlusi dal nucleoide
La formazione di un setto a un polo porta alla formazione di minicellule
(cellule di dimensioni più piccole e anucleate)
minicellula
Formazione del setto e divisione in E. coli
Una fase precoce della divisione nei batteri è la formazione dello Z ring a
livello del sito di divisione. La formazione dello Z ring è dovuta alla capacità
della proteina FtsZ di polimerizzare
Z-ring
FtsZ-GFP
La sintesi dello Z-ring parte da un sito di nucleazione localizzato a livello della
membrana citoplasmatica e prosegue bidirezionalmente fino a formare un anello
Sito di nucleazione
GTP
FtsZ
GDP
Z-ring
Lo Z-ring va incontro a una successiva costrizione fornendo la forza che
porta all’invaginazione della membrana citoplasmatica seguita dagli altri
strati fino alla completa chiusura del setto e divisione della cellula
Oltre FtsZ altre proteine partecipano alla formazione del setto:
ZipA, FtsA, FtsI, FtsL, FtsN, FtsQ, FtsK, FtsW
A)
I geni che codificano per le proteine
del setto sono essenziali
Per ogni gene sono stati isolati mutanti
letali condizionali (termosensibili)
B)
Questi mutanti, a temperatura non
permissiva (42°C), hanno un fenotipo
caratteristico (foto A): formano lunghi
filamenti non settati o con il setto
appena accennato
Fts = Filaments
Foto B) Mutanti Fts a temperatura
permissiva (30°C)
FtsZ
E’ la proteina principale del setto e quella che interviene per prima
Proteina citoplasmatica di 40.000 D presente in circa 20.000 copie per cellula
E’ omologa alle tubuline eucariotiche
E’ una GTPasi. E’ capace di polimerizzare. La polimerizzazione richiede GTP
La polimerizzazione è inibita da SulA (controllo della replicazione) e da MinC
(controllo spaziale della localizzazione del setto)
Tra le proteine Fts è quella più conservata tra i procarioti: è assente solo
nelle clamidie. E’ presente in alcuni mitocondri e cloroplasti
FtsA
Proteina citoplasmatica presente in circa 200 copie per cellula
E’ omologa all’actina e ha una attività ATPasica
Interagisce direttamente con FtsZ ed è la seconda a far parte del setto
ZipA
E’ una proteina di membrana.
Interagisce direttamente con FtsZ. Ancora lo Z-ring alla membrana
FtsI, L, N, K, Q, W
Sono proteine di membrana. Si associano allo Z-ring dopo FtsA e ZipA
Il loro ruolo è ancora sconosciuto (FtsI è responsabile della sintesi del
peptidoglicano a livello del setto)
I virus
I virus: caratteristiche generali
I virus sono agenti infettivi con le seguenti caratteristiche distintive:
dimensioni estremamente piccole
parassitismo intracellulare obbligato
organizzazione strutturale semplice non cellulare
presenza di un solo tipo di acido nucleico (DNA o RNA)
si moltiplicano per sintesi separata e successiva riunione
dei loro componenti
Spettro d’ospite. I virus si moltiplicano solo in particolari cellule
ospiti e di conseguenza vengono suddivisi in tre classi principali:
virus batterici (o batteriofagi)
virus animali
virus vegetali
Origine dei virus
Evoluzione retrograda
I virus sarebbero forme degenerate di parassiti intracellulari: i loro
genomi si sarebbero progressivamente ridotti in quanto la maggior parte
delle funzioni vengono fornite dalla cellula ospite.
Questa ipotesi potrebbe spiegare l’origine di virus complessi quali i
Poxvirus
Teoria della “evasione dei geni”
I virus si sarebbero evoluti da elementi genetici capaci di replicazione
autonoma (plasmidi e trasposoni)
Struttura dei virus
Le particelle virali, o virioni, sono costituite da una o più molecole di
DNA o RNA rivestite da un involucro proteico o capside.
In alcuni virus il nucleocapside (acido nucleico + capside) può essere
avvolto da un rivestimento membranoso (envelope)
Le dimensioni possono variare da 10 a 400 nm
In base alle caratteristiche morfologiche è possibile individuare 4
gruppi principali:
Virioni con capside icosaedrico
Virioni con capside elicoidale
Virioni dotati di envelope
Virioni complessi
Sia i capsidi elicoidali che icosaedrici sono strutture macromolecolari
costruite da una o poche subunità proteiche, dette capsomeri, ripetute un
gran numero di volte. Questa strategia costruttiva ha il vantaggio di utilizzare
con la massima efficienza l’informazione immagazzinata nel genoma virale
Capsidi a simmetria elicoidale
Di forma cilindrica, costituiti da un unico tipo di capsomero avvolto a
spirale attorno all’asse centrale contenente l’acido nucleico
I capsidi possono essere rigidi (virus del
mosaico del tabacco e il batteriofago M13) o
flessibili (virus influenzali)
Esempio: virus del mosaico del tabacco (TMV)
Capside cilindrico rigido di 15-18 nm di diametro
e circa 300 nm di lunghezza. L’unica proteina del
capsomero è di 158 aa
Acido nucleico: RNA a singolo filamento di
6000 nucleotidi
Cristallizzato da Stanley nel 1935
Spesso la ripetizione di una sola proteina
rappresenta l’unica possibilità di rivestire un
acido nucleico di piccole dimensioni
Capsidi a simmetria icosaedrica
Icosaedro: solido regolare a 20
facce triangolari e 12 vertici
I capsomeri sono formati da 5 o 6 protomeri che possono essere anche tutti
identici
I pentoni o pentameri possiedono 5 subunità e
sono disposti ai vertici dell’icosaedro; gli esoni
o esameri hanno 6 subunità e costituiscono le
facce
Il numero totale di capsomeri per i diversi virus
assume valori definiti da equazioni derivate
dalla cristallografia; sono possibili solo i valori
12, 32, 42, 60, 72, 92, 162, 252 ecc
Esempi: i batteriofagi X174, MS2, Qb
parvovirus (12 capsomeri), poliovirus (32 capsomeri),
papilloma virus (72 capsomeri), adenovirus (252)
L’auto-assemblaggio dei virus
Protomeri e capsomeri sono legati tra loro mediante legami deboli non
covalenti. Infatti, i capsidi (vuoti) sono facilmente dissociabili nei loro
costituenti.
Una volta sintetizzati ed esposti alle opportune condizioni, i protomeri
interagiscono tra loro in modo specifico, associandosi spontaneamente a
formare il capside
L’autoassemblaggio generalmente non richiede alcun intervento dall’esterno.
Alcuni virus più complessi codificano per particolari fattori che, pur non
essendo incorporati nel virione, risultano necessari per l’assemblaggio del
virus
Virus dotati di envelope
Presentano il nucleocapside rivestito da strutture membranarie complesse (involucro
pericapsidico)
I virus con involucro sono comuni nel mondo animale, ma si conoscono anche virus batterici
L’involucro pericapsidico è generalmente costituito da un doppio strato lipidico in
cui sono inserite glicoproteine (spicole)
La membrana deriva dalla cellula infettata in
seguito al processo di liberazione dei virioni;
le glicoproteine sono di origine virale
Il capside all’interno può avere simmetria elicoidale o icosaedrica
Virus con capsidi a simmetria complessa
Hanno una struttura complessa non assimilabile né a quella elicoidale né
a quella icosaedrica
Rappresentanti: i poxvirus e i grandi batteriofagi
I poxvirus, di forma ovoidale o a mattone, sono i virus animali più grandi e
possono essere osservati anche al microscopio ottico
Il genoma (DNA a doppio filamento) è associato a proteine e racchiuso in
un nucleoide, a forma di disco biconcavo, delimitato da una membrana. Due
corpi laterali, di forma ellittica, sono disposti tra il nucleoide e l’involucro
esterno. L’involucro è di natura membranaria, rivestito da tubuli e fibre
I genomi virali
Per la maggior parte dei virus a RNA il materiale genetico è rappresentato
da un singolo filamento di RNA (ss RNA)
Se la sequenza corrisponde a quella dell’mRNA virale il filamento è definito
positivo o +
Esempi: il virus polio, il virus del mosaico del tabacco, e il virus del sarcoma
di Rous sono virus con genoma a ssRNA positivo
Se la sequenza è complementare a quella dell’mRNA virale il filamento è
definito negativo o Esempi: il virus della rabbia, il virus del morbillo, e il virus dell’influenza
sono virus con genoma a ssRNA negativo
I virus animali con filamento positivo presentano modificazioni tipiche dei
messaggeri eucariotici: 7-metilguanosina (cap) al 5’ e coda di poli-A al 3’
Molti genomi a RNA risultano segmentati e si ritiene che ogni segmento
codifichi per una proteina virale
Virus con capsidi a simmetria complessa
I batteriofagi della serie T pari (T2, T4, T6)
testa
colletto
asse tubulare
(cavo)
guaina
elicoidale
fibre
caudali
spine
placca basale
esagonale
Virus con capsidi a simmetria complessa
I batteriofagi T1, T5 e l hanno una testa icosedrica, la coda è priva di
guaina, e non termina con una base piatta ma con rudimentali fibre caudali
Il batteriofago lambda
testa icosaedrica
coda non contrattile
I batteriofagi T3 e T7 possiedono una testa icosedrica, la coda breve non
contrattile e priva di fibre caudali
Tutti questi batteriofagi sono detti virus a simmetria binaria, poiché hanno
sia la simmetria icosaedrica (testa) che elicoidale (coda)
I genomi virali
I virus contengono un unico tipo di acido nucleico in tutte e quattro le
combinazioni:
DNA a singola elica
DNA a doppia elica
RNA a singola elica
RNA a doppia elica
Anche la struttura del genoma è varia nei diversi virus. Il materiale
genetico può essere organizzato in :
un’unica molecola lineare
un’unica molecola circolare (DNA)
segmentato in più molecole
I genomi virali
La capacità codificante va dalle 3-4 proteine dei virus più piccoli (MS2 e Qb)
alle oltre 100 proteine dei batteriofagi della serie T-pari e dei Poxvirus
Nella maggior parte dei virus a DNA il genoma è costituito da un doppio
filamento (ds DNA) che può essere lineare oppure circolare
Circolarizzazione del DNA di l
GGGCGGCGACCT
CCCGCCGCTGGA
GGGCGGCGACCT
CCCGCCGCTGGA
La formazione dell’RNA messaggero virale
Affinché possano essere sintetizzate le nuove proteine virali, è prima
necessario che siano sintetizzati nuovi messaggeri virali
Virus a DNA
+
-
+
ds DNA
ss DNA
RNA pol usa come stampo il filamento
DNA pol
+
-
RNA pol
-
mRNA (+)
mRNA (+)
La formazione dell’RNA messaggero e replicazione virale
N.B. Sia nelle cellule eucariotiche che procariotiche sono presenti solo RNA
polimerasi DNA dipendenti (non possono usare RNA come stampo)
Virus a RNA positivo
+
funziona direttamente da mRNA
ss RNA
+
mRNA (+)
Replicasi
(RNA polimerasi RNA dipendente)
Il filamento + funge direttamente da mRNA che codifica, oltre alle altre proteine
necessarie, anche la Replicasi (Rna pol RNA dipendente). Questa inizialmente sintetizza RNA
negativi usando come stampo il filamento positivo del genoma, e poi li usa come stampi per
produrre ulteriori molecole positive che costituiranno i genomi della progenie virale
La formazione dell’RNA messaggero e replicazione virale
Virus a RNA negativo
-
ss RNA
mRNA (+)
-
RNA polimerasi RNA dipendente
associata al virus
L’RNA genomico non può funzionare come mRNA né le cellule ospiti hanno polimerasi
capaci di trascrivere mRNA usando come stampo RNA.
Come può essere sintetizzato RNA messaggero?
Per superare il problema questi virus portano, nel loro capside, alcune molecole di
Trascrittasi (Rna pol RNA dipendente) che inizialmente sintetizza RNA positivo che
funge da messaggero, e da questo, in seguito sintetizzera gli RNA negativi che
costituiranno il genoma della progenie virale
La formazione dell’RNA messaggero e replicazione virale
Virus a RNA a doppio filamento
Trascrittasi
+
-
ds RNA
RNA polimerasi RNA dipendente
associata al virus
usa come stampo il filamento
+
-
mRNA (+)
Replicasi
(RNA polimerasi RNA dipendente)
Anche in questo caso l’RNA genomico non può funzionare come RNA messaggero
Alla sintesi del messaggero provvede una Trascrittasi associata al virus e inettata
insieme al genoma virale. Il messaggero virale codifica per un’altra RNA pol RNA
dipendente che svolge la funzione di Replicasi e sintetizzerà RNA a doppio filamento
per le nuove particelle virali.
La formazione dell’RNA messaggero e replicazione virale
Retrovirus
Trascrittasi inversa
+
ss RNA
DNA polimerasi RNA dipendente
associata al virus
+
+
ss RNA
mRNA (+)
(virione)
Sono virus a singolo filamento di RNA + ma si differiscono da tutti gli altri perché
sintetizzano i propri mRNA e replicano il proprio genoma passando attraverso un
intermedio a DNA
Sono caratteristici anche per il fatto di essere “diploidi”. Infatti, nel loro capside sono
presenti sempre due filamenti di RNA. Associati al genoma sono anche la Trascrittasi
inversa (DNA polimerasi RNA dipendente) e tRNA di origine virale che svolge la funzione
di primer per la sinstesi del DNA virale
La trascrizione inversa
3’
5’
tRNA virale
ibrido RNA/DNA
attività RNAsi H
degradazione RNA
sintesi del filamento complementare di DNA
3’
5’
Coltivazione dei virus
I virus animali vengono coltivati inoculando animali sensibili o uova
embrionate di pollo (uova fecondate e incubate per 6-8 giorni)
Di recente i virus vengono coltivati su monostrati di cellule animali; questa
tecnica mette in evidenza aree localizzate di lisi cellulare chiamate placche,
o foci proliferativi (virus oncogeni)
I virus vegetali possono essere coltivati impiegando colture di tessuti
vegetali, oppure piante intere. Le foglie si inoculano meccanicamente
spalmandole con una miscela di virus e una sostanza abrasiva. Nelle aree
infette si sviluppa una lesione necrotica localizzata
I batteriofagi sono i virus più facilmente coltivabili. L’infezione di una
coltura di batteri sensibili porta nel giro di circa un’ora alla lisi dei batteri
con produzione di una notevole quantità di virus (lisato)
Metodi di saggio dei virus
È possibile determinare la quantità di virus presente in un campione:
1) contando il numero di particelle virali
2) misurando la concentrazione di unità infettanti
Il numero delle particelle virali può essere anche 1 milione di volte
superiore al numero delle unità infettanti
Conteggio del numero di particelle virali
diretto: microscopio elettronico
indiretto: saggio di emoagglutinazione
Conteggio delle unità infettanti
saggio delle placche
dose infettante (ID50) e dose letale (LD50)
Conteggio al ME
Il campione virale viene mescolato a microscopiche sferette di latex a
concentrazione nota. Per una determinata superficie si fa il rapporto tra il
numero di virus e il numero di sfere e si risale alla concentrazione virale
Saggio di emoagglutinazione
eritrociti
virus
Diluizioni seriali di sospensione virale vengono aggiunte ai globuli rossi. Il titolo
emoagglutinante è dato dalla diluizione più alta che dà ancora agglutinazione
diluizione
10
20
40
80
160
320
640
1280
2560
5120
Saggio delle placche
infezione
4-5 ml di top agar
placche di lisi
su tappeto
batterico
ultima
diluizione
virale
dopo 24h
0,2 ml di batteri
Ogni placca ha avuto origine da una singola particella virale, quindi, il numero
delle placche prodotte equivale al numero di virioni infettanti o di unità
formanti placca (PFU).
Dal numero delle placche e considerando la diluizione effettuata, si risale alla
concentrazione di particelle infettanti.
Il saggio delle placche può essere applicato anche a virus animali e vegetali
quando si dispone di cellule sensibili all’infezione che crescano in vitro
In alternativa, molti virus animali possono essere inoculati sulla membrana
corionallantoidea degli embrioni di pollo dove producono lesioni localizzate
dette pustole
Per i virus vegetali si spalma una sospensione di virus con una sostanza
abrasiva su foglie sensibili. I virus daranno delle lesioni necrotiche
In tutti i casi, moltiplicando il fattore di diluizione per il numero di pustole
o lesioni necrotiche si ottiene la concentrazione di unità infettanti di una
sospensione virale
Quando è impossibile ricorrere al saggio delle placche si usa il metodo della
diluizione finale. Organismi viventi o colture cellulari sono inoculati con
diluizioni seriali di una sospensione virale per determinare la diluizione che
uccide o danneggia il 50% delle cellule o degli organismi impiegati.
LD50 = dose letale per il 50% del campione
ID50 = dose infettante per il 50 % del campione
Efficienza di piastramento
Il rapporto quantitativo tra il numero delle unità infettanti e il numero delle
particelle virali (determinato mediante conteggio al microscopio elettronico)
è definita efficienza di infezione.
E.I =
Unità Infettanti
Conteggio al ME
I conteggi ottenuti con il saggio delle placche sono sempre inferiori a quelli
ottenuti con il microscopio elettronico. L’efficienza con cui i virus infettano le
cellule ospiti raramente raggiunge il 100% e spesso può essere
considerevolmente inferiore.
Una efficienza inferiore al 100% può essere in parte dovuta alla presenza di
particelle virali difettive, ma più spesso è dovuta al fatto che, nelle condizioni
usate, l’infezione da parte di queste particelle non ha avuto successo.
Mentre per i virus batterici l’efficienza di piastramento supera spesso il
50%, nel caso di molti virus animali può essere molto bassa, tra lo 0,1 e 1%.
Anche se sono necessarie molti virus per infettare una cellula, l’infezione è
iniziata sempre da un solo virus
Le fasi della moltiplicazione dei batteriofagi
1. Adsorbimento
2. Iniezione dell’acido nucleico
3. Fase replicativa precoce
4. Replicazione del genoma virale
5. Sintesi delle proteine capsidiche
6. Assemblaggio del capside e impacchettamento del genoma virale
7. Rilascio dei virus maturi (lisi)
Esperimento di crescita a ciclo unico (one-step)
Delbruck e Ellis, 1939
L’ esperimento one-step permette di seguire le varie fasi della moltiplicazione
virale partendo da un’infezione sincrona di una coltura batterica
1) infezione di una coltura batterica (T=0 dell’esperimento). Tutti i
batteriofagi inizieranno il ciclo di moltiplicazione contemporaneamente
2) lavaggio della coltura per eliminare i batteriofagi non ancora adsorbiti
3) ad intervalli di tempo viene determinato il numero di batteriofagi
liberati mediante il saggio delle placche
Curva di crescita a ciclo unico
Numero dei batteriofagi
periodo di latenza
periodo di crescita
plateau
periodo di eclisse
volume di scoppio
=
N. fagi al plateau
N. fagi latenza
Tempo (minuti)
periodo di eclisse: periodo durante il quale non è possibile rilevare la presenza di particelle
virali infettive neanche all’interno dei batteri
periodo di latenza: periodo di sviluppo dei fagi nel batterio infettato. I fagi non sono
ancora liberati all’esterno. Nella fase finale sono presenti particelle mature nel batterio.
periodo di crescita: le cellule vanno incontro a lisi liberando i fagi infettanti. Quando tutti i
batteri infettati saranno lisati si raggiunge il plateau
L’adsorbimento dei batteriofagi
I batteriofagi non aderiscono a un punto qualunque della superficie batterica
ma riconoscono recettori specifici
I recettori sono normali componenti della superficie dell’ospite quali proteine
della parete, polisaccaridi, lipopolisaccaridi, acidi teicoici, flagelli e pili
In assenza di siti recettoriali il virus non può adsorbirsi
Se un recettore è alterato, l’ospite diventa resistente alla infezione da parte
del virus che usa quel recettore. Tuttavia, anche i virus possono mutare per la
struttura che riconosce il recettore tornando, così, capaci di infettare un
ospite resistente
Fago PBSI di B. subtilis
Penetrazione dell’acido nucleico
Nella gran maggioranza dei batteriofagi è solo l’acido nucleico virale a entrare
nel batterio.
I meccanismi di penetrazione differiscono notevolmente tra i diversi fagi
finora studiati e sono in gran parte ancora oscuri.
Nei fagi T-pari l’adsorbimento è dovuto al contatto tra le fibre caudali e il recettore
(proteina della membrana esterna). La penetrazione del DNA si verifica dopo che la
placca basale si è adagiata sulla superficie cellulare e si verificano cambiamenti
conformazionali sia nella placca che nella guaina. Quest’ultima si contrae permettendo
all’asse tubulare di penetrare attraverso la parete. Infine, con un meccanismo ancora
sconosciuto, il DNA dalla testa, passando attraverso l’asse tubulare, entra nella cellula.
E. coli infettato da T4
Regolazione dell’espressione dei geni fagici
I geni con funzioni correlate sono generalmente raggruppati per essere
espressi contemporaneamente in un preciso istante del ciclo di replicazione
I geni introdotti nella cellula ospite vengono espressi secondo una ordinata sequenza
temporale che consente una efficiente coordinazione degli eventi che portano alla
produzione della progenie virale
Geni precoci immediati: trascritti subito dopo l’ingresso, codificano per prodotti che
partecipano alla replicazione del DNA virale e che bloccano le attività del batterio. La
loro trascrizione si arresta nel giro di pochi minuti.
Geni intermedi o precoci ritardati: partecipano alla replicazione e alla ricombinazione
del DNA. Possono essere trascritti per l’intero ciclo replicativo
Geni tardivi: codificano per proteine capsidiche, fattori che partecipano
all’assemblaggio e proteine litiche
Il passaggio da una fase all’altra è geneticamente controllato attraverso la sintesi di
nuova RNA polimerasi (T7) o fattori che alterano la specificità della RNA polimerasi
batterica
Regolazione della trascrizione in T4
Il controllo della trascrizione in T4 coinvolge la sintesi di proteine che modificano la
specificità della RNA polimerasi dell’ospite in modo da farle riconoscere i diversi
promotori fagici. I promotori precoci immediati sono letti direttamente dall RNA
polimerasi attraverso il fattore sigma dell’ospite
geni precoci immediati
s
blocca il fattore sigma
Una delle proteine precoci immediate ha la funzione
di bloccare il fattore sigma e quando raggiunge una
certa concentrazione, i geni precoci immediati sono
bloccati
geni precoci ritardati
fattori che
modificano
l’RNA polimerasi
geni ritardati
Replicazione e sintesi delle proteine del fago T4
T4 ha uno dei più grandi genomi virali (1,7 x 105 bp) costiutito da DNA a
doppio filamento lineare
Immediatamente dopo l’ingresso del DNA vengono sintetizzate le
proteine precoci: sono gli enzimi coinvolti nella trascrizione e
replicazione del DNAvirale e enzimi che degradano il DNA batterico per
fornire nucleotidi
Vengono interrotte le attività del batterio (DNA, RNA e proteine batteriche non vengono
più sintetizzati).
Dopo 5 minuti ha inizio la replicazione del DNA virale
La sintesi del DNA di T4 richiede una notevole preparazione perché contiene
idrossimetilcitosina al posto della citosina, che successivamente viene anche glucosilata.
Questi resudui glucosilati proteggono il DNA di T4 dall’attacco di alcune endonucleasi di
E. coli, dette anche enzimi di restrizione, che altrimenti distruggerebbero il DNA virale
effettuando tagli in siti specifici.
Questo meccanismo di difesa batterica è detto restrizione.
Il DNA batterico è protetto dall’azione di questi enzimi grazie alla presenza di
enzimi di modificazione che effettuano modificazioni nei siti specifici riconosciuti dagli
enzimi di restrizione
Organizzazione del genoma del fago T4
1
2
3
4
5
6
7
8
9
1
2
3
4
5
6
7
8
9
1
2
3
4
5
6
7
8
9
1
2
3
4
5
6
7
8
9
1
2
3
4
Il genoma di T4 è un dsDNA lineare e presenta il fenomeno della permutazione
circolare e della ridondanza terminale (una stessa sequenza ripetuta alle
estremità del DNA)
La replicazione del DNA di T4
concatenamero
Dopo diversi cicli di replicazione si vengono a formare concatenameri di
lunghezza considerevole (fino a 20 copie di DNA)
Assemblaggio delle particelle fagiche
I geni tardivi dirigono la sintesi di tre tipi diversi di proteine:
1) proteine strutturali del fago
2) proteine che intervengono nell’assemblaggio ma non faranno parte del fago
3) proteine coinvolte nel processo di lisi batterica e liberazione dei fagi
Le proteine necessarie all’assemblaggio vengono sintetizzate contemporameamente e
sono utilizzate in “catene di montaggio” quasi indipendenti fino ad avere il fago maturo
L’impacchettamento del DNA avviene quando la testa è matura: circa 500 mm di DNA
devono essere inseriti in una cavità di appena 0,1 mm.
L’impacchettamento avviene secondo il meccanismo detto “a testa piena” a
partire dai concatenameri sintetizzati. Quando il volume della testa è
totalmente riempito il concatenamero viene tagliato. In questo modo entra il
102% del genoma virale.
Questo processo è responsabile della ridondanza terminale e della
permutazione circolare del genoma di T4
Ridondanza terminale e permutazione circolare del genoma di T4
123456789123456789123456789123456789123456789
102%
1234567891
102%
2345678912
102%
3456789123
102%
4567891234
X174 e il fenomeno dei geni sovrapposti
X174 è un piccolo batteriofago icosaedrico con genoma a DNA circolare a
singolo filamento
È un esempio di come virus contenenti genomi di lunghezza limitata sfruttino al
massimo l’informazione genetica attraverso il fenomeno dei geni sovrapposti
I 5386 nucleotidi del genoma di X174 non
sarebbero sufficienti a codificare tutte le
proteine virali identificate. Si è scoperto,
quindi, che una sequenza può codificare anche
per due proteine: i geni sono sovrapposti.
5386
Il gene E è contenuto all’interno di D su un altro
schema di lettura.
La proteina A* è parte della proteina A ed è
ottenuta tramite un re-inizio della traduzione.
A sintesi della forma replicativa
A* inibizione della sintesi di DNA dell’ospite
E lisi della cellula ospite
La replicazione a “cerchio rotante” di X174
+
FR
polarità +
forma replicativa a doppia elica
enzima che taglia la singola elica
elica nascente
filamento spiazzato
punto di crescita
taglio e circolarizzazione di
un genoma completo ad
opera della proteina virale A
filamento +
una rotazione
completa
+
Caratteristiche di un batterio lisogeno
I batteri lisogeni hanno il DNA virale (profago) integrato nel loro
cromosoma (nel caso del fago P1 il DNA provirale e libero nel citoplasma).
Il DNA del profago viene duplicato insieme a quello dell’ospite e trasmesso
da una generazione di batteri a quella successiva.
All’interno di una popolazione di batteri lisogeni una piccola frazione
(tra 10-3 e 10-6) delle cellule produce virus e muore.
Un batterio lisogeno per un fago è immune all’infezione con lo stesso fago.
Rimane sensibile all’infezioni di fagi differenti. L’immunità è dovuta agli
stessi sistemi regolativi virali che bloccano la via litica.
In particolari condizioni quasi tutta la popolazione di batteri lisogeni può
andare incontro a lisi e produrre nuovi fagi. Il fenomeno va sotto il nome
di induzione.
Un fago temperato può indurre cambiamenti fenotipici nel batterio. Tale
cambiamento è detto conversione lisogena. Es.: il fago temperato b di
Corynebacterium diphteriae produce la tossina responsabile della difterite
Batteriofagi temperati e lisogenia
ciclo litico
ciclo lisogenico
induzione
profago
batterio lisogeno
Il batteriofago lambda
testa icosaedrica
64 nm
coda non contrattile
150 nm
sito cos
GGGCGGCGACCT
dsDNA 48.502 bp
5’
5’
CCCGCCGCTGGA
sito cos
Fase precoce immediata
Solo due geni vengono
espressi: cro e N.
Sono gli unici due geni i cui
promotori sono riconosciuti
dalla RNA polimerasi
batterica.
Il prodotto del gene N
(antiterminatore) permette
il passaggio alla fase
successiva.
Regione d’immunità
Fase precoce ritardata
La trascrizione procede verso
destra per i geni della
replicazione e verso sinistra
per i geni della ricombinazione.
Il prodotto del gene Q
(antiterminatore) permette il
passaggio alla fase successiva.
A questo punto viene
effettuata la scelta tra il ciclo
litico e il ciclo lisogenico
Fase tardiva
È stata scelta la via litica. I geni
precoci non vengono più
trascritti.
Vengono sintetizzate le proteine
strutturali della coda e della
testa e gli enzimi responsabili
della lisi.
Se viene scelta la via litica il
cromosoma di l non si integra
nel cromosoma batterico
La via lisogenica
Il cromosoma di l si integra
nel cromosoma batterico.
Una volta integrato un solo
gene di l è responsabile del
mantenimento dello stato
lisogenico: il gene cI
attP
attB
La regione di immunità di l
Fase precoce immediata
cIII
N
cro
cI
PL/OL
cII
PR/OR
Cro
antiterminatore
responsabile della via litica
I promotori PR/OR e PL/OL sono riconosciuti dalla RNA polimerasi batterica
gene spento
gene trascritto
RNA polimerasi batterica
La regione di immunità di l
Fase precoce ritardata
cIII
N
cro
cI
PL/OL
cIII
protegge cII
gene spento
gene trascritto
RNA polimerasi batterica
cII
PR/OR
cII
regolatore positivo di cI
La regione di immunità di l
Fase precoce ritardata
cIII
N
cro
cI
PR/OR
PL/OL
cII
PE
cII + cIII
Repressore cI
responsabile del mantenimento
dello stato lisogenico
Il repressore cI di lambda
236
132
92
1
C
C
C
N
N
N
Il dominio N-terminale costituisce il sito di legame all’operatore
Il dominio C-terminale è responsabile della formazione del dimero
Il repressore è capace di legarsi al DNA solamente come dimero
La regione di immunità di l
cIII
N
cro
cI
PR/OR
PL/OL
PR
OR3
trascrizione di cI
PRM
OR2
OR1
cII
PE
trascrizione di cro
L’interruttore genetico di l
PR
OR3
OR2
trascrizione di cro
OR1
PRM
trascrizione di cI
Il legame di Cro in OR3 blocca la sintesi del repressore cI.
E’ stata scelta la via litica
L’interruttore genetico di l
trascrizione di cro
PR
OR3
OR2
OR1
PRM
trascrizione di cI
Il repressore cI ha una maggiore affinità per il sito OR1
Il legame del repressore cI in OR1 blocca la sintesi di Cro.
L’interruttore genetico di l
trascrizione di cro
PR
OR3
OR2
OR1
PRM
trascrizione di cI
L’affinità per OR2 è inferiore all’affinità per OR1
Il legame del dimero in posizione OR1 favorisce il legame di un
secondo dimero in posizione OR2 (legame cooperativo)
L’interruttore genetico di l
trascrizione di cro
PR
OR3
OR2
OR1
RNA
polimerasi
PRM
trascrizione di cI
controllo
positivo
controllo
negativo
Il legame del dimero in posizione OR2 regola positivamente la trascrizione
del repressore stesso (regolazione autogena )
Fase precoce immediata
cIII
N
cro
cI
PL/OL
cII
PR/OR
Cro
antiterminatore
responsabile della via litica
I promotori PR/OR e PL/OL sono riconosciuti dalla RNA polimerasi batterica
Fase precoce ritardata
cIII
N
PL/OL
cIII
protegge cII
cro
cI
cII
PR/OR
cII
regolatore positivo di cI
Fase precoce ritardata
cIII
N
cro
cI
PR/OR
PL/OL
cII
PE
cII + cIII
Repressore cI
Ciclo litico
cIII
N
cro
cI
PR/OR
PL/OL
cII
PE
PR
OR3
OR2
OR1
PRM
Verso la fine della fase precoce ritardata Cro si lega anche a OR2 e OR3
come pure a PL/OL bloccando la trascrizione dei geni precoci
Ciclo lisogenico
cIII
N
cro
cI
PR/OR
PL/OL
cII
PE
PR
OR3
OR2
OR1
PRM
La continua trascrizione di cI assicura il mantenimento dello stato lisogenico.
Un eccesso di cI viene ricondotto alla norma grazie al legame in posizione OR3
L’immunità alla superinfezione
Batterio lisogeno per l risulata immune all’infezione di l
cI
Il repressore codificato dal profago blocca la trascrizione dei geni precoci
del DNA infettante legandosi a PR/OR e PL/OL
Induzione del ciclo litico
L’induzione rappresenta una risposta a fattori ambientali (luce UV) o
mutageni chimici che danneggiano il DNA dell’ospite
Come parte della risposta SOS, aumenta la concentrazione della
proteina RecA che normalmente è responsabile della ricombinazione
genetica
Ad alta concentrazione RecA interagisce con cI
C
attività endoproteolitica
dell’estremità C-terminale
del repressore
C
RecA
N
N
Induzione del ciclo litico
trascrizione dei geni litici
Mutazioni a carico dei geni cI, cII e cIII danno placche dal fenotipo “chiaro”
Mutazioni a carico dei siti OR e OL possono essere “virulente”
Per la classificazione vengono prese in considerazione le caratteristiche morfologiche e la
composizione in acido nucleico
Adsorbimento dei virus animali
Adsorbimento avviene mediante l’incontro casuale tra il virus e la
cellula ospite. Generalmente una glicoproteina sulla membrana
plasmatica della cellula ospite costituisce il recettore
La capacità di un virus di infettare un determinato tipo di cellula
o di tessuto risiede in gran parte nella distribuzione dei
recettori
Il sito di legame sulla superficie del virus può essere rappresentato
semplicemente da una proteina strutturale del capside come ad esempio
le fibre che si estendono dai vertici degli icosaedri negli adenovirus, o
le spicole dei virus dotati di envelope
Penetrazione e spoliazione
La penetrazione consiste nel passaggio del virus attraverso la membrana
plasmatica.
La spoliazione è l’allontanamento del capside e la liberazione dell’acido
nucleico virale.
In alcuni virus nudi, come il poliovirus, dopo l’adsorbimento alla membrana
cellulare si verificano cambiamenti strutturali nel capside che portano alla
liberazione nel citoplasma del solo acido nucleico.
In virus rivestiti come i paramyxovirus, l’envelope virale si fonde con la
membrana citoplasmatica e il nucleocapside entra nella cellula.
Tuttavia la maggior parte dei virus con envelope entra nella cellula attraverso
un processo di endocitosi mediato dall’interazione tra il virus e i recettori
localizzati in porzioni specializzate della membrana citoplasmatica rivestite,
sul lato citoplasmatico, di una proteina detta clatrina
In seguito al contatto la membrana si introflette formando delle vescicole
contenenti le particelle virali. Le vescicole si fondono con i lisosomi e
l’acidificazione sembra favorire l’espoliazione. La membrana della vescicola
e l’envelope si fondono e il nucleocapside viene liberato nel citoplasma.
La spoliazione termina con la disgregazione del capside e la liberazione
dell’acido nucleico.
Trascrizione e replicazione
L’espressione dei geni precoci serve a ottenere il controllo della cellula ospite
e a replicare il genoma virale.
Alcuni virus animali inibiscono la sintesi di DNA, RNA e proteine dell’ospite ma
generalmente non degradano il DNA cellulare.
Virus a DNA
Per quanto riguarda i virus a DNA, la replicazione del DNA virale avviene nel
nucleo cellulare con l’eccezione dei poxvirus: la loro replicazione avviene nel
citoplasma grazie alla sintesi di una DNA polimerasi specifica.
Spesso la trascrizione dei virus a DNA avviene ad opera della RNA
polimerasi cellulare ma alcuni virus possono anche portarsi una RNA
polimerasi specifica (poxvirus).
Trascrizione e replicazione
Virus a RNA
Nella maggior parte dei virus a RNA la replicazione avviene nel citoplasma.
Virus a RNA positivo
+
funziona direttamente da mRNA
ss RNA
+
mRNA (+)
Replicasi
(RNA polimerasi RNA dipendente)
Il filamento + funge direttamente da mRNA che codifica, oltre alle altre proteine
necessarie, anche la Replicasi (Rna pol RNA dipendente). Questa inizialmente sintetizza RNA
negativi usando come stampo il filamento positivo del genoma, e poi li usa come stampi per
produrre ulteriori molecole positive che costituiranno i genomi della progenie virale
Trascrizione e replicazione
Virus a RNA negativo
-
ss RNA
mRNA (+)
-
RNA polimerasi RNA dipendente
associata al virus
L’RNA genomico non può funzionare come mRNA né le cellule ospiti hanno polimerasi
capaci di trascrivere mRNA usando come stampo RNA.
Come può essere sintetizzato RNA messaggero?
Per superare il problema questi virus portano, nel loro capside, alcune molecole di
Trascrittasi (Rna pol RNA dipendente) che inizialmente sintetizza RNA positivo che
funge da messaggero, e da questo, in seguito sintetizzera gli RNA negativi che
costituiranno il genoma della progenie virale
Trascrizione e replicazione
Virus a RNA a doppio filamento
Trascrittasi
+
-
ds RNA
RNA polimerasi RNA dipendente
associata al virus
usa come stampo il filamento
mRNA (+)
-
+
Anche in questo caso l’RNA genomico non può funzionare come RNA messaggero
Alla sintesi del messaggero provvede una Trascrittasi associata al virus e inettata
insieme al genoma virale. Il messaggero virale codifica per un’altra RNA pol RNA
dipendente che svolge la funzione di Replicasi e sintetizzerà RNA a doppio filamento
per le nuove particelle virali.
Trascrizione e replicazione
Retrovirus
Trascrittasi inversa
+
ss RNA
DNA polimerasi RNA dipendente
associata al virus
+
+
ss RNA
mRNA (+)
(virione)
Sono virus a singolo filamento di RNA + ma si differiscono da tutti gli altri perché
sintetizzano i propri mRNA e replicano il proprio genoma passando attraverso un
intermedio a DNA
Sono caratteristici anche per il fatto di essere “diploidi”. Infatti, nel loro capside sono
presenti sempre due filamenti di RNA. Associati al genoma sono anche la Trascrittasi
inversa (DNA polimerasi RNA dipendente) e tRNA di origine virale che svolge la funzione
di primer per la sinstesi del DNA virale
Sintesi e assemblaggio
I geni tardivi dirigono la sintesi delle proteine capsidiche, che si assemblano
spontaneamente come avviene per la morfogenesi dei batteriofagi.
L’assemblaggio di virus con envelope è generalmente simile a quello dei virus
nudi.
Liberazione dei virioni
I meccanismi di liberazione di virus nudi differiscono da quelli dei virus
dotati di envelope.
I virus nudi in genere vengono liberati mediante lisi della cellula ospite.
Nei virus con envelope la formazione dell’envelope è contemporanea
alla liberazione e la cellula ospite può continuare a riversare virus
all’esterno per un certo periodo di tempo
Inizialmente le proteine codificate dal
virus vengono incorporate nella membrana
citoplasmatica, poi per gemmazione del
nucleocapside si forma l’envelope.
Infezioni acute: sono caratterizzate da una comparsa abbastanza rapida e da
una durata ridotta; le cellule infettate generalmente vanno incontro a morte
(effetto citopatico) e vengono liberati numerosi virioni.
Le infezioni acute sono tipiche dei picornavirus, herpesvirus e adenovirus.
Effetti citopatici
1)
2)
3)
4)
5)
inibizione della sintesi di DNA, RNA e proteine
danneggiamento dei lisosomi con liberazione di enzimi
idrolitici
alterazione della membrana plasmatica per inserimento
delle proteine virali (attacco del sistema immunitario)
alta concentrazione delle proteine virali
rotture cromosomiche
Infezioni persistenti o croniche: possono durare anche molti anni. I virus
possono replicarsi più lentamente senza causare una sintomatologia clinica.
Sono presenti anticorpi diretti contro il virus infettante.
Il virus dell’epatite B, il virus di Epstein-Barr e il virus del morbillo possono
dare infezioni persistenti.
Infezioni latenti: dovute a virus che, dopo una iniziale fase moltiplicativa,
arrestano il proprio ciclo di sviluppo e diventano quiescenti. La fase di latenza
può durare un certo periodo dopo il quale può verificarsi una recudescenza
dell’infezione con una manifestazione spesso diversa da quella iniziale.
I virus erpetici possono dare infezioni latenti.
L’ herpes simplex di tipo 1 infetta in genere i bambini e rimane quiescente
per anni a livello dei gangli del sistema nervoso; periodicamente può
attivarsi e dare le caratteristiche lesioni erpetiche (herpes labiale)
Il virus della varicella zoster (herpes zoster) provoca la varicella come prima
manifestazione; non viene eliminato dal sistema immunitario ma diventa
quiescente nei gangli dei nervi spinali. La manifesatzione ricorrente è il “fuoco
di Sant’Antonio” per la riattivazione del virus a livello dei nervi intercostali

![Lezione 15 Virus [modalità compatibilità]](http://s1.studylibit.com/store/data/000771737_1-84b1cca561c5813066d1b76125338a98-300x300.png)










