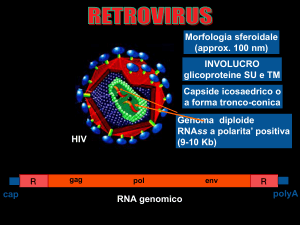
I genomi dei virus a RNA
Polarità (+)
il genoma ha la STESSA polarità degli mRNA
viene direttamente tradotto come un mRNA
Polarità (–)
il genoma è COMPLEMENTARE (polarità opposta) agli mRNA
il virus utilizza il genoma per trascrivere gli mRNA
il virus deve contenere una trascrittasi (RNA-polimerasi RNA-dipendente)
per trascrivere gli mRNA
Doppio filamento (–)/(+)
il virus trascrive gli mRNA messaggeri utilizzato il filamento
negativo del genoma.
il virus deve contenere una trascrittasi (RNA-polimerasi RNA-dipendente)
1
per trascrivere gli mRNA
Morfologia dei virus a RNA
Tutti I virus a RNA (-) presentano involucro
Alcuni virus a RNA (+) presentano involucro (Togavirus)
I virus a RNA ds (Reovirus) hanno un capside doppio
2
VIRUS a RNA
i virus ad RNA replicano nel citoplasma
eccezioni: virus dell’influenza e retrovirus
Svantaggi
Le cellule non esprimono costitutivamente gli enzimi necessari
per replicare o trascrivere molecole di RNA (il virus deve
codificare la sua propria RNA polimerasi RNA-dipendente:
replicasi/trascrittasi)
Vantaggi
la RNA polimerasi-RNA dipendente non richiede “primer” (la
trascrizione e/o replicazione dell’ RNA inizia all’estremità della
molecola lineare)
la sintesi de novo inizia all’estremità della molecola stampo
Sintesi 5’
3’
3
La RNA polimerasi RNA-dipendente dei virus a RNA
RdRP codificata dal virus
non ha funzioni di “editing” (highly error prone), responsabile
dell’alto tasso di mutazione e dell’evoluzione dei virus a RNA
può necessitare di proteine accessorie di origine virale o cellulare
alcune richiedono come primer: - proteina legata covalentemente al 5’
- strutture cap di derivazione cellulare
Caratteristiche delle RNA polimerasi RNA dipendenti
Resistenti a sostanze che inibiscono le RNA polimerasi DNAdipendenti (actinomicina D)
4
TRASCRIZIONE DEI VIRUS ad RNA
i virus ad RNA non hanno elementi di controllo
dell’espressione genica simili a quelli dei virus a
DNA
meccanismi differenti per regolare l’espressione
dei geni virali
gli mRNA virali devono essere organizzati e tradotti
come gli mRNA cellulari
i virus a RNA eucariotici devono avere una
struttura
genomica
che
genera
mRNA
monocistronici
5
Enterovirus
virus Polio (1, 2, 3)
virus Coxsackie A (1-24)
virus Coxsackie B (1-6)
virus ECHO* (1-34)
Enterovirus (68-71)
Enterovirus 72 (Epatite A)
22-30 nm
capside icosaedrico
nudo
Rhinovirus
120 sierotipi
*Enteric Cytopathogenic Human Orphan
6
Genoma a RNAss di polarita’ positiva
7441 b
vPg clivata da enzimi cellulari: il genoma diventa un mRNA
240 KDa
attive nella forma di
precursore
2C e 3AB ancorano la
replicasi alle membrane
ER
Replicasi
3D 7
POLIOVIRUS
PVR =
PolioVirus Receptor
(Ig-like membrane
glycoprotein)
RHINOVIRUS
ICAM-1 =
Adhesion molecule
8
Ruolo di VPg nella replicazione del genoma
di Poliovirus
Sintesi del filamento (-):
3AB (precursore di VPg) ancora vRNA
alle membrane del ER
3Dpol
3Cpro
lega 3AB
processa 3AB
VPg
VPg-
funge da primer per il processo di
elongazione da parte di 3Dpol
9
Poliovirus: regolazione della replicazione del genoma
A. RNA(+) non incapsidato
RNA(+) neoformato = mRNA
B. RNA(+) incapsidato
Non partecipa al processo di
replicazione/traduzione
10
Togavirus
Gruppo IV: Virus a RNA (+)
Famiglia
Genere
Specie
Ospite
Togaviridae
Alphavirus*
Sindbis virus
Vertebrati
Rubivirus
Rubella virus
Vertebrati
Particelle di 80 nm
con capside icosaedrico ed involucro
Genoma a RNAss di polarita’ positiva
11.7 kb simile a mRNA cellulare
(5’cap e 3’poly-A)
11
Strategia replicativa del virus Sindbis:
“RNA subgenomici”
proteasi (nsP2)
codificata dal virus
genoma
(subgenomic
promoter)
proteasi virali e cellulari
proteine
strutturali
del virione
RNA subgenomici: strategia comune dei virus delle piante a RNA (+)
controllo temporale dell’espressione genica virale
12
Genoma a RNAss di polarita’ positiva
Envelope (80-220 nm)
Capside elicoidale
HE
E2/S = fusione
E1/M = matrice
E = (9-12 kD)
N = nucleoproteina
HE = emagglutininaesterasi
(solo in alcuni tipi)
Malattie respiratorie (vie aeree superiori) - Gastroenteriti
13
CLASSIFICAZIONE
ORDINE
Nidovirales
FAMIGLIA
Coronaviridae
GENERE
Coronavirus
SARS virus
GROUP IV
15 specie di HCoV note
14
Genoma a RNAss di polarita’ positiva 7 x 106 d
Trasporto in vescicole
secretorie
Endocitosi o
fusione
a
Genoma RNAss(+)
b
Quick Time™ e un decompressore TIFF (Non compresso) sono necessari per visualiz zare quest'immagine.
Gemmazione
dal Golgi
a) Genoma RNAss (+)
b) Genoma RNAss (+)
polimerasi
antigenoma RNAss
Trascrizione di mRNA subgenomici per proteine strutturali con sequenze
15
identiche al 3’ e identiche sequenze leader non-tradotte al 5’ (72 nt)
VIRUS PARAINFLUENZALE
Genoma a RNAss di polarita’ negativa (17-20 Kb)
QuickTime™ and a
GIF decompressor
are needed to see this picture.
16
Terminazione e Ri-iniziazione nelle Regioni Intergeniche
Modello “Start-Stop
Pol (P+L) inizia la sintesi al terminale 3’
L: RdRP. P: recruita L sul templato.
sintesi della sequenza Leader
la Pol si ferma alla sequenza IR
la trascrizione ricomincia al 3’ del gene N
la trascreizione di N mRNA si blocca
alle sequenze IR
Pol ricomincia la sintesi al 3’ del gene P
“Start-Stop” continua fino alla sintesi dei 5 mRNA virali
17
Sintesi della coda poli-A e del cap: Ruolo delle sequenze EIS
Sintesi di sequenze A7 da U7
Sintesi continua per
scivolamento di sequenze poliA
(fino a 200A) all’estremità 3’
Terminazione del trascritto
Iniziazione e “capping” del mRNA successivo
dinucleotide
NA non copiato
18
Trascrizione polarizzata
Sequenze EIS
L
P
3’
5’
(mRNA cascade)
mRNA provvisti di sequenze cap e poly A
19
Transizione tra trascrizione e replicazione del genoma
dei PARAMYXOVIRUS
bassi livelli di NP: favoriscono la sintesi di mRNA (non incapsidato)
alti livelli di NP: la RpRd continua la sintesi di RNA attraverso le
sequenze EIS
20
VIRUS PARAINFLUENZALE
FUSIONE
Trascrizione mRNA
Trascrizione vRNA
Assemblaggio e
gemmazione
21

![Lezione 15 Virus [modalità compatibilità]](http://s1.studylibit.com/store/data/000771737_1-84b1cca561c5813066d1b76125338a98-300x300.png)