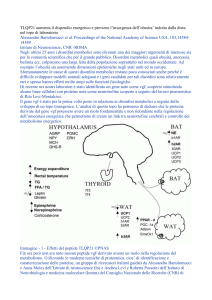
Determinazione della concentrazione proteica
mediante spettrofotometria
Spettro di assorbimento UV-VIS di una proteina
Assorbimento
Picco di assorbimento di
Tirosine e
Triptofani
220
240
260
280
300
320
340
360
Lunghezza d’onda “l” (nm)
380
400
Spettro di assorbimento UV-VIS di un RNA
Abs260nm pari ad 1 corrisponde a 40 µg di RNA
SAGGIO DI ATTIVITA’
ENZIMATICA
• SPECIFICO
• QUANTITATIVO
SAGGIO KUNITZ
CARATTERISTICHE:
• Il substrato è RNA di lievito 0,5 mg/ml
• La reazione avviene ad un pH pari a 5
• Il saggio è condotto ad una temperatura di 25°C
• La durata del saggio è di 1 minuto
• La digestione dell’RNA provoca una diminuzione di Abs300 nm
• Tale diminuzione è dovuta alla minore capacità di
assorbimento, a tale lunghezza d’onda, dei prodotti di
degradazione dell’RNA rispetto all’RNA non degradato.
Nel saggio Kunitz un’unità di attività enzimatica è
definita come la quantità di enzima in grado di
determinare una variazione di Abs per minuto pari a
∆A1min/∆Atot, dove ∆Atot rappresenta la variazione di
Abs rilevata in seguito a completa digestione del
substrato.
Il valore di ∆Atot viene determinato sperimentalmente.
RNasi A da pancreas bovino
A
H1
AS
Esempio di catalisi
acido-base enzimatica
Agiscono come catalizzatori
generali acido-basici
His 12, fungendo da base, sottrae un protone
da un gruppo 2’-OH dell’RNA, promuovendo
così l’attacco nucleofilico sull’adiacente atomo
di fosforo.
L’His 119 svolge la funzione di acido e
favorisce la scissione del legame protonando il
gruppo uscente.
L’H2O entra nel sito attivo e l’intermedio
2’,3’-ciclico è idrolizzato attraverso un
meccanismo che è essenzialmente l’inverso
della prima tappa.
L’His 12 ora agisce da acido generale e
l’His 119 da base generale per l’RNA
idrolizzato e l’enzima nel suo stato originale.
Proteine
totali
(mg)
Miscela
iniziale
RNasi A
(U)
Resa
(100%)
Purezza
(100%)
Attività
specifica
(U/mg)
100%
Frazione
Frazione
Frazione
Frazione
U di proteina di interesse nella frazione
Resa= U di prot. di interesse presenti all’inizio della purificazione
U di proteina di interesse nella frazione
s = mg di proteine totali presenti nella frazione
A
• La conoscenza della sequenza amminoacidica di una proteina costituisce il prerequisito
per la determinazione della sua struttura tridimensionale ed è essenziale per
comprenderne il suo meccanismo d’azione molecolare
• I confronti tra le sequenze di proteine analoghe ottenute da specie differenti consentono
di approfondire le nostre conoscenze sulla funzione di queste proteine e svelano le
relazioni evolutive tra le proteine e gli organismi che le sintetizzano
• Numerose malattie ereditarie sono causate da mutazioni che determinano una
sostituzione amminoacidica in una proteina → le analisi delle sequenze
amminoacidiche possono essere di ausilio nello sviluppo di test diagnostici e di terapie
efficaci
La proteina da sequenziare deve essere scissa in frammenti sufficientemente
brevi da poter essere sequenziati individualmente, poi, partendo dalla sequenza
dei tratti sovrapposti, si ricostruisce la struttura primaria della proteina intatta
DEGRADAZIONE DI EDMAN
1.
2.
3.
Il reagente di Edman, fenilisotiocianato (PITC), reagisce con il gruppo amminico
N-terminale di un polipeptide in condizioni blandamente alcaline, per formare il
feniltiocarbamile (PTC)
Tale addotto è trattato con acido trifluoroacetico (F3CCOOH) anidro, che
libera il residuo N-terminale sotto forma di derivato tiazolinonico, senza
idrolizzare gli altri legami peptidici
Il derivato tiazolinone-amminoacido è estratto selettivamente con un solvente
organico ed è convertito nel più stabile derivato feniltioidantoina (PTH) mediante
trattamento con un acido in soluzione acquosa
H
R
H
R
N C S
+
O
Condizioni basiche
NH23
O
N
N C NH
N
H S
R'
O
R'
O
H
TFA 100% anidro
O
Analisi tramite HPLC
N
R
Condizioni blande acide
in acqua
N
+
N
NH
H
S
Derivato feniltioidantoina (PTH)
(più stabile)
S
O
R
NH3
O
Derivato tiazolinonico
conversione
R'
Identificazione del PTH-aa N-terminale
mediante cromatografia
Il tipo di amminoacido è identificato con una cromatografia per
mezzo di amminoacidi standard di riferimento
Separazione dei 20 PTH-aa
Dopo circa 30 cicli diventa difficile
determinare la sequenza
amminoacidica. Ciò accade perché,
dopo un numero elevato di cicli, gli
effetti cumulativi di reazioni
incomplete e di reazioni secondarie
del processo di degradazione di
Edman rendono impossibile una
identificazione certa
dell’amminoacido rilasciato e quindi
della sequenza amminoacidica.
Per tale motivo la proteina da
sequenziare è scissa in piccoli
frammenti polipeptidici.
Una volta determinata la sequenza di
ognuno di essi, si procede con la
determinazione della sequenza della
proteina completa mediante il
principio della
sovrapposizione dei peptidi.
Proteina di 150 aa
Nter-----------K------K------R------------R---------K----------Cter
Idrolisi con TRIPSINA, peptidasi dotata di
elevata specificità: taglia i legami peptidici
dal lato C (verso il terminale carbossilico)
dei residui carichi positivamente Arg (R) e
Lys (K), se il residuo successivo non è Pro
Nter-----------Kcter
Nter------Kcter
1° peptide
2° peptide
Nter------Rcter
3° peptide
Nter------------Rcter
4° peptide
Nter---------Kcter
5° peptide
Nter----------Cter
6° peptide
Separazione dei PEPTIDI mediante cromatografia
Determinazione della sequenza N-terminale di ciascun peptide
Nter-----------Kcter
Nter------Kcter
1° peptide
2° peptide
Nter------Rcter
3° peptide
Nter------------Rcter
4° peptide
Nter---------Kcter
5° peptide
Nter----------Cter
6° peptide
3
6
5 2
4
1
•E’ necessario adoperare un metodo diverso per generare una
serie di frammenti peptidici differenti
• Anche in questo caso si procederà alla determinazione della
sequenza N-terminale di ciascun peptide ottenuto
• Le due serie di sequenze peptidiche sovrapposte consentono
di ricostruire la sequenza di ciascun polipeptide
I risultanti gruppi sulfidrilici liberi sono alchilati,
di norma mediante trattamento con iodoacetato,
per prevenire la riformazione dei ponti disolfurici
attraverso ossidazione da parte di O2
Ala-Gly-Asp-Phe-Arg-Gly
i. Determinazione della composizione in ammionoacidi
cromatografia a scambio ionico
eluzione in funzione del pH
•
Scambiatore cationico forte
•
pH 1-2 (tutti gli aa si legano)
•
Eluizione tramite aumento di pH e forza ionica:
1.
2.
3.
•
aa acidi (es. aspartato e glutammato)
aa neutri (es. glicina e valina)
aa basici (es. lisina e arginina)
Rivelazione tramite ninidrina
ii. Identificazione del residuo amino terminale
(a)
marcatura con cloruro di dansile
idrolisi
Il composto fluorescente dansil cloruro reagisce con le ammine primarie formando polipeptidi
dansilati. Il trattamento di un polipeptide dansilato con un acido in soluzione acquosa ad alta
temperatura idrolizza i suoi legami peptidici ed il residuo N-terminale risulta così libero.
Questo derivato può poi essere separato dagli altri amminoacidi per via cromatografica ed
identificato grazie alla sua intensa fluorescenza gialla.
(b)
degradazione di Edman: rimozione di un amminoacido
alla volta dall’estremità NH2-terminale
a-c Ala
c-e Gly
Degradazione di Edman Lunghezza massima 50 residui
perdita di attendibilità
Tagli specifici con metodi chimici o enzimatici
Reagente
Taglio chimico
Bromuro di cianogeno
O-iodobenzoato
Idrossilammina
2-Nitro-5-tiocianobenzoato
Taglio enzimatico
Tripsina
Clostripaina
Proteasi dello Stafilococco
Sito di taglio
Lato carbossilico di residui Met
Lato carbossilico di residui Trp
Legami Asp-Gly
Lato aminico di residui di Cys
Lato carbossilico di residui Lys e Arg
Lato carbossilico di residui Arg
Lato carbossilico di residui Asn e Gln
Prima proteina sequenziata
Sanger F., Tuppy H. (1951) “The amino-acid sequence in the
phenylalanyl chain of insulin 2. The investigation of peptides
from enzymic hydrolysates.” Biochem. J. 49:481-490
Si dimostró che la sequenza era costituita solo da L-aminoacidi
uniti tra loro da legami peptidici fra i gruppi a-amminici e i gruppi a-carbossilici