Genomica strutturale
Interesse primario della GENOMICA è di:
a) Determinare la SEQUENZA COMPLETA dei genomi di interesse
b) Identificare quanti più polimorfismi (VARIANTI ALLELICHE) possibili,
presenti nei genomi
Emblematico per le importantissime applicazioni della
genomica è stato il
PROGETTO GENOMA UMANO
Sistemi per la MAPPATURA DEI GENI
Il metodo più classico è quello dell’ASSOCIAZIONE tra
marcatori genetici, ciò prevede:
1. L ’ assegnazione di geni a determinati cromosomi (la più
semplice è risultata l’associazione con il cromosoma X)
2. La prova genetica che più geni siano assieme sullo STESSO
CROMOSOMA
3. Il calcolo della distanza tra due loci ASSOCIATI allo stesso
cromosoma mediante la frequenza di ricombinazione
Con questo sistema dell’ASSOCIAZIONE genetica classica,
solo un limitato numero di geni umani sono stati MAPPATI sui cromosomi
Le tecniche del DNA ricombinante hanno
cambiato il concetto genetico di locus
•Mendel e Morgan identificavano un locus genetico
deducendo la sua esistenza da diversi fenotipi dovuti ad
alleli differenti
•Nella GENETICA MODERNA il concetto di locus (inteso
come “concetto FISICO”) può essere separato dal concetto
di gene (identificabile con una “funzione” o fenotipo)
La costruzione di mappe genetiche (di ASSOCIAZIONE), citologiche e
fisiche è alla base del sequenziamento dei genomi
Le mappe genetiche a bassa densità sono costruite sulla base di frequenze di ricombinazione 1cM= 1UM=1%
Le mappe GENETICHE a bassa densità sono costruite sulla base di frequenze di ricombinazione -> 1cM
ricombinazione.
= 1UM = 1% frequenza di ricombinazione.
Le mappe genetiche ad alta densità, con marcatori spaziati da brevi intervalli, vengono costruite utilizzando dei
Le mappe
GENETICHE
ad alta densità, con marcatori spaziati da brevi intervalli, vengono costruite
marcatori
molecolari
come RFLP.
utilizzando dei MARCATORI MOLECOLARI come RFLP.
Le mappe citologiche sono basate sul bandeggio dei cromosomi.
Le mappe CITOLOGICHE sono basate sul bandeggio dei cromosomi.
Le mappe fisiche come le mappe di restrizione sono basate sulle distanze molecolari che separano i vari siti sul DNA.
Le mappe FISICHE, sono basate sulle distanze effettive espresse come:
•numero di coppie di basi
tra i siti
di restrizione
sul DNA dei
(mappe
di restrizione)
Le•distanze
mappe fisiche
specificano
le localizzazioni
cloni genomici
sovrappopsti o contig e sequenze nucleotidiche
•posizioni
e
distanze
tra
sequenze
nucleotidiche
uniche
chiamate sequenze “etichetta” o STS
uniche chiamate STS
(Sequence Target Sites)
•localizzazioni dei cloni genomici sovrapposti (“contig”) a coprire una regione cromosomica
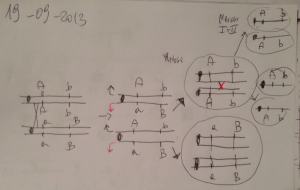
Esempio di associazione con marcatori genetici:
la sindrome Nail-Patella (NPS)
• È una sindrome che interessa le unghie (ipoplasia) e la rotula (riduzione o assenza)
• Si trasmette come un carattere autosomico dominante
00
BO
I
1
2
II
1
BO
2
BO
3
B0
4
B0
5
00
6
00
7
0
8
B0
9
00
10
00
11
00
È stato determinato il gruppo sanguigno (sistema AB0) degli individui sani e malati della famiglia
La maggior parte delle volte che è presente il gruppo sanguigno B, che mappa sul cromosoma 9 in posizione
9q34, si presenta la malattia (tranne che in soli due casi II3 3 II5); questo sta ad indicare che il locus che determina
la sindrome NPS è associato al locus del sistema ABO e quindi MAPPA sul cromosoma 9 in vicinanza della
posizione 9q34. Quanto sono vicini lo si può definire dalla frequenza di ricombinazione.
Esiste un parametro che si chiama LOD-SCORE che indica se l’ipotesi che i due loci siano associati è
statisticamente probabile (LOD SCORE = +3 ->le probabilità dell’associazione sono di 1000:1, questo è il valore
minimo di LOD SCORE per stabilire l’associazione tra due geni); poi mediante la FREQUENZA di RICOMBINAZIONE
se ne stabilisce la distanza
RFLP 2
80
RFLP 3
50
Gene Z
20
Gene Y
0
RFLP 1
Gene X
Correlazione tra mappa GENETICA, CITOLOGICA e FISICA di un cromosoma
100
125
150
MAPPA
GENETICA (cM)
MAPPA
CITOLOGICA
0
Kb 0
5
25
10
50
Kb 0
75
500
100
125
1000 Kb 0
MAPPA FISICA
(megabasi)
150
500
1000
La mappa fisica per eccellenza corrisponde alla
“sequenza completa” della regione genomica interessata!!!
2
3
4
Clone YAC 1
Mappa di
restrizione
Mappa
di STS
Mappa
di “contig”
MAPPE FISICHE
Molti loci genomici si presentano
“polimorfici”
Se si clonasse e si sequenziasse la STESSA REGIONE cromosomica di circa 250kb che comprende un
gene responsabile di una malattia, da due cromosomi omologhi della maggior parte degli individui
SANI, si troverebbe una differenza ogni 1000bp, cioè circa 250 differenze.
Si potrebbe sospettare che uno sia l'allele selvatico, mentre l'altro un allele mutante. Nessuna delle
differenze riscontrate ha però necessariamente un effetto sulla funzione reale del gene in esame;
entrambi gli omologhi potrebbero avere, infatti, un'attività selvatica.
I genetisti considerano queste variazioni trovate in qualsiasi punto del genoma come "alleli"
alternativi di un locus.
Quando in un locus di DNA esistono 2 o più alleli il locus si considera POLIMORFICO .
(Un allele per
essere considerato UN POLIMORFISMO e non una MUTAZIONE deve avere una frequenza
maggiore di 1/100, nella popolazione ).
Se questo locus polimorfico è poi usato per studi di malattie o di mappature è detto “marcatore del
DNA”.
I loci polimorfici nell'uomo potrebbero essere più di 100 milioni e questa è una enorme riserva di
potenziali marcatori del DNA.
MARCATORI MOLECOLARI per MAPPE GENETICHE
Sono utili sia per le analisi di ASSOCIAZIONE con geni responsabili di
malattie, sia per determinare le frequenze di ricombinazione tra
questi marcatori e i geni delle malattie.
SNPs ( Single Nucleotide Polymorphism)
MINISATELLITI o VNTR (Variable Number Tandem Repeat)
MICROSATELLITI o STR (Simple Tandem Repeat)
Altre modificazioni
Caratteristiche dei MARCATORI MOLECOLARI
•Sono dei veri MARCATORI GENETICI
•Corrispondono a posizioni definite nel genoma e mostrano
variabilità da un individuo ad un altro
•Le differenti forme, che corrispondono alla stessa posizione sul
genoma sono FORME ALLELICHE e vengono trasmesse secondo le
regole MENDELIANE
•Nell’ ETEROZIGOTE i due alleli sono entrambi riconoscibili :
si tratta quindi di ALLELI CODOMINANTI
Classi di polimorfismi del DNA
Classe
Dimensione totale del locus
Numero di alleli
Numero di loci nella popolazione
Frequenza di mutazione
Uso
SNPs
Singola coppia di basi
2
100 milioni
10-9
Mappatura di associazione
Microsatelliti o
STR
con ripetizioni
di 1-6 basi
da 5 a 50 volte
30-300 bp
da 2 a 10
200.000
10-3
Mappatura di associazione e
genotipizzazione
Minisatelliti o
VNTR
con ripetizioni
di 20-200 basi
da 50 a 200 volte
1000-20000 bp
da 2 a 10
30.000
10-3
Mappatura di associazione e
genotipizzazione
SNPs ( Single Nucleotide Polymorphism)
•La più semplice ed anche la più utile classe di polimorfismi si origina dalla sostituzione di
una singola coppia di basi.
•Questo tipo di cambiamenti possono essere indotti da sostanze chimiche mutagene,
oppure da errori durante la replicazione (forme tautomeriche delle basi) e sono
denominate polimorfismi di singoli nucleotidi o SNP (snips).
•Quasi tutti gli snips sono biallelici.
•Il sequenziamento della stessa regione genomica da individui diversi permette
l'identificazione di un elevato numero di SNPs.
•Esiste un'associazione (il consorzio internazionale per gli SNPs) che da Dicembre del 2001
ha identificato e mappato più di 5 milioni di SNPs umani.
•Nonostante sia possibile che gli SNPs possano alterare la sequenza aminoacidica di
proteine, la maggiore parte degli snips si verificano in "loci anonimi".
Microsatelliti o STR
•Sono elementi di DNA composti di sequenze semplici da 1 a 6 basi ripetute in tandem
da 5 a 50 volte.
•I microsatelliti si originano spontaneamente da eventi casuali, che inizialmente
producono una breve sequenza ripetuta 4 o 5 volte.
•Dopo che si è originato, un microsatellite può espandersi in una sequenza più lunga a
causa dell'appaiamento errato che può verificarsi durante la replicazione del DNA
("balbuzie durante la replicazione”).
Microsatelliti o STR
•Sono elementi di DNA composti di sequenze semplici da 1 a 6 basi ripetute in tandem
da 5 a 50 volte.
•I microsatelliti si originano spontaneamente da eventi casuali, che inizialmente
producono una breve sequenza ripetuta 4 o 5 volte.
•Dopo che si è originato, un microsatellite può espandersi in una sequenza più lunga a
causa dell'appaiamento errato che può verificarsi durante la replicazione del DNA
("balbuzie durante la replicazione”).
•Diversamente dagli SNPs che sono biallelici e non cambiano dopo che si sono originati,
i microsatelliti continuano a cambiare generando alleli multipli.
•La frequenza dei nuovi alleli è più alta della frequenza di mutazione di un singolo
nucleotide, allo stesso tempo è sufficientemente bassa da fare in modo che i
cambiamenti non avvengano nel corso di poche generazioni, anche in famiglie molto
grandi e per ciò possono essere usati come MARCATORI, abbastanza stabili e altamente
polimorfici del DNA negli studi di associazione.
Minisatelliti o VNTR
•Furono scoperti nel 1980 da Alec Jeffreys, che scoprì per primo che tra
individuo ed individuo esistevano delle variazioni che che riguardavano
sequenze ripetute migliaia di volte, che chiamò appunto minisatelliti.
•Questi loci tendono ad essere altamente polimorfici.
•Ogni 5-10 loci nel genoma c'è un MINISATELLITE
•Utilizzando diverse sequenze di minisatelliti come sonde di ibridazione
si può avere direttamente una visione globale di tutto il genoma (“DNA
fingerprint”).
Altre modificazioni: delezioni, duplicazioni e inserzioni
in loci non ripetuti
•Eventi mutazionali casuali possono causare anche una vasta categoria di
eventi che espandono o contraggono la lunghezza di un locus non ripetuto.
•Le piccole delezioni o duplicazioni si possono generare durante la
ricombinazione meiotica
•Le piccole inserzioni, che vanno da centinaia a migliaia di basi, possono
essere
causate da
ELEMENTI
TRASPONIBILI
che
si
integrano
casualmente nel genoma.
SNPs, microsatelliti, minisatelliti e delezioni inserzioni in loci non ripetuti
forniscono le basi per la MAPPATURA GENETICA ed anche, più in generale,
per il riconoscimento delle DIFFERENZE GENOTIPICHE tra gli individui
(“DNA fingerprint”).
Genotipizzazione di un individuo
Un approccio generale per determinare il GENOTIPO di un
individuo ad un particolare locus polimorfico dovrebbe consistere
nell' estrarre il DNA genomico dall'individuo, ottenere cloni
rappresentativi del locus, sequenziare il DNA.
Questo approccio sarebbe molto dispendioso in termini di tempo,
soldi e lavoro. Quindi bisogna ricorrere a strategie diverse che
permettano di analizzare centinaia di migliaia di campioni per gli
scopi, per esempio, di GENOTIPIZZAZIONE o di MAPPATURA DEI
GENOMI.
Tecniche per una DIRETTA rivelazione dei polimorfismi del DNA,
e quindi per una determinazione diretta del GENOTIPO
Si tratta di strategie diverse che permettono di analizzare centinaia
di migliaia di campioni per scopi come la GENOTIPIZZAZIONE o la
MAPPATURA DEI GENOMI
IDENTIFICAZIONE DIRETTA DEGLI SNPs
(se la sostituzione nucleotidica riguarda un SITO DI RESTRIZIONE polimorfico):
1)Tecniche del Southern blot (RFLP)
2)Tecnica della amplificazione del DNA mediante PCR
Rilevare un SNP ad un sito di restrizione mediante RFLP
Frammento di 3 kb
riconosciuto dalla sonda
Allele SNP1
GAATTC
CTTAAG
sonda
Eco RI
GAA TTC
CT T AAG
GAATTC
CTTAAG
Eco RI
Eco RI
Sito di restrizione
POLIMORFICO per EcoRI
Eco RI
Allele SNP2
Eco RI
GAATTC
CTTAAG
sonda
GAG TTC
CT C AAG
GAATTC
CTTAAG
Frammento di 5kb riconosciuto dalla sonda
5 kb 5 kb
3 kb 3 kb
Rilevare un SNP mediante amplificazione con PCR
Se l’ SNP riguarda un sito di restrizione POLIMORFICO e se è nota la sequenza che si trova ai lati del
sito di restrizione, il polimorfismo può essere determinato attraverso la PCR in 3 passaggi:
1) amplificazione della regione polimorfica compresa tra i due primers (alcune centinaia di coppie di basi)
2) esposizione del prodotto di amplificazione all'enzima di restrizione per cui il sito è polimorfico
3) analisi dei frammenti ottenuti su gel di agarosio.
Upper primer
Pro Glu Glu
CCT GAG GAG
GGA CTC CT C
Allele emoglobina A normale
amminoacido n. 6 Lower primer
modificato
Sito
MstII
Pro Val Glu
CCT GTG GAG
GGA CAC CT C
Sito di restrizione assente
La
lunghezza
totale
del
frammento amplificato è di circa
500 bp -> se il sito è presente
(allele
A,
normale)
si
produrranno due frammenti di
200 e 300 bp; se il sito è assente
(allele S, mutato) si produrrà un
unico frammento di 500 bp
500 bp
300 bp
200 bp
Allele emoglobina S
anemia falciforme (in questo caso
l’SNP riguarda una sequenza CODIFICANTE)
Trasmissione ereditaria di un allele RFLP associato
ad un carattere che si trasmette come un carattere
AUTOSOMICO DOMINANTE
L’esame dell’albero
ed
il
Southern
indicano che i figli
colpiti
hanno
ereditato
un
cromosoma che porta
l’allele A. Se questa
modalità
viene
confermata in grandi
famiglie ed in più
generazioni, l ’ allele
mutato relativo alla
PATOLOGIA
è
localizzato
sullo
stesso
cromosoma
dell’allele A.
sonda
Strategia per associare un RFLP o un altro MARCATORE
polimorfico ad un locus cromosomico che può essere
responsabile di una malattia
È necessario disporre di :
1. una famiglia numerosa con più generazioni in cui sia presente
e trasmessa una patologia genetica
2. una raccolta di sequenze clonate che rilevi gli RFLP
(almeno una per ogni cromosoma umano).
Si costruisce un albero genealogico per determinare le modalità di trasmissione del
carattere e per identificare i membri colpiti. Si analizzano poi i marcatori RFLP
specifici dei singoli cromosomi, nei componenti della famiglia.
Se la patologia genetica ed il marcatore RFLP specifico del cromosoma
vengono ereditati insieme in varie generazioni, la patologia genetica e
l'RFLP devono essere vicini sullo stesso cromosoma.
Mappatura di un gene associato ad una malattia mediante RFLP
GENiTORE A con
la malattia X
Mutazione responsabile
della malattia X
a localizzazione sconosciuta
Cromosoma del
genitore che ha
la malattia
Durante le meiosi dell’individuo, ce ne saranno alcune
in cui non avverrà la ricombinazione ed altre in cui
questa avverrà e ci saranno nuove combinazioni di alleli
Per ogni cromosoma umano sono attualmente
disponibili decine di marcatori RFLP
In seguito alla FECONDAZIONE molti individui della
generazione successiva presentano la malattia X
ed i marcatori RFLP
c
c
c
c
c
c
Ogni volta che si eredita la malattia sul cromosoma 6 è
presente il marcatore RFLP c; quando c ’ è l ’ allele c ’
l’individuo è sano!
Formazione di un nuovo allele per la “ balbuzie” della DNA
polimerasi
Allele con (CA)15
5’ C A C A C A C A C A C A C A C A C A C A C A C A C A C A C
3’ G T G T G T G T G T G T G T G T GT G T G T G T G T G T G
C
A
C
A
C
Filamento nuovo DNA polimerasi
A
C AC
C
A
C
A
C
A
C
A
C
A
C
A
5’
3’ G T G T G T G T G T G T G T G T G T G T G T G T G T G T G
La DNA polimerasi fa una pausa; all’estremità del filamento di
nuova sintesi la doppia elica si “apre”
3’
5’
3’
5’
CA
CA
CA
C A C 3’
5’ C A C A
3’ G T G T G T G T G T G T G T G T GT G T G T G T G T G T G 5’
AC
C A
Per caso, i due filamenti si
riappaiano fuori registro
CA
CA
CA
C A C 3’
5’ C A C A C A C A C A C A C A
3’ G T G T G T G T G T G T G T G T GT G T G T G T G T G T G 5’
AC
Completamento della replicazione
C A
5’ C A C A C A C A C A C A C A C A C A C A C A C A C A C C 3’
3’ G T G T G T G T G T G T G T GT G T G T G T G T G T G T 5’
Riparazione del DNA nel filamento stampo
Nuovo allele (CA)17
5’ C A C A C A C A C A C A C A C A C A C A C A C A C A C A C A C A C 3’
3’ G T G T G T G T G T G T G T G T GT G T G T G T G T G T G G T G T 5’


![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)