MARCATORI POLIMORFICI DEL DNA
Restriction Fragment Length Polymorphism (RFLP) (Studi di
associazione)
Variable Number of Tandem Repeats (VNTR)
Minisatelliti (Diagnostica forense)
Microsatelliti (Studi di associazione e diagnostica forense)
Single Nucleotide Polymorphism (SNP) (Studi di associazione
anche per caratteri quantitativi)
ENZIMI DI RESTRIZIONE
1) Sequenza di riconoscimento (da 4 a 8 basi) detta
palindrome: rispetto ad un asse di simmetria esiste la
stessa sequenza di basi su entrambi i filamenti quando
questi vengono letti in direzione 5’- 3’;
2) Sporgenze al 5’ o al 3’;
3) Estremità coesive o non coesive;
4) Isoschizomeri (enzimi diversi che riconoscono la
stessa sequenza);
5) Enzimi con sequenze di riconoscimento diverse ma
che creano estremità compatibili (isoschizomeri) (es:
BamHI e MboI).
Restriction Fragment Length Polymorphism (RFLP)
Polimorfismo per lunghezza di frammenti di restrizione
Identificazione di RFLP attraverso Southern blot
Identificazione di RFLP attraverso Southern blot
Identificazione dell’allele S per la falcemia attraverso un RFLP nel
gene beta della globina
MINISATELLITI
Sequenze ripetitive ipervariabili disperse nel genoma principalmente
nei telomeri (9-64 bp in blocchi da 0,1 a 20 kb).
Variabilità inter-individuale dovuta al numero di ripetizioni in tandem
Un locus
1
Un altro locus
2
Un altro locus
3
Esempio:
Sequenza GGGCAGGAXC (core) simile alla sequenza “chi” di E. coli,
segnale per la ricombinazione generalizzata
Uso del fingerprint del
DNA per scopi medicolegali
(minisatelliti di Jeffreys)
(A) test di paternità
(B) test per casi di
violenza sessuale
MICROSATELLITI o STR (SHORT TANDEM REPEATS)
Simple sequence repeats (1-5 bp) disperse nel genoma
(blocchi inferiori a 100 bp)
Variabilità inter individuale per numero di ripetizioni in tandem
Microsatelliti analizzati con PCR e gel di acrilammide
Eredità di microsatelliti in una famiglia
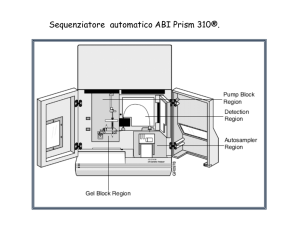
Microsatelliti amplificati con PCR multiplex e analizzati con
sequenziatore
Marcatura di un primer
di ogni PCR con
diverso fluoroforo
Microsatelliti amplificati con PCR multiplex e analizzati con
sequenziatore
Microsatelliti analizzati con sequenziatore
Fingerprinting genetico mediante microsatelliti
1
sbagliato!
IDENTIFICAZIONE DEL SESSO MEDIANTE PCR Y-SPECIFICA
MAPPA CROMOSOMICA DEI GENI AmelX E AmelY
Primo esone del gene AmelY più lungo di 6 basi rispetto al gene AmelX.
Nel maschio: due bande di 106bp (AmelX) e 112bp (AmelY).
Nella femmina: due bande sovrapposte di 106bp (AmelX).
Amelogenina: proteina presente nello smalto dei denti
Single Nucleotide Polymorphism (SNP)
Solitamente 2 alleli
Frequenti nel genoma (1 ogni kb)
Tasso mutazione: ca 10-9
Tipizzazione con procedura automatizzata
1000 Genomes Project: identificati oltre 11 milioni di SNP
Single Nucleotide Polymorphism (SNP)
Soggetto
eterozigote
C/T
APLOTIPO
Specifica combinazione di due o più alleli di marcatori del
DNA situati vicino l’uno all’altro sulla stessa molecola di
DNA.
I polimorfismi per singolo nucleotide (SNP) sono i marcatori
più frequenti utilizzati per costruire aplotipi.
Gli aplotipi si generano per caso quando una serie di
mutazioni strettamente associate si accumulano nel corso
del tempo in cromosomi discendenti da un cromosoma
ancestrale
FORMAZIONE DI APLOTIPI NEL CORSO DI UN TEMPO EVOLUTIVO
Comparsa di 2
mutazioni
Comparsa di 3
mutazioni
Diversi per 5
SNP
APLOTIPO
introne
esone
3’ UTR
+/-
+/-
Locus genico
Alleli RFLP
+/C/T
Alleli SNP
Popolazione A
1
Aplotipi RFLP 2
3
Aplotipi SNP 1
2
3
4
A/G
C/T
+
+
-
+
+
+
+
-
-
C
T
T
C
G
G
C
C
A
C
C
A
Ricerca geni malattia in popolazioni isolate
Tutti (o quasi) i malati hanno una regione aplotipica identica che
circoscrive il gene responsabile della malattia
MAPPATURA RISPETTO AD APLOTIPO CON MARCATORI
MICROSATELLITI DEL DNA
Possibile
localizzazione
gene malattia
Rispetto ai marcatori sul braccio lungo del cromosoma, il gene
patologico mappa prossimalmente rispetto a S129.