Luglio-Settembre 2013 • Vol. 43 • N. 171 • Pp. 179-185
Frontiere
Il contributo della genetica alla comprensione
delle basi eziopatogenetiche del diabete di tipo 1
Maristella Pitzalis1, Magdalena Zoledziewska1, Francesco Cucca1,2
1
2
Istituto di Ricerca Genetica e Biomedica (IRGB) del Consiglio Nazionale delle Ricerche (CNR), Monserrato
Dipartimento di Scienze Biomediche, Università di Sassari, Sassari
Riassunto
Il diabete di tipo 1 (DT1) è causato dalla distruzione selettiva delle β cellule pancreatiche producenti l’ormone insulina da parte di linfociti T autoreattivi. Il
processo autoimmune alla base della malattia dipende dalla complessa correlazione tra numerose varianti genetiche di suscettibilità distribuite nell’intero
genoma e fattori ambientali di rischio ancora sconosciuti. La ricerca dei fattori genetici coinvolti nella malattia è iniziata nei primi anni ’70 attraverso una
lunga serie di studi di associazione, che hanno portato all’identificazione di varianti di rischio nel complesso maggiore di istocompatibilità (MHC-HLA) e,
nei 30 anni successivi, a livello dei geni INS, CTLA4, PTPN22 e IL2RA. Tali studi si basavano sul disegno sperimentale del gene candidato e sul successivo
mappaggio fine, un laborioso approccio genetico-statistico volto ad identificare le varianti casuali – ovvero quei polimorfismi genici il cui prodotto proteico è
direttamente implicato nella patogenesi della malattia. A partire dal 2007, gli studi di associazione su tutto il genoma (GWAS) hanno rivoluzionato la ricerca
delle basi genetiche della malattia. Tali studi, attraverso un approccio più sistematico in grado di analizzare centinaia di migliaia di varianti in casistiche di
decine di migliaia d’individui, hanno finora svelato oltre 50 loci di suscettibilità per la malattia. Nel loro complesso, questi risultati delineano con sempre
maggiore chiarezza i complessi meccanismi alla base della patogenesi della malattia.
Summary
Type 1 diabetes (T1D) results from the T lymphocytes’ attack on the insulin-producing beta cells. This autoimmune process depends on the complex interplay between several co-inherited susceptibility alleles interspersed throughout the genome and still unknown environmental factors. The role of specific
variants within the MHC/HLA region, in the insulin gene (INS) promoter region and at the CTLA4, PTPN22 and IL2RA genes have been clarified through a long
series of association studies which began in the early ’70s. These studies were largely based on a candidate gene approach with subsequent fine mapping
to reduce the associated regions to the essential elements, whose protein products are directly involved in disease pathogenesis. Since 2007, Genome Wide
Association Studies (GWAS), based on the analyses of tens of housands of individuals and interrogating hundreds of thousands of variants, have revolutionized the analysis of the genetic component of T1D, leading to the discovery of over 50 susceptibility loci. Overall these results have highlighted mechanisms
and pathways involved in disease pathogenesis.
Parole chiave: diabete di tipo 1, studi di associazione su tutto il genoma, varianti genetiche
Key words: type 1 diabetes, genome wide association study, genetic variants
Metodologia della ricerca bibliografica effettuata
La ricerca bibliografica effettuata su PubMed ha preso in esame la
letteratura scientifica recente sulla genetica del diabete di tipo 1
(DT1) e qualche richiamo su lavori scientifici antecedenti utili per
inserire le nuove acquisizioni in un contesto storico.
Introduzione
Il DT1 è un esempio paradigmatico di malattia multifattoriale causata
dal concorso di numerosi fattori di rischio genetici in presenza di fattori ambientali permissivi ancora sconosciuti. Che i geni abbiano un ruolo importante nel conferire suscettibilità al DT1 è noto da molto tempo,
come dimostrato dall’osservazione elementare che il rischio di malattia aumenta quanto più vicino è il grado di parentela con una persona
affetta. Il rischio di malattia in un fratello e in un gemello monozigote
di un paziente con DT1 è per esempio, rispettivamente circa 15 e
100 volte più alto rispetto a quello della popolazione generale. Negli
ultimi decenni quest’osservazione epidemiologica è stata corroborata
da risultati sperimentali, a cominciare da quelli accumulati a partire
dai primi anni ’70 che hanno mostrato con risoluzione via via maggiore l’associazione di varianti nella regione del complesso maggiore di
istocompatibilità (MHC-HLA) con la malattia. Successivamente sono
stati identificati altri fattori di rischio, in particolare alcune varianti polimorfiche nella regione promotrice del gene che codifica per l’insulina
(INS) (Bell et al., 1984), nel gene codificante per la proteina Cytotoxic
T-Lymphocyte Antigen 4 (CTLA-4) (Ueda et al., 2003), nella proteina
tirosina fosfatasi N22 espressa nei linfociti (PTPN22) (Bottini et al.,
2004) e nel recettore alfa dell’interleuchina-2 (IL2RA, CD25) (Vella et
al., 2005). A fronte di questi successi la ricerca delle basi genetiche
del DT1, così come quella di altre malattie multifattoriali, è stata per
lungo tempo caratterizzata da una lunga serie di studi contrassegnati
da evidenze deboli e mai replicate.
Lo studio delle componenti genetiche del DT1 e di altre malattie
a trasmissione ereditaria multifattoriale, ha subito finalmente una
rapida accelerazione nel corso degli ultimi anni grazie all’avvento
degli studi di associazione sull’intero genoma (GWAS). Questo metodo d’analisi ad alta risoluzione, grazie ad un incremento considerevole del potere statistico, ha consentito l’identificazione di oltre
50 associazioni inequivocabili tra specifiche varianti polimorfiche
ed aumento di rischio di malattia (Figg. 1 e 2). Tali studi si basano
su ampie casistiche di diverse migliaia e in molti casi decine di
migliaia di individui nei quali viene stabilito il profilo genetico a
livello di diverse centinaia di migliaia e più recentemente di mi-
179
M. Pitzalis, M. Zoledziewska, F. Cucca
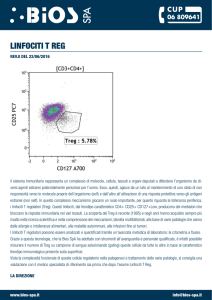
Figura 1.
In tabella sono stati riportati tutti i loci inequivocabilmente associati al diabete di tipo 1 - ovvero con significatività statistica (Pvalue) <5x10-8 o con
più moderata evidenza (Pvalue <10-4) se la stessa regione è già un locus confermato in altre malattie autoimmuni (www.t1dbase.org modificata).
Chiave di lettura per i nomi delle malattie. BD- Disordine Bipolare, Celiac- Celiachia, Crohn- Malattia di Crohn, Graves- Malattia di Graves, IBD- malattia infiammatoria intestinale, JRA- Artrite Reumatoide giovanile, MS- Sclerosi Multipla, PBC- Cirrosi Primaria Biliare, Psoriasis- Psoriasi, RA- Artrite
Reumatoide, SLE- Lupus Eritematosus Sistemico, SS- Sclerodermia sistemica, Sjogren- Sindroma di Sjogren , Spondylitis- spondilite anchilosante,
T1D- Diabete di tipo 1, T2D- Diabete di tipo 2, UC- Colite Ulcerativa, Vitiligo- Vitiligine
180
Il contributo della genetica alla comprensione delle basi eziopatogenetiche del diabete di tipo 1
lioni di varianti polimorfiche a singolo nucleotide (SNP). Tutto ciò
è reso possibile dagli avanzamenti tecnologici ed analitici legati
all’avvento dei microchip, al completamento del sequenziamento
dell’intero genoma umano e successivi sforzi volti a caratterizzare con una risoluzione sempre maggiore la variabilità genetica in
differenti popolazioni. Appare anche evidente che la componente
genetica del DT1 non spiega interamente l’eziopatogenesi della
malattia. La sua incidenza è infatti costantemente aumentata nei
paesi sviluppati a partire dal dopoguerra e previsioni allarmanti
prospettano che, entro il 2020, l’incidenza potrebbe raddoppiare
nei bambini sotto i 5 anni di età (Patterson et al., 2009). Questo
aumento di incidenza sta avvenendo in un arco di tempo troppo
breve – corrispondente a poche generazioni – per poter essere
causato da cambiamenti nell’assetto genetico, che per insorgere
e fissarsi nella popolazione richiede tempi evoluzionistici. È molto
verosimile che modifiche del contesto ambientale nel corso degli
ultimi ’60 anni abbiano giocato un ruolo nell’aumento dell’incidenza. La ricerca dei fattori ambientali è peraltro complicata dalla
lunga latenza tra l’inizio del processo alla base della malattia e il
suo esordio clinico quando oltre l’85% delle cellule beta pancreatiche sono andate progressivamente distrutte. Un’altra difficoltà
nella ricerca dei fattori ambientali predisponenti deriva dalla natura sostanzialmente non rigenerativa dell’organo bersaglio dell’attacco autoimmune: le cellule beta pancreatiche. Questi fattori
impediscono l’introduzione di test basati sulla somministrazione
e rimozione di potenziali fattori ambientali scatenanti, che hanno
consentito l’identificazione del glutine come fattore ambientale casuale della celiachia.
A dispetto di tali difficoltà è stato da lungo tempo proposto un ruolo
predisponente per specifici RNA virus, in particolare specifici sierotipi di virus Coxsackie, il cui coinvolgimento nella malattia è supportato anche da evidenza più recente (Dotta et al. 2007). Ma lo
spettro dei fattori ambientali coinvolti sia in senso predisponente
che protettivo potrebbe essere più ampio. Nei paesi sviluppati, tra gli
anni ’40 e ’50 c’è stato un netto miglioramento delle condizioni igienico-sanitarie che ha ridotto l’incidenza di alcune malattie, specie
batteriche e parassitarie. È stato quindi ipotizzato, in base alla cosiddetta “teoria igienica” che l’aumentata incidenza di malattia sia
legata alla scomparsa o comunque riduzione d’infezioni protettive.
Inoltre, durante i ~200.000 anni trascorsi dalla comparsa della nostra specie (Francalacci et al., 2013) il nostro sistema immunitario si
è evoluto per far fronte ad infezioni di vario tipo. Questo meccanismo
potrebbe aver contribuito alla selezione di varianti genetiche utili per
le nostre difese contro i patogeni ma attualmente, in un ambiente
molto meno settico, potenzialmente dannose in quanto in grado di
determinare una condizione di iper-attivazione del sistema immune
predisponente nei confronti dell’autoimmunità. Tale modello potrebbe essere supportato da alcune nuove associazioni, come quella tra
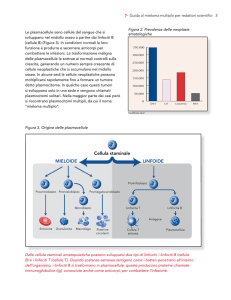
Figura 2.
Rappresentazione grafica dei meccanismi di immunotolleranza coinvolti nell’eziopatogenesi del diabete di tipo 1. Le molecole in nero parteciperebbero all’attivazione delle cellule T mediante il loro recettore (TCR) o con eventi di segnalazione a valle, mentre le molecole in rosso agirebbero
da inibitori di questi processi.
181
M. Pitzalis, M. Zoledziewska, F. Cucca
Figura 3.
Rappresentazione grafica dei loci noti per il diabete di tipo 1 (www.t1dbase.org modificata). In rosso i loci identificati mediante studi di associazione
su tutto il genoma (GWAS). In nero sono stati invece indicati i geni svelati mediante approcci di associazione del gene-candidato.
il DT1 e una specifica variante puntiforme rs3184504, Arg262Trp,
del gene SH2B3, la cui fissazione nelle popolazioni occidentali sembra il risultato di una forte selezione positiva legata all’incremento di
specifiche cellule del sistema immune (Orrù et al. 2013).
Si dovrà comunque tener presente che l’effetto dei geni, anche sommato a quello dei fattori ambientali, non potrà spiegare completamente la malattia, come testimoniato dal fatto che gemelli monozigoti (MZ), geneticamente identici, e che spesso condividono anche
l’ambiente in cui crescono dal concepimento fino all’esordio della
malattia, sono concordanti rispetto alla malattia in circa il 50% dei
casi. Anche considerando modelli murini di malattia, in particolare
il topo diabetico non-obeso (NOD), animali geneticamente identici, che condividono negli stabulari lo stesso ambiente, hanno una
condivisione della malattia inferiore al 100%. Deve esistere quindi
anche una componente stocastica e/o epigenetica in grado di influenzare l’insorgenza della malattia.
Dai geni ai meccanismi
Coerentemente con la natura autoimmune della malattia, la maggior parte dei loci di suscettibilità per il DT1, fino ad ora identificati,
mappa in geni che influenzano la risposta immunitaria. Tali varianti
identificano una seria di vie patogenetiche implicate nello sviluppo
della malattia.
La via patogenetica di maggior rilievo che emerge dagli studi genetici fin qui effettuati riguarda i meccanismi di presentazione antigenica a livello centrale (timo) e periferico (isole pancreatiche e linfo-
182
nodi satelliti) mediata da specifiche varianti alleliche e aplotipiche
codificate a livello della regione HLA-MHC. In particolare, durante
l’induzione intra-timica dell’immunotolleranza le molecole MHC partecipano alla selezione positiva e negativa dei linfociti T attraverso la
presentazione di auto-antigeni espressi nel timo.
Il coinvolgimento della regione HLA nella predisposizione al DT1 fu
dimostrato inizialmente nei primi anni ’70 utilizzando un approccio
del tipo gene candidato e un disegno sperimentale caso-controllo
(Cudworth e Woodrow, 1974). L’elevato grado di correlazione genetica (linkage disequilibrium) delle varianti presenti nella regione
che tende a mantenere insieme nella popolazione specifiche combinazioni di alleli di classe I, III e II ha reso difficoltosa l’attribuzione
della suscettibilità a specifici loci e varianti. Appare ora evidente che
specifici aplotipi codificati dai loci di classe II HLA-DQB1 e -DRB1 e
dai loro ortologhi murini IA e IE, sono primariamente associati con
la malattia e rappresentano la principale componente genetica di
predisposizione nella regione. Un ulteriore contributo è apportato da
una combinazione di geni di suscettibilità, cosiddetti “modificatori”,
localizzati a livello del locus di classe II HLA-DPB1 (Cucca et al.,
2001a) e della classe I (Nejentsev et al., 2007).
L’associazione della regione HLA con la malattia mostra un continuum che va da aplotipi predisponenti ad aplotipi fortemente protettivi.
Uno degli aspetti distintivi del DT1 è rappresentato dai gradi estremi
di resistenza alla malattia conferiti da specifiche varianti alleliche e
aplotipiche, cosiddette protettive (Cucca et al., 1995, Cucca et al.,
2001b). Risultati simili sono stati ottenuti sperimentalmente anche
nel modello murino di DT1: il topo NOD.
Il contributo della genetica alla comprensione delle basi eziopatogenetiche del diabete di tipo 1
L’analisi della struttura proteica codificata dagli alleli MHC di suscettibilità per il DT1 nell’uomo e nel topo ha mostrato rimarchevoli
gradi d’identità. Marcate similitudini sono state osservate anche fra
gli alleli di protezione umani e murini. I meccanismi patogenetici alla
base della resistenza e della suscettibilità alla malattia sono quindi
verosimilmente conservati in specie diverse (Cucca et al., 2001b).
Nel loro insieme i dati suggeriscono un modello patogenetico in
cui la protezione dalla malattia sarebbe direttamente proporzionale
all’affinità ed efficienza del legame fra le molecole MHC, il peptide
diabetogenico e il recettore dei linfociti T (TCR) nel timo (Ridgway et
al., 1998). Le molecole MHC protettive esibirebbero grande affinità
per il peptide diabetogenico e per questa ragione determinerebbero
una selezione negativa, o delezione timica, dei linfociti in quanto se
questi dovessero uscire dal timo sarebbero ad altissimo rischio di
autoreattività (Cucca et al., 2001b). Al contrario, in presenza delle
molecole predisponenti, i linfociti T diabetogenici sfuggirebbero alla
delezione intratimica, in quanto presenterebbero una minore affinità
per il peptide diabetogenico. Le molecole predisponenti sarebbero
comunque in grado di presentare un autoantigene diabetogenico ai
linfociti T autoreattivi circolanti a livello nell’organo bersaglio: le cellule beta pancreatiche.
Le evidenze sperimentali indicano che esisterebbero, inoltre, altri meccanismi mediati da molecole MHC protettive quali la selezione positiva
di linfociti T con funzioni di tipo soppressivo/regolatorio (Luhder et al.,
1998). Esisterebbe quindi un sistema sofisticato con molteplici punti di
controllo dell’autoimmunità: delezione clonale dei linfociti T autoreattivi
nel timo e generazione di risposte regolatorie/soppressive in grado di intercettare eventuali linfociti T autoreattivi presenti nel sangue periferico.
Il DT1 può essere considerato quindi come una malattia causata da un
difetto di questo complesso meccanismo volto al mantenimento della
tolleranza verso uno specifico autoantigene pancreatico.
Numerose evidenze indicano che l’insulina e i suoi precursori rappresentano gli autoantigeni primari nel DT1. Tale modello è supportato anche dai dati genetici. Il gene INS rappresenta il locus di suscettibilità con
più alto effetto dopo la regione HLA e la sua associazione è nota fin dal
1984 attraverso uno studio del tipo gene-candidato (Bell et al., 1984).
Studi successivi hanno evidenziato che tre varianti, rappresentate da
una sequenza ripetuta VNTR e dagli SNP -23HphI (rs689) e +1140A/C
(rs3842753) localizzate a livello del gene INS e in forte linkage disequilibrium tra di loro, sono le più associate con il DT1 nella regione (Barratt et
al., 2004). Sebbene il VNTR rimanga per i suoi potenziali effetti sulla trascrizione del gene il polimorfismo causale più plausibile – con gli alleli
“corti” (di classe I con 26-63 ripetizioni) predisponenti e gli alleli “lunghi”
(di classe III con 140-200 ripetizioni) protettivi – è stato recentemente proposto che anche la variante rs689 abbia importanti effetti sulla
trascrizione del gene INS e possa rappresentare anch’essa la variante
causale (Kralovicova e Vorechovsky, 2010). A prescindere dall’identificazione della specifica variante responsabile dell’associazione con la malattia, esiste una chiara evidenza a supporto del fatto che le tre varianti
predisponenti nei confronti del DT1 correlino con una ridotta espressione della preproinsulina nel timo, che favorirebbe la selezione positiva di
linfociti T autoreattivi, mentre gli alleli protettivi correlerebbero con una
sua incrementata espressione timica che faciliterebbe i meccanismi di
induzione della tolleranza immunologica nei suoi confronti (Pugliese et
al., 1997, Vafiadis et al., 1997).
Altre associazioni genetiche con il DT1 evidenziano ulteriori meccanismi coinvolti nell’eziologia del DT1. In particolare, è stata descritta
(Bottini et al., 2004) e mappata finemente (Zoledziewska et al., 2008)
la variante 1858 C>T del gene PTPN22. Questo ultimo codifica per
una tirosina fosfatasi specifica delle cellule linfoidi (Lymphoid Phosphatase, Lyp) implicata nella regolazione negativa dell’attivazione
T cellulare. La variante 1858 C>T, che rappresenta il terzo segnale
di associazione più forte nel DT1 dopo l’HLA e il gene INS, causa
una sostituzione dell’amminoacido arginina in triptofano nel codone
620 della proteina Lyp (Vang et al., 2005, Bottini et al., 2006), che
dà luogo a una mutazione con acquisizione di funzione con effetto
negativo nei confronti dell’attivazione dei linfociti T.
Come PTPN22 anche i geni CTLA4 e UBASH3A sono importanti regolatori negativi dell’attivazione delle cellule T. CTLA4 codifica per un
corecettore transmembrana espresso sulla superficie dei linfociti T
CD4 e CD8 attivati. Esso ha la funzione d’inibire la proliferazione dei
linfociti T, promuovendone l’apoptosi attraverso l’incremento della
produzione di IL-2. UBASH3A è specificamente espresso nei linfociti
e codifica per una fosfatasi che regola negativamente l’attivazione del TCR. L’allele di rischio per il DT1 (Concannon et al., 2008) è
associato ad acquisizione di funzione e studi sperimentali indicano
che esso incrementi l’espressione di UBASH3A (Dixon et al., 2007).
Appare quindi evidente che nel loro insieme le varianti di rischio per
il DT1 dei geni PTPN22, UBASH3A e CTLA4, determinano, attraverso
un’acquisizione di funzione di un segnale inibitorio, una ridotta attivazione del TCR (Fig. 3). Una possibile spiegazione per questi risultati complicati è legata al ruolo chiave del timo nell’induzione della
tolleranza – dove una ridotta attivazione delle cellule T determinerebbe la maturazione e fuoriuscita dal timo di linfociti autoreattivi e
viceversa un’incrementata attivazione delle cellule T risulterebbe in
una deplezione dei linfociti T autoreattivi --e/o a livello di cellule con
funzioni regolatorie – dove l’iperattivazione si accompagnerebbe ad
un incremento del loro effetto protettivo (Fig. 4).
Tra i loci più recentemente descritti in associazione al DT1 sono presenti geni legati alla produzione di citochine e dei loro recettori: IL2,
IL10, IL27,CCR5, IL2RA e IL2RB (Barrett et al., 2009, Smyth et al.,
2008, Cooper et al., 2008).
Uno dei meccanismi principali nell’eziopatogenesi del DT1 è la regolazione genetica della produzione di IL-2 e del legame con il suo
recettore, codificato da tre geni; IL2RA(CD25), IL2RB, IL2G. Il maggiore
effetto genetico è del locus IL2RA (rischio relativo >3.5) che codifica per la subunità alfa del recettore dell’IL-2. Le varianti alleliche di
suscettibilità al DT1 a livello di IL2RA determinano una riduzione di
una particolare sottoclasse di cellule T memoria caratterizzate da una
marcata espressione della molecola CD25 e dalla mancata espressione del fattore trascrizionale FOXP3 (CD45-, CD25hi non-regulatory T
cells) (Dendrou et al., 2009; Orrù et al. 2013). Queste cellule eserciterebbero il loro ruolo protettivo grazie alla loro capacità di produrre
localmente IL-2 che è a sua volta essenziale per la sopravvivenza e la
proliferazione delle cellule T regolatorie. La carenza di IL-2 causa una
ridotta attivazione delle cellule Treg e conseguente alterazione della
tolleranza periferica. Sono attualmente in corso trial clinici di fase I
e II allo scopo di valutare i rischi e benefici della somministrazione di
basse dosi di IL-2 per indurre/stimolare le cellule Treg nei pazienti DT1
(clinicaltrials.gov/ct2/show/NCT01353833).
Coerentemente con l’ipotesi che gli RNA virus giochino un ruolo
scatenante nella patogenesi del processo autoimmune, un’altra categoria di loci recentemente associati alla malattia comprende geni
che svolgono un ruolo nella risposta immunitaria antivirale: IFIH1
(Interferon Induced with helicase C domain 1) ed EBI2 (Ebstein Barr
Virus induced 2) (Barrett et al. 2009, Heinig et al 2010). Di particolare interesse l’associazione con IFIH1 che codifica per una proteina citoplasmatica in grado di riconoscere l’RNA dei picornavirus
e di mediare l’attivazione della risposta immune anche attraverso
l’incremento dell’espressione delle molecole MHC di classe I a loro
volta essenziali per il riconoscimento e la distruzione, da parte delle
cellule T Citotossiche CD8+, delle cellule infettate dai virus. In parti-
183
M. Pitzalis, M. Zoledziewska, F. Cucca
colare è stata riportata un’associazione negativa con il DT1, e quindi
un effetto protettivo nei confronti della malattia, da parte di varianti
rare di IFIH1 associate ad una perdita di funzione del gene (Nejentsev et al., 2009). Questo indicherebbe che l’iperattivazione di questo
sistema di risposte agli RNA virus, scatenato dalle stesse infezioni
virali, è predisponente nei confronti del DT1.
Conclusioni e prospettive future
Nel corso degli ultimi anni gli studi GWAS hanno migliorato considerevolmente le nostre conoscenze sulle basi genetiche del DT1
dimostrando inequivocabilmente il coinvolgimento di numerosi
geni associati con questa e altre malattie autoimmuni. I prodotti
proteici dei geni associati suggeriscono inoltre alcune delle principali vie patogenetiche implicate nella patogenesi della malattia
e confermano una complessa disregolazione del sistema immune.
Nonostante questi successi, una parte della componente genetica della
malattia rimane ancora inspiegata ed è probabilmente legata a varianti
rare per le quali gli studi fin qui effettuati non possedevano adeguato potere statistico. La dissezione delle componenti genetiche della malattia
potrà essere completata grazie ad un’ancora più elevata “estrazione”
dell’informazione genetica dovuta ai nuovi approcci di sequenziamento
dell’intero genoma, accoppiati ad un ulteriore aumento della casistica
esaminata, nell’ordine di >50.000 pazienti e >50.000 controlli.
Infine, nonostante gli evidenti progressi degli ultimi anni, la nostra visione dei meccanismi implicati nel DT1 è ancora parziale, con molti e
importanti dettagli ancora da delucidare. Infatti, alcune delle varianti
associate con la malattia risultano criptiche, ovvero senza una chiara
correlazione con i meccanismi alla base della patologia in esame. Da ciò
deriva la necessità di integrare i disegni sperimentali esistenti con nuovi
approcci metodologici. Particolarmente promettente appare in tal senso
lo studio combinato di un tratto dicotomico quale il DT1 e di endo-fenotipi potenzialmente correlati, rappresentati da tratti quantitativi misurabili,
quali i livelli delle cellule e delle molecole circolanti del sistema immune.
Figura 4.
Rappresentazione grafica del meccanismo di immunotolleranza nel timo. A livello della corticale timica vengono selezionati i linfociti T in base al
grado di affinità tra il loro recettore (TCR) ed il complesso HLA – autoantigene. I linfociti T con bassa affinità andranno incontro a morte per mancata
stimolazione, mentre quelli che esibiscono media o alta affinità verranno selezionati positivamente. Nella midollare timica avviene una seconda
fase di selezione; in questo processo i linfociti T che esibiscono alta affinità per il complesso HLA – auto antigene moriranno per delezione clonale.
I soli linfociti T che esibiscono media o bassa affinità completeranno il processo di maturazione ed usciranno dal timo.
184
Il contributo della genetica alla comprensione delle basi eziopatogenetiche del diabete di tipo 1
L’identificazione di associazioni genetiche coincidenti, ovvero la stessa
variante implicata sia nel rischio di malattia che nell’influenzare i livelli
di una cellula o molecola, non solo rivelerà meccanismi e vie patogenetiche alla base del DT1 ma consentirà anche l’individuazione di nuovi
bersagli terapeutici per futuri programmi di prevenzione della malattia.
Ringraziamenti
Questo lavoro e dedicato alla memoria del Prof. Antonio Cao e del Prof.
Renzo Galanello.
Box di orientamento
Che cosa si sapeva prima
Il diabete di tipo 1 è una patologia autoimmune multifattoriale complessa. L’eziopatogenesi è definita dall’interazione della componente genetica e della componente ambientale. Fin dal 1970 è noto il forte contributo di varianti alleliche e aplotipiche codificate a livello della regione HLA e definite nel corso degli anni successivi con sempre maggiore risoluzione, insieme ad alcune altre varianti a livelli dei geni INS, PTPN22, CTLA4 e IL2RA localizzate al di fuori della regione HLA.
Cosa sappiamo adesso
Gli studi recenti di associazione su tutto il genoma hanno notevolmente incrementato le conoscenze sui fattori genetici implicati nel diabete di tipo 1.
Attualmente sono noti oltre 50 loci (www.t1dbase.org) di predisposizione alla malattia. Nel loro insieme, i dati genetici esistenti suggeriscono alcuni dei
principali meccanismi coinvolti nella predisposizione e protezione nei confronti del DT1, che sono in parte supportati anche da studi funzionali mirati
sulle cellule primarie (in particolare linfociti T) dei pazienti e su opportuni modelli murini di malattia.
Quali ricadute sulla pratica clinica
La conoscenza approfondita delle basi genetiche del diabete di tipo 1 rappresenta la chiave d’accesso per la definizione dei meccanismi e delle vie coinvolte
nell’eziopatogenesi della malattia. Sebbene sia già possibile selezionare in fase preclinica bambini a forte rischio genetico di sviluppare la malattia, in assenza di
una terapia preventiva efficace le ricadute sulla pratica clinica rimangono ancora limitate. Le future ricadute cliniche includeranno la selezione di bersagli terapeutici
sempre più numerosi e sempre più specifici per la terapia preventiva e la stessa stratificazione degli individui a rischio in base all’assetto genetico individuale.
Bibliografia
Barratt BJ, Payne F, Lowe CE, et al. Remapping the Insulin Gene/IDDM2 Locus in Type
1 Diabetes. Diabetes 2004;53:1884-9.
Barrett JC, Clayton DG, Concannon P, et al. Genome-wide association study and metaanalysis find that over 40 loci affect risk of type 1 diabetes. Nat Genet 2009;41:703-7.
Bell GI, Horita S, Karam JH. A polymorphic locus near the human insulin gene is associated with insulin-dependent diabetes mellitus. Diabetes 1984;33:176-83.
Bottini N, Musumeci L, Alonso A, et al. A functional variant of lymphoid tyrosine phosphatase is associated with type I diabetes. Nature Genet 2004;36:337-8.
Bottini N, Vang T, Cucca F, et al. Role of PTPN22 in type 1 diabetes and other autoimmune diseases. Semin Immunol 2006;18:207-13.
Cooper JD, Smyth DJ, Smiles AM, et al. Meta-analysis of genome-wide association
study data identifies additional type 1 diabetes risk loci. Nat Genet 2008;40:1399-401.
Concannon P, Onengut-Gumuscu S, Todd JA, et al. A human type 1 diabetes susceptibility locus maps to chromosome 21q22.3. Diabetes 2008;57:2858-61.
Cucca F, Dudbridge F, Loddo M, et al. The HLA-DPB1-associated component of the
IDDM1 and its relationship to the major loci HLA-DQB1, -DQA1, and -DRB1. Diabetes
2001a;50:1200-5.
Cucca F, Lampis R, Congia M, et al. A correlation between the relative predisposition
of MHC class II alleles to type 1 diabetes and the structure of their proteins. Hum Mol
Genet 2001b;10:2025-37.
Cucca F, Lampis R, Frau F, et al. The distribution of DR4 haplotypes in Sardinia suggests a primary association of type I diabetes with DRB1 and DQB1 loci. Hum Immunol
1995;43:301-8.
Cudworth AG, Woodrow JC. HLA antigens and diabetes mellitus. Lancet 1974;2:1153.
Dendrou CA, Plagnol V, Fung E, et al. Cell-specific protein phenotypes for the autoimmune locus IL2RA using a genotype-selectabl human bioresource. Nat Genet
2009;41:1011-5.
Dixon AL, Liang L, Moffatt MF, et al. A genome-wide association study of global gene
expression. Nature Genet 2007;39:1202-7.
Dotta F, Censini S, van Halteren AG, et al. Coxsackie B4 virus infection of beta cells and
natural killer cell insulitis in recent-onset type 1 diabetic patients. Proc Natl Acad Sci
U S A 2007;104:5115-20.
Francalacci P, Morelli L, Angius A et al. Low pass DNA sequencing of 1,200 Sardinians
reconstructs European Y chromosome phylogeny. Science 2013:341:565-9.
Heinig M, Petretto E, Wallace C, et al. A trans-acting locus regulates an anti-viral expression network and type 1 diabetes risk. Nature 2010;467:460-4.
Kralovicova J, Vorechovsky I. Allele-speciWc recognition of the 3 splice site of INS
intron 1. Hum Genet 2010;128:383-400.
Lühder F, Katz J, Benoist C, et al. Major histocompatibility complex class II molecules
can protect from diabetes by positively selecting T cells with additional specificities. J
Exp Med 1998;187:379-87.
Nejentsev S, Walker N, Riches D, et al. Rare variants of IFIH1, a gene implicated in
antiviral responses, protect against type 1 diabetes. Science 2009;324:387-9.
Nejentsev S, Howson JM, Walker NM. Localization of type 1 diabetes susceptibility to
the MHC class I genes HLA-B and HLA-A. Nature 2007;450:887-92.
Orrù V, Steri M, Sole G et al. Genetic variants regulating immune cell levels in health
and disease. Cell 2013;155:242-6.
Patterson CC, Dahlquist GG, Gyurus E, et al. Incidence trends for childhood type 1 diabetes in Europe during 1989-2003 and predicted new cases 2005-20: a multicentre
prospective registration study. Lancet 2009;373:2027-33.
Pugliese A, Zeller M, Fernandez Ajr, et al. The insulin gene is transcribed in the human thymus and transcription levels correlated with allelic variation at the INS VNTR_
IDDM2 susceptibility locus for type 1 diabetes. Nature Genet 1997;15:293-7.
Ridgway WM, Ito H, Fassò M, et al. Fathman CG. Analysis of the role of variation of
major histocompatibility complex class II expression on nonobese diabetic (NOD) peripheral T cell response. J Exp Med 1998;188:2267-7.
Smyth DJ, Plagnol V, Walker NM, et al. Shared and distinct genetic variants in type 1
diabetes and celiac disease. N Engl J Med 2008;359:2767-77.
Ueda H, Howson JM, Esposito L, et al. Association of the T_cell regulatory gene CTLA4
with susceptibility to autoimmune disease. Nature 2003;423:506-11.
Vafiadis P, Bennett ST, Todd JA, et al. Insulin expression in human thymus is modulated
by INS VNTR alleles at the IDDM2 locus. Nature Genet 1997;15:289-92.
Vang T, Congia M, Macis MD, et al. Autoimmune-associated lymphoid tyrosine phosphatase is a gain-of-function variant. Nat Genet. 2005;37:1317-9.
Vella A, Cooper JD, Lowe CE, et al. Localization of a type 1 diabetes locus in the
IL2RA/CD25 region by use of tag singlenucleotide polymorphisms. Am. J. Hum
Genet 2005;76:773-9.
Zoledziewska M, Perra C, Orrù V, et al. Further evidence of a primary, causal association of the PTPN22 620W variant with type 1 diabetes. Diabetes
2008;57:229-34.
Corrispondenza
Francesco Cucca, Istituto di Ricerca Genetica e Biomedica (IRGB) del Consiglio Nazionale delle Ricerche (CNR), Cittadella Universitaria di Cagliari 09042 Monserrato CA. Tel.: +39 070 6754543, Fax: +39 070 6754652. E-mail: [email protected]
185