ISTITUTO DI GENETICA DELLE POPOLAZIONI DEL CNR DI ALGHERO
L’ Istituto di Genetica delle Popolazioni del CNR di Alghero, ha come suoi progetti
principali lo studio di malattie genetiche nella popolazione sarda. Questi studi si sono articolati in
due direzioni diverse: studio di pazienti provenienti da tutta la Sardegna e studio di piccoli villaggi
isolati di una regione particolare dell’Isola, l’Ogliastra.
La Sardegna ha una popolazione con particolari caratteristiche genetiche dovute all’isolamento
geografico e al basso tasso di immigrazione che hanno causato una “deriva genetica” che la
differenzia dalle altre popolazioni europee. La sua posizione centrale nel Mediterraneo ha
certamente permesso l’approdo sulle sue sponde di diverse popolazioni in diverse epoche
preistoriche. Un tale fatto giustifica le leggere differenze genetiche che si riscontrano in diverse aree
dell’ isola. Tuttavia la popolazione mostra una sorprendente uniformita` nella frequenza di certi
marcatori genetici quali gruppi sanguigni rari o antigeni HLA che suggeriscono un unico gruppo
comune di “fondatori”. L’isola e` stata molto studiata dal punto di vista genetico soprattutto per
quel che riguarda malattie monogeniche qui particolarmente diffuse quali la talassemia e la carenza
di G6PD. L’osservazione piu` importante e` infatti dovuta al fatto che tutte le malattie monogeniche
studiate in Sardegna sono state associate ad una singola mutazione diffusa in tutta l’isola che porta
ad ipotizzare un effetto fondatore all’interno di una piccola popolazione originale. A differenza
delle malattie monogeniche, le malattie multifattoriali presentano un quadro molto più complesso e
di difficile soluzione.
Alcuni paesi isolati della regione Ogliastra posseggono quelle caratteristiche di isolamento
geografico (vedi foto) e caratteristiche istorico/demografiche tali da renderli dei modelli ideali per
lo studio di malattie multifattoriali. Tali paesi hanno un’origine antica, hanno subito una lenta
crescita senza immigrazioni dall’esterno, hanno vissuto in grande isolamento fino a tempi recenti
con alti tassi di endogamia e di consanguineità. Manifestano quindi per lo meno nelle generazioni
più vecchie quelle caratteristiche di omogeneita` genetica ed ambientale che li rende
particolarmente adatti ad essere usati come modelli per tali studi. Questi studi sono facilitati
dall’omogeneita` ambientale riscontrata in questi paesi dovuta alla mancanza di industrie inquinanti
e al conservatorismo culturale e comportamentale delle popolazioni che hanno mantenuto abitudini
e tradizioni stratificate per eta` e per sesso.
Il coinvolgimento delle popolazioni locali ha favorito la creazione del Parco Genetico
dell’Ogliastra, un Consorzio a cui partecipano oltre ad enti finanziatori privati ed enti pubblici di
ricerca, alcuni paesi Ogliastrini quali Talana e Perdasdefogu.
Il Parco Genetico dell’ Ogliastra (Parco Genos) ha avuto un grande impatto territoriale I
paesi membri hanno infatti creato delle strutture di supporto alla ricerca:
Talana ha allestito un ambulatorio/laboratorio di 70 mq fornito di apparecchiature per la
separazione e conservazione di campioni di sangue (vedi foto) e Perdasdefogu ha allestito un
ambulatorio/laboratorio di 340 mq che da` anche la possibilita` di svolgere programmi di
formazione per giovani diplomati dell’Istituto Tecnico locale, finanziati da fondi regionali. (vedi
foto)
Il progetto di ricerca dell’IGP ha ricevuto una grande attenzione da parte della stampa e delle
televisioni sia nazionali che internazionali attratte dall’idea che l’isolamento geografico potesse
diventare un vantaggio da sfruttare dal punto di vista genetico e che potesse rappresentare per le
popolazioni locali un veicolo di sviluppo culturale e sociale.
Tanta attenzione mediatica ha stuzzicato la curiosità di un potenziale investitore, il Dr. Renato Soru,
che dall’incontro con Mario Pirastu ha fatto nascere la società Shardna Lifesciences. Con l’apporto
di capitali privati, tale società ha potenziato il progetto di ricerca in Ogliastra assumendo medici per
condurre la ricerca epidemiologica sul campo, ingegneri informatici per creare il supporto
informatico necessario alla gestione della enorme mole di dati generati. Un Protocollo di Intesa
redatto con il Consiglio Nazionale delle Ricerche che partecipa come azionista alla Società, ha
sigillato per la prima volta in Italia una partnership mista pubblico/privato. (vedi foto relative alla
firma del documento) Una parte del personale dell’IGP si è pertanto trasferito in comando presso la
società.
PROGETTO OGLIASTRA: Identificazione di Geni associati a Malattie Multifattoriali
attraverso lo studio di isolati genetici
Obiettivi del Progetto:
Studio demografico, epidemiologico e genetico di popolazioni isolate per l’identificazione di geni
associati a malattie multifattoriali comuni e a Tratti Quantitativi ad esse associati come fattori di
rischio.
Introduzione
La ricerca sull’identificazione dei fattori di rischio genetici associati a malattie multifattoriali
comuni si e` negli ultimi anni concentrata sullo studio di isolati genetici per trovare soluzioni ai
problemi posti dalla complessita` generata dalla molteplicita` di variabili coinvolte in tali studi. Si
sono pertanto moltiplicati a livello internazionale gli studi di popolazioni che sono geograficamente
isolate (Iceland, Sardegna, ) o che hanno avuto un’origine ben definita nel tempo (Quebec, Tristan
da Cunha, Hutterites) o che, per isolamento culturale seppur senza isolamento geografico, hanno
mantenuto un certo grado di inbreeding (i.e. Bedouin Arabs, Hasidic Jews). Numerosi geni alterati
sono stati identificati come causa di rare malattie monogeniche grazie a studi condotti in isolati
genetici dove il quoziente di kinship e` piu` alto che in popolazioni aperte ed il Linkage
Disequilibrium si estende su ampie regioni cromosomiche (Puffenberg et al. 1994; Scott et al. 1995;
Hästbacka et al. 1992; de la Chapelle 1998; Peltonen et al. 1999). Nel caso delle malattie
multifattoriali il successo pero` e` stato molto meno eclatante anche perche`, per il mappaggio di
geni associati a malattie complesse, non tutti gli isolati sono equivalenti ed alcune caratteristiche
sembrano essere particolarmente importanti (Peltonen 2000). E` di fondamentale importanza una
conoscenza approfondita della storia demografica e sociale locale e la determinazione del numero di
fondatori, la modalita` di crescita e l’attuale dimensione della popolazione, il tasso di immigrazione,
di endogamia, di consanguineita` e la presenza di deriva genetica (Freimer et al. 1997; Kruglyak
1999; Wright et al. 1999; Risch 2000). Inoltre, in alcune popolazioni l’isolamento geografico ha
prodotto anche una certa omogeneita` culturale che si traduce in conservatorismo nelle abitudini
alimentari e stili di vita. Questa omogeneita` ambientale, pur stratificata per sesso e per eta`,
comporta una riduzione del background “noise” ambientale e quindi facilita l’identificazione di
fattori puramente genetici (Terwilliger and Weiss 1998, Terwilliger and Goring 2000).
La scelta delle popolazioni isolate da studiare deve anche tenere conto della possibilita` di
ricostruire accurate e veritiere genealogie grazie alla disponibilita` di documenti scritti quali i
registri ecclesiastici e demografici. Questa risorsa da` un vantaggio molto significativo nella
pianificazione degli studi genetici (Peltonen et al. 2000). Come sappiamo, la Chiesa Cattolica ha
creato una preziosa fonte di documentazione demografica attraverso la compilazione dei Quinque
Libri in ogni parrocchia del mondo cattolico sin dalla fine del sedicesimo secolo.
Quando l’intera popolazione e` essenzialmente un grande albero genealogico, e` molto probabile
che gli individui affetti abbiano ereditato gli stessi alleli di predisposizione “identici per
discendenza” (IBD) da un antenato comune. Grandi alberi genealogici che includono individui
affetti dalla stessa patologia aumentano il potere dell’analisi di linkage dato che la probabilita` a
priori di condivisione di un segmento genomico e` estremamente bassa nel caso di parenti lontani e
quindi l’evidenza di condivisione IBD indica la presenza di fattori genetici che contribuiscono alla
patologia. L’analisi di aplotipi condivisi IBD in parenti affetti ha quindi un grande potere quando si
utilizzano tutte le informazioni genealogiche disponibili.
In ogni caso, oltre alla ricostruzione genealogica, e` importantissimo stimare accuratamente il
numero di fondatori della popolazione sotto studio. Infatti la probabilita` di identificare varianti
geniche associate a malattie comuni e` maggiore in popolazioni isolate derivanti da pochi fondatori
(10 – 100) non imparentati, perche` si ipotizza che le varianti geniche presenti in un a tale
popolazione siano in numero minore di quelle presenti in popolazioni aperte (Gillian and Todd
2000; Wright et al. 1999; Kruglyak 1999; Bourgain et al. 2001).
Un altro fattore molto importante di cui tenere conto e` la dinamica di crescita della popolazione
(Wright et al. 1999; Kruglyak 1999). Studi condotti attraverso simulazioni computerizzate hanno
dimostrato che una lenta crescita durante la prima fase della formazione della popolazione, aumenta
la perdita di eterogeneita` genetica (Kruglyak 1999). Un lungo periodo di crescita costante seguito
da una espansione recente aumenta il Linkage Disequilibrium nella popolazione mentre
un’espansione iniziale seguita da una lenta crescita lo diminuisce.
In popolazioni “inbred” di formazione abbastanza recente (10-20 generazioni) sara` facilitata
l’identificazione di segmenti “identici per discendenza” (IBD) in individui che manifestano lo
stesso fenotipo vista la diminuita eterogeneita` genetica presente nella popolazione (Houwen et al.
1994; Nikali et al. 1995; Wright et al. 1999; Peltonen 2000).
Lo studio condotto dall’IGP nei villaggi isolati dell’Ogliastra ha applicato le seguenti metodologie
per stabilire quanto tali popolazioni corrispondano ad il modello ideale per essere utilizzate per un
veloce ed economico raggiungimento degli obiettivi della ricerca
Metodologie applicate
Tale studio si è pertanto basato su un approccio multidisciplinare per esaminare:
L’evoluzione demografica dell’isolato e la sua origine che ne possono determinare
l’estenzione del Linkage Disequilibrium presente nella popolazione attuale. Studi condotti
attraverso simulazioni hanno dimostrato che una lenta crescita durante la prima fase della
formazione della popolazione seguito da una espansione recente aumenta il Linkage Disequilibrium
nella popolazione, mentre un’espansione iniziale seguita da una lenta crescita lo diminuisce. Si sono
condotti pertanto studi storici e archivistici sulle popolazioni in oggetto.
Il numero di fondatori. La probabilita` di identificare varianti geniche associate a malattie
comuni e` maggiore in popolazioni isolate derivanti da pochi fondatori (10 – 100) non imparentati,
perche` si ipotizza che le varianti geniche presenti in una tale popolazione siano in numero minore
di quelle presenti in popolazioni aperte. Si sono condotte pertanto analisi delle linee ereditarie
paterne e materne attraverso lo studio di marcatori del cromosoma Y in maschi non imparentati per
parte materna e del DNA mitocondriale in un campione random della popolazione.
La ricostruzione genealogica della popolazione. Quando l’intera popolazione e`
essenzialmente un grande albero genealogico, e` molto probabile che gli individui affetti abbiano
ereditato gli stessi alleli di predisposizione “identici per discendenza” (IBD) da un antenato
comune. Grandi alberi genealogici che includono individui affetti dalla stessa patologia aumentano
il potere dell’analisi di linkage dato che la probabilita` a priori di condivisione di un segmento
genomico e` estremamente bassa nel caso di parenti lontani e quindi l’evidenza di condivisione IBD
indica la presenza di fattori genetici che contribuiscono alla patologia. L’analisi di aplotipi condivisi
IBD in parenti affetti ha quindi un grande potere quando si utilizzano tutte le informazioni
genealogiche disponibili. Si sono quindi raccolti tutti i dati anagrafici per gli ultimi 400 anni
contenuti negli archivi vescovili e negli archivi municipali.
Indagine epidemiologica per identificare le patologie prevalenti nella popolazione isolata
sotto studio, la loro distribuzione all’interno di grandi famiglie estese ed i tratti quantitativi (QT) da
analizzare ad esse associati. Teams di medici conducono visite generali e specialistiche delle
popolazioni oggetto di studio; vengono raccolti campioni di sangue e sono sottoposti ad analisi
ematologiche di routine e specialistiche; vengono raccolti dati storici relativi allo stato di salute
della popolazione negli archivi degli ospedali locali.
Analisi genetica. La selezione degli individui da sottoporre a genotipizzazione e` resa
particolarmente efficiente ed economica attraverso la conoscenza della posizione genealogica di
ciascun individuo all’interno di famiglie estese discendenti da antenati comuni. La genotipizzazione
e` condotta in fasi successive che utilizzano mappe di marcatori polimorfici e SNPs (Single
nucleotide polimorphisms) via via piu` densamente distribuiti sulla piccola porzione del genoma
identificata come associata.
Creazione di un sistema informatico integrato per il trattamento e l’analisi delle
informazioni raccolte
La Base Dati è stata realizzata utilizzando la versione 8.1.7 del sistema di gestione di basi di dati
relazionale Oracle che è attualmente il database più utilizzato dalle compagnie Biotecnologiche. I
dati raccolti, sono di tipo genealogico, genetico, fenotipico e ambientale provenienti dai diversi
villaggi in studio. La raccolta delle informazioni avviene secondo due modalità differenti: sia come
import automatico dei dati da sequenziatore, analisi di laboratorio, dati raccolti sul territorio
(fenotipi, dati anagrafici etc.) che tramite maschere di immissione dati. Sono stati elaborati degli
applicativi che permettono agli utenti di estrarre i dati che soddisfano a criteri da essi impostati per
poi processarli tramite software standard di analisi statistica. Contestualmente si è provveduto alla
progettazione ed implementazione di nuovi algoritmi di analisi maggiormente adatti alle esigenze
determinate dallo studio di popolazioni isolate.
Analisi statistica. Nella ricerca si utilizzano due principali study designs: l’analisi
qualitativa comporta lo studio di un numero anche limitato di soggetti affetti da una data malattia
ma opportunamente relazionati fra loro in famiglie o discendenti da comuni antenati. L’analisi
quantitativa comporta l’analisi di tutta la popolazione e di tutte le famiglie che la compongono e
studia i Tratti Quantitativi (QT). I due approcci comportano percio` una diversa selezione dei
campioni da analizzare e diversi metodi di analisi statistica. Una popolazione omogenea permette di
ridurre la variabilità dei fattori eziologici (sia genetici, che ambientali) e famiglie estese che
comprendano soggetti “geneticamente omogenei” (relativamente alla presenza dello stesso gene di
suscettibilità) risulta in un’analisi più potente. Nell’analisi qualitativa, l’utilizzo di famiglie estese,
con più soggetti affetti implica una maggiore probabilità che vi siano gli stessi geni di suscettibilità
coinvolti (anche se questa condizione dipende dalla frequenza del gene-malattia nella popolazione).
In aggiunta, la scelta delle famiglie per l’analisi di linkage massimizza il numero delle meiosi
informative relative al gene di suscettibilità che deve avere un’alta probabilità di segregare nella
famiglia. Tra i diversi metodi di analisi proposti per l’analisi di fenotipi quantitativi finalizzati alla
identificazione e alla localizzazione tramite linkage di loci di suscettibilità (QTL), il metodo delle
componenti della varianza si sta rivelando un approccio molto potente ed efficace. Nell’analisi dei
tratti quantitativi è possibile considerare tutte le informazioni fenotipiche raccolte a livello di
popolazione utilizzando congiuntamente tutte le informazioni genealogiche contenute nel database.
Il metodo delle componenti della varianza rappresenta un’estensione dei metodi classici che si
basano sulla ripartizione della varianza fenotipica totale in componenti dovute a fattori genetici
(rappresentati dalla componente poligenica) e ambientali, per poterne valutare il loro contributo
relativo. Per ogni posizione del genoma viene valutato se una significativa parte della varianza
fenotipica possa essere attribuita ad un QTL presente in quella data posizione cromosomica. Per
ciascun marker e per ciascuna posizione cromosomica, la varianza fenotipica totale viene suddivisa
nelle seguenti componenti: una componente dovuta ad un gene principale in linkage con il marker
(o i markers, nell’analisi multipoint) studiato, una componente poligenica dovuta ad altri geni non
in linkage con il locus-malattia e una componente ambientale.
Analisi bioinformatica
Con la disponibilità della sequenza del genoma umano è diventato possibile “in silico” la rapida
identificazione di tutti i geni all’interno della regione di interesse seguito dallo screening di
mutazione del gene candidato aiutato dalle informazioni sulla sua struttura genica.
Nella prima fase le sequenze nucleotidiche genomiche della regione in esame, definita dai marcatori
molecolari individuati dall’analisi statistica, sono prelevate dalle varie banche dati (NCBI, Celera
Genomics) e comparate fra loro con l’obiettivo di creare un’unica sequenza di riferimento
(consensus sequence). Una prima analisi bioinformatica permette di identificare e “mascherare” le
sequenze ripetute che rappresentano una considerevole quantità del nostro DNA genomico e che
possono alterare l’attendibilita’ dei risultati.
La sequenza “mascherata” di DNA viene comparata con le sequenze dei geni noti depositate nelle
banche dati con una conseguente identificazione e localizzazione dei suddetti. Seguendo la strategia
del cosidetto “candidate approach”, di tali geni, vengono sequenziate le regioni codificanti e
regolative sia in individui affetti che in individui di controllo.
Risultati raggiunti
Analisi demografiche e ricostruzioni genealogiche: raccolta di dati primari negli archivi comunali
e nell’archivio Vescovile di Lanusei.
Sono stati raccolti tutti i dati contenuti nei Quinque Libri relativi ai paese di Talana, Perdasdefogu,
Urzulei e Triei. Tali dati sono relativi ai battesimi, alle cresime, ai matrimoni, alle morti e allo
“stato delle anime” degli abitanti di questi paesi a partire dal 1590 per Perdasdefogu ed Urzulei, e
dal 1643 per Talana e Triei. I dati di nascita, matrimonio e morte, conservati negli archivi comunali
sin dal 1868, sono stati raccolti fino al 1928 (secondo le regole imposte dalla legge sulla privacy). I
dati relativi alla popolazione attualmente residente nel paese sono stati raccolti oralmente
intervistando i residenti previo consenso informato. In totale sono stati finora inseriti nel database
26.890 individui collegati da 22.300 legami di parentela.
E’ stato creato un database anagrafico, storico e contemporaneo, per la ricostruzione genealogica di
tutta la popolazione. Sono state compilate schede cartacee raccogliendo le informazioni relative agli
atti di battesimo, di matrimonio e di morte per tutti e 3 paesi in studio. I dati raccolti nell’Archivio
vescovile di Lanusei sono ora trasferiti su supporto informatico dai borsisti di Perdasdefogu
utilizzando un software appositamente creato dal personale informatico di SharDNA sul database
Oracle in cui ogni individuo viene identificato attraverso un numero di ID unico generato dal
computer e messo in relazione con gli ID dei genitori. Attraverso questo sistema è possibile
identificare per ogni set di persone prescelte tutti gli antenati da loro condivisi. Il database contiene
sino a questo momento i dati genealogici, sierologici, clinici e genetici dei paesi sinora analizzati. Il
sistema informatico messo a punto garantisce l’anonimato dei partecipanti al progetto dato che i dati
“sensibili” raccolti attraverso le indagini cliniche, genetiche ed epidemiologiche, vengono riferiti
esclusivamente a dei numeri e non ai nomi delle persone. Le identità delle persone vengono criptate
e le relazioni tra gli individui sono elaborate da un piccolo gruppo di persone estranee alla ricerca.
Il database ed il programma PedNavigator messo a punto dalla Società Shardna permettono di
ricostruire in maniera automatica gli alberi genealogici relativi alle persone inserite nel database, di
identificare i potenziali antenati comuni relativi a individui presenti nel database e calcolare quindi
gli step meiotici che li separano, il grado di consanguineità e di inincrocio.
L’analisi degli archivi ecclesiastici e municipali fornisce la possibilià di fare uno studio
demografico e di determinare il ritmo di crescita dei vari paesi. Inoltre l'analisi dei dati archivistici
relativi ai matrimoni permette un calcolo approssimativo del coefficiente di consanguineità della
popolazione dato che la consanguineità degli sposi richiede una dispensa vescovile e pertanto il
grado di consanguineità viene sempre specificato.
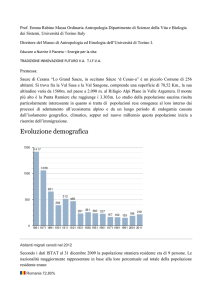
L’analisi demografica è un fondamentale mezzo di conferma che il paese scelto per lo studio abbia
effettivamente tutte quelle caratteristiche che lo rendono assimilabile al modello di popolazione
isolata ipotizzata come ideale per questo tipo di studio.
Creazione di una biobanca e di una seroteca
Con la supervisione dei ricercatori dell’Istituto di Genetica delle Popolazioni del CNR, e con il
personale del Parco Genos è stata creata una biobanca contenente varie aliquote di DNA, plasma,
siero e globuli bianchi di ogni individuo prelevato. Sono stati raccolti e catalogati sinora circa 2800
campioni di sangue da ciascun adulto della popolazione di Talana e di Perdasdefogu. I tecnici di
laboratorio hanno provveduto all’estrazione del DNA dal sangue intero fresco di tutti gli individui
partecipanti al progetto. Le piccole aliquote di plasma, siero e globuli bianchi vengono conservate
nel freezer a –80o C per essere utilizzati in seguito per analisi biochimiche speciali o ulteriori
estrazioni di DNA.
Data l’enorme quantità di aliquote provenienti da ciascun campione accumulate sino ad ora, si sta
provvedendo a razionalizzare ed organizzare la conservazione e la catalogazione. E’ in fase di
elaborazione un sistema di controllo dell’uso di ciascuna aliquota conservata ed un sistema di
“magazzino” per la gestione della Biobanca e della Seroteca.
Su ogni campione di sangue prelevato sono state eseguite le analisi cliniche sierologiche e
l’emocromo in collaborazione con la ASL di Lanusei. Per ogni individuo sono stati valutati i
seguenti parametri: emocromo e formula leucocitaria, azotemia, glicemia, creatininemia, sodiemia,
potassiemia, calcemia ,fosforemia, cloremia, magnesiemia, colesterolemia totale, colesterolo HDL
,colesterolo LDL, trigliceridemia, uricemia, transaminasi seriche (got/ast-gpt/alt), bilirubina totale,
bilirubina diretta, bilirubina indiretta, proteine totali, albumina, fosfatasi alcalina, colinesterasi e
htsh ultrasensibile.
Indagine epidemiologica:
Identificazione di malattie complesse prevalenti nei villaggi ogliastrini prescelti.
E’ stata condotta un’indagine epidemiologica per identificare le malattie complesse presenti sul
territorio. In collaborazione con i medici della Società Shardna, sono state condotte visite generali e
specialistiche nei paesi di Talana, di Perdasdefogu e di Urzulei volte a determinare la prevalenza di
malattie comuni multifattoriali in tali popolazioni.
Sono state compilate schede informative attraverso interviste individuali con i residenti dei paesi
coinvolti.
Screening Generale
condotto sulla popolazione di Talana, di Perdasdefogu e di Urzulei. Nel corso del 2003 il gruppo di
lavoro ha completato 1480 visite a Perdasdefogu, 982 visite a Urzulei mentre a Talana ne sono state
condotte finora 442 e sono ancora in corso.
Sono stati rilevati i seguenti dati
Dati socio-anagrafici
Dati sulle abitudini di vita (principali fattori di rischio)
Dati relativi all’anamnesi personale ed eventuale familiarità
Esame obiettivo con rilevazione delle misure antropometriche (peso, altezza) e di altri tratti
quantitativi (per es. pressione arteriosa)
Analisi chimico cliniche sui campioni biologici raccolti. I campioni di sangue sono stati
sottoposti ad analisi per circa trenta parametri quali l’Emocromo, la Glicemia, l’azotemia, la
creatininemia, l’uricemia, gli elettroliti sierici, i livelli di ormone tiroideo ed il profilo
lipidico.
Gli screening specialistici sono stati effettuati su tutta la popolazione basandosi sulla prevalenza di
alcune patologie. Tali screening specialistici consistono sia di esami clinici che di esami
strumentali.
Screening oculistico
Il progetto di ricerca e` svolto in collaborazione con la Clinica Oculistica dell’Università di
Cagliari. Gli obiettivi generali sono determinare la prevalenza di patologie oculari nella popolazione
di Talana e condurre uno studio dei tratti quantitativi della morfometria oculare. Il disegno dello
studio è di tipo osservazionale trasversale. Nel periodo compreso fra ottobre 2001 e luglio 2002 è
stato effettuato uno screening oculistico sulla popolazione del paese. Lo screening è stato condotto
da due oculisti specialisti che hanno visitato 791 persone, di età compresa fra i 5 e gli 89 anni; tutte
le persone hanno aderito alla ricerca in conformità alle norme etiche sul consenso informato e alla
legge sulla privacy (L. 675/96). La visita oculistica prevede una anamnesi oculare specialistica
personale e familiare, l’esame obiettivo con osservazione biomicroscopica del segmento anteriore e
posteriore dell’occhio, la valutazione dell’acuità visiva e l’esame della refrazione, l’oftalmometria,
la valutazione della motilità oculare (stereopsi), l’esame del senso cromatico mediante test di
Ishihara, la misurazione della pressione intraoculare, l’esame biometrico e la retinografia. Gli
esami strumentali sono stati eseguiti con l’ausilio di: lampada a fessura, ottotipo, autorefrattometro,
tonometro ad applanazione e biometro oculare. Personale che opera sul campo: due oculisti
specializzati.
I tratti della morfometria oculare quali la lunghezza assiale, la profondità della camera, lo spessore
del cristallino e il raggio di curvaturea corneale sono i principali componenti della refrazione
oculare. I vizi di refrazione (miopia, ipermetropia, astigmatismo ecc) sono una manifestazione delle
relazioni che intercorrono fra tali componenti oculari. Sebbene da molti studi risulta che la genetica
giochi un ruolo determinante nella morfometria oculare non sono ancora stati identificati loci che ne
influenzano l’espressione.
Screening dei disturbi cognitivi e della demenza
Per l’identificazione di soggetti con sindrome dementigena al di sopra del 60mo anno di eta` in una
prima fase è stata condotta\ un’ anamnesi specialistica patologica e familiare dei disturbi di memoria
e altri deficit cognitivi. Verrà condotto un esame obiettivo per la valutazione dello stato fisico
generale, un esame neurologico ed una valutazione cognitiva, comportamentale e funzionale globale.
Sui soggetti identificati nella prima fase verrà condotta una valutazione neuropsicologica ed esami
di laboratorio (VES, emocromo con formula, glicemia, ASAT, ALAT, creatinina, azotemia,
elettroliti sierici, Vitamina B12 e folati, TSH, VDRL). Verranno utilizzati inoltre i seguenti Test per
la valutazione dei disturbi cognitivi e comportamentali: Mini Mental State Examination, Activity of
Daily Living, Instrumental Activity of Daily Living, Geriatric Depression Scale, Test dell’Orologio,
Geriatric Depression Scale, Hachinsky Ischaemic Scale, Racconto di Babcock, 15 Parole di Rey,
Fluenza fonetica e semantica, Token test, Matrici attenzionali e numeriche, Copia disegni
Screening per l’osteoporosi
Il progetto di studio sull’Osteoporosi ha l’obiettivo di determinare la prevalenza di tale patologia
nella popolazione di Talana e di quantificarne e caratterizzarne la componente genetica. Il disegno
dello studio è di tipo osservazionale trasversale. Contestualmente alle visite dell’indagine
epidemiologica generale, si conduce una rilevazione strumentale della densita` minerale ossea
tramite un’analisi automatica osteosonometrica ed osteosonografica del tessuto osseo (DBM Sonic
Bone Profiler). Tale esame è utile per l’identificazione di soggetti a rischio di osteoporosi, e inoltre
fornisce il tratto quantitativo densità minerale ossea. Al momento a Talana sono state effettuate 333
misurazioni. Il personale che opera sul campo e`costituito da due medici specialisti e un’infermiera
professionale addestrati all’utilizzo dello strumento.
Screening per l’ipertensione arteriosa essenziale
Questa patologia e` gia` stata individuata nello studio condotto dall’IGP a Talana come prevalente
nella popolazione con un’incidenza del 10%, paragonabile a quella della popolazione caucasica.
Come parte dello studio vengono eseguite visite cardiologiche come parte dello studio
sull’ipertensione a Talana. Gli obiettivi sono di due ordini: approfondire la caratterizzazione
fenotipica dei soggetti che fanno parte dello studio sull’ipertensione e studiare i tratti quantitativi
della morfometria del cuore. Il disegno dello studio è di tipo osservazionale trasversale. Lo studio
prevede di effettuare un elettrocardiogramma (ECG) ed un ecocardiogramma color doppler a tutti
gli abitanti di Talana che partecipano all’indagine epidemiologica generale.
L’ECG intende rilevare: segni di ipertrofia ventricolare sinistra, disturbi della conduzione, disturbi
del ritmo (ovvero ritmi sopraventricolari, ritmi ventricolari e battiti prematuri), sindrome del QT
allungato, alterazioni dell’onda T e del tratto ST. In occasione dell’ECG, ad ogni soggetto viene
anche rilevata la pressione arteriosa seguendo il protocollo delle linee guida suggerite dalla Società
Europea dell’ipertensione.
L’ecocardiogramma viene effettuato in tre modalità. Normale (misurazioni mono e bidimensionali), per rilevare gli spessori e la morfologia del cuore al fine di valutare il rischio
cardiovascolare. Eco doppier che consente di effettuare le misurazioni del flusso trans mitralico con
cui si valutano la funzionalità cardiaca diastolica e i fattori di rischio per l’ipertensione. Eco color
doppler, che mappando le velocità con il colore (parametri di cinetica) consente di valutare la
cinetica del ventricolo sinistro e i fattori di rischio per la cardiopatia ischemica. Al momento le
visite effettuate a Talana sono state 200 e si prevede di terminare alla fine del 2004. Il personale che
opera sul campo e`formato da un cardiologo specializzato per l’ecocardiogramma e due medici
specialisti addestrati per l’ECG.
Screening per la Nefrolitiasi
Anche questa patologia e` gia` stata identificata nello studio preliminare condotto a Talana
dall’IGP. Vengono quindi raccolte anamnesi specialistiche riferite alla patologia nefrolitiasica e
sugli affetti individuati ed i loro familiari vengono condotte le seguenti indagini strumentali:
Ecografia e Radiografia renale, Ecografia renovescicale. Sono raccolte le urine delle 24 ore ed urine
fresche per l’analisi della uricosuria e determinazione del pH urinario. Vengono esaminati gli
eventuali calcoli espulsi dai pazienti e viene esaminato il sedimento urinario. Su un campione di
sangue vengono determinati gli elettroliti sierici e l’uricemia.
Vengono esaminate, con la collaborazione degli uffici di Igiene, le acque delle sorgenti identificate
come fonti di acqua potabile utilizzate dalle popolazioni in esame.
Screening per l’obesita`
Il progetto si propone di studiare le abitudini alimentari della popolazione di Perdasdefogu, per
caratterizzare gli alimenti tipici locali in riferimento ai macro e micronutrienti (per integrare i data
base alimentari nazionali), individuare le associazioni tra obesità e alimentazione e infine
quantificare e caratterizzare la componente genetica dell’obesità. Il disegno dello studio è di tipo
caso controllo e prevede 2 gruppi di casi (soprappeso e obesi) e 1 gruppo di controlli (normopeso).
La selezione del campione da studiare è effettuata tramite una procedura di campionamento
stratificato non proporzionale. La base di campionamento è costituita dalla lista dei 1517 soggetti,
63% dei residenti a Perdasdefogu, di età superiore ai 18 anni compiuti che hanno partecipato
all’indagine epidemiologica svolta da Shardna nel corso del 2002-2003 e dei quali si dispone di un
campione di sangue, del DNA in stock, della valutazione dei principali parametri di assetto lipidico
e del dato sull’indice di massa corporea. La base di campionamento viene suddivisa in tre strati in
base ai valori del BMI che qualificano il soggetto come normopeso (n = 702), soprappeso (n = 545)
e obeso (n = 270). Da ogni strato vengono estratti casualmente 110 individui. Ai soggetti selezionati
viene mandata un lettera che li invita a partecipare allo studio. Tutti i soggetti che aderiscono allo
studio vengono sottoposti al protocollo di indagine che prevede un’anamnesi ponderale, fisiologica,
patologica e farmacologica, la rilevazione delle misure antropometriche per il calcolo del BMI, le
circonferenze del polso, della vita, dei fianchi e dell’addome, un esame strumentale
(bioimpedenziometria) per la misurazione della quantità di massa grassa e massa magra e infine la
compilazione un questionario quali-quantitativo per la valutazione delle abitudini alimentari. Il
questionario comprende due parti: una relativa ad abitudini alimentari generali (quanti pasti
consumano giornalmente, che tipo di acqua bevono, il consumo di spezie ecc) e l’altra comprende
un elenco di circa 270 alimenti (compresi quelli tipici) per i quali è necessario indicare la quantità di
consumo abituale (servendosi di fotografie per le diverse dimensioni della porzione oppure
indicando direttamente la quantità in peso dell’alimento) e la frequenza di consumo (n° volte a
settimana, mese o anno). Al momento a Perdasdefogu sono state effettuate 325 visite ed è in corso
l’analisi dei dati.
Screening per le Tireopatie
La prima fase di screening della popolazione di Talana ha gia` permesso di identificare le tireopatie
come problema molto diffuso nella popolazione. Pertanto, per l’identificazione di soggetti affetti da
tireopatie si conduce un’ anamnesi specialistica ed un esame obiettivo seguito da analisi strumentale
(Ecocolor Doppler del collo) e da un prelievo ematico (Fase I: dosaggio ormonale del TSH,
dosaggio del TPOAb; Fase II: dosaggio delle frazioni libere degli ormoni tiroidei FT3 e FT4; Fase
III: esami di medicina nucleare, esami citologici)
Screening per le cefalee
Il progetto di ricerca viene svolto in collaborazione con il Centro Cefalee dell’Università di Ferrara.
Gli obiettivi sono ricercare i geni rilevanti per la eziopatogenesi dell’emicrania e i geni rilevanti per
la diversa risposta antiemicranica ai farmaci analgesici di tipo antinfiammatorio non steroideo
(FANS). Il disegno dello studio è di tipo osservazionale trasversale. La parte clinica del progetto si
basa sulla caratterizzazione del fenotipo emicranico e nel quadro di questa caratterizzazione clinica
della individuazione del tipo di risposta all’azione antidolorifica dei FANS nell’attacco acuto di
emicrania. La prima fase dello studio rientra nell’indagine epidemiologica generale sulla
popolazione di Perdasdefogu, Talana e Urzulei. Tutti coloro che nelle visite dell’indagine
epidemiologica sono risultati affetti da emicrania costituiscono la base di campionamento per lo
studio sulle cefalee vero e proprio. Una volta identificato un gruppo di pazienti, essi verranno
chiamati per sottoporsi alla visita per la cefalea che ha il fine di diagnosticare l’emicrania e definire
il tipo di risposta ai FANS (ovvero definire se e quanto l’attacco emicranico viene trattato
efficacemente con farmaci di tipo FANS, inclusa l’aspirina). Il ruolo del medico neurologo che
effettua la visita è quello di effettuare l’esame clinico ed anamnestico dei soggetti.
Lo strumento utilizzato nelle visite per la diagnosi di cefalea è l’anamnesi. Si tratta della raccolta
della storia personale della malattia che deve essere eseguita con domande mirate in modo tale che
le risposte fornite permettano di individuare gli aspetti più salienti per la corretta diagnosi di
emicrania. Il cui primo scopo è quello di distinguere una cefalea primaria (tra cui rientra
l’emicrania) da una secondaria espressione di un processo patologico più o meno grave.
I dati anamnestici fondamentali sono: familiarità per cefalea, età di comparsa della cefalea ed eventi
eventualmente associati a tale esordio, durata e periodicità della malattia, durata e frequenza del
singolo attacco cefalalgico, qualità e intensità del dolore, sede del dolore, disturbi che
accompagnano (o eventualmente precedono) il dolore, fattori scatenanti, aggravanti e migliorativi,
trattamenti utilizzati. La visita si conclude con una valutazione neurologica generale (esame della
postura, della forza e del tono muscolare, della sensibilità, dei riflessi e dei nervi cranici) per
escludere la presenza di condizioni patologiche in grado di determinare una cefalea sintomatica. Al
momento sono state effettuate 170 visite a Perdasdefogu, 40 ad Urzulei e 40 a Talana dove sono
tutt’ora in corso.
Studio delle malattie complesse e Tratti Quantitativi
Analisi di aplotipi e livelli circolanti dell’enzima convertitore dell’angiotensina I.
Il sistema Renina Angiotensina (SRA) è ritenuto responsabile del controllo della pressione arteriosa
e del metabolismo del sodio. Il SRA coinvolge principalmente quattro molecole:
l’angiotensinogeno, la renina, l’enzima convertitore dell’angiotensina I e il recettore di tipo I
dell’angiotensina II (codificati rispettivamente dai geni AGT, REN, DCP1 e AGTR1). Il nostro
studio si è occupato in particolare dell’enzima convertitore dell’angiotensina I e dei suoi livelli
circolanti. La variante D del polimorfismo inserzione/delezione del gene DCP1 è stata associata ad
alti livelli di ACE circolante ed è stata collegata ad un incremento del rischio di malattie
cardiovascolari. I dati sinora raccolti non permettono una visione chiara del fenomeno e
suggeriscono che l’associazione tra genotipo ACE e malattie cardiovascolari possa essere
influenzata sia da fattori genetici che da fattori non genetici. Abbiamo studiato alcune varianti
molecolari del gene DCP1 e la concentrazione plasmatica dell’ACE nella popolazione di Talana.
Per ogni campione è stato definito il livello sierico di enzima e sono stati analizzati 10 polimorfismi
biallelici (SNPs), che coprono la regione di nostro interesse. Questi polimorfismi sono stati scelti
sulla base di precedenti studi che ne hanno dimostrato l’associazione con i livelli di ACE circolante.
I livelli di enzima nella nostra popolazione hanno una distribuzione normale e mostrano una
correlazione significativa (P< 0.05) sia con il sesso che con l’età. La distribuzione del Linkage
Disequilibrium (LD) mostra due zone di ricombinazione tra ext1 e lo SNP1 and tra SNP 2 e il 4. Il
valore Massimo di LD (D’> 0.9) è compreso tra gli SNPs 4, 5, 6, 7 e tra l’1 e il 5. L’analisi di
associazione di ogni singolo SNP suggerisce una correlazione tra tutti gli SNPs del gene DCP1 con
la media dell’attività enzimatica dell’ACE circolante. Attraverso l’analisi degli aplotipi abbiamo
ristretto la zona di interesse dallo SNP 4 al 7, dove sono stati identificati due aplotipi comuni DGG2
(50%) e IAA3 (40%), descritti precedentemente in altri gruppi etnici. Abbiamo riscontrato un
valore medi di 26.1 U/l per i livelli di ACE negli omozigoti IAA3, 43.5 U/l negli omozigoti DGG2
e di 34.1 U/l negli eretozigoti IAA3/DGG2. Le medie dei livelli di ACE del genotipo DGA3
combinato con IAA3 e DGG2 mostrano un valore intermedio tra quello osservato tra gli eterozigoti
(IAA3/DGG2) e gli omozigoti (IAA3 and DGG2). La ricombinazione di questi due aplotipi più
comuni ha creato degli aplotipi rari (10%) che differiscono per un solo o per alcuni SNPs inclusi in
piccoli blocchi di Linkage Disequilibrium (LD). L’effetto additivo di questi marcatori può essere
osservato tramite il riarrangiamento degli aplotipi , in cui differenti blocchi di LD, definiti dagli
SNP 6–7 e SNP 8–9, mostrano variazioni nelle medie dei livelli di ACE. I due aplotipi più frequenti
sono stati identificati precedentemente in altre popolazioni ma mostravano effetti differenti
sull’attività enzimatica dell’ACE. Il nostro studio mostra che le differenze nei blocchi di aplotipi
sono responsabili della variazione nell’attività dell’ACE suggerendo che queste regione genomiche
contengano importanti varianti funzionali del gene DCP1.
Identificato un gene di suscettibilità per la nefrolitiasi da acido urico.
La nefrolitiasi da acido urico è una malattia complessa abbastanza frequente che rappresenta il 20%
di tutte le nefrolitiasi. Era da noi stato identificato un locus per la suscettibilità a tale patologia sul
cromosoma 10q21-q22 studiando un piccolo gruppo di pazienti di Talana. Per restringere la regione
e per identificare eventuali geni di suscettibilità abbiamo studiato altri pazienti appartenenti
all’isolato genetico. Consultando diversi data base abbiamo creato una sequenza di consenso ed
abbiamo identificato 36 nuovi SNPs contenuti nei 1.2 Mb della regione critica. Abbiamo quindi
identificato un aplotipo di 200 Kb in linkage disequilibrium (LD) più prevalente nei casi che nei
controlli. Nei controlli che presentavano questo aplotipo il pH urinario risultava essere molto basso
e questo è uno dei fattori di rischio più importanti per la calcolosi renale.
Attraverso l’analisi di sequenza e una ricerca nei database abbiamo potuto stabilire che un nuovo
gene è contenuto in questo intervallo. La caratterizzazione molecolare che è stata condotta usando
analisi di EST e strategie di RT-PCR e RACE, ha rivelato che era composto da 15 esoni che
coprono una regione di circa 280 Kb che per splicing alternativo generano alemno 4 proteine di
407, 333, 462 e 216 aminoacidi. L’ultima proteina è interamente contenuta nel blocco in linkage
disequilibrium associato con la nefrolitiasi.
L’analisi della struttura condotta al computer suggerisce che questa sia una proteina di membrana
con vari siti di glicosilazione nella parte N terminale.
L’analisi mutazionale della proteina ha rivelato una singola variante in un singolo nucleotide che
causa un missenso nell’esone 4 (Ala62Thr) in forte associazione con la nefrolitiasi da acido urico
(p= 0.0096)
L’analisi conformazionale computerizzata ha rivelato che la presenza della Treonina in posizione 62
causa un notevole cambiamento nella struttura secondaria del giro dell’alfa elica suggerendo che
tale mutazione abbia una potenziale ricaduta biochimica.
Analisi della componente lipidica nel siero
Abbiamo ipotizzato che le popolazioni da noi studiate possano essere strumentali all'identificazione
di loci implicati nella regolazione di QTLs come i livelli di lipidi del siero. Il nostro studio è stato
svolto sulla popolazione di Talana su un unico pedigree complesso di 16 generazioni. Il campione
in studio ha incluso 758 oggetti, per i quali sono stati misurati :colesterolo totale (CT), LDL e livelli
HDL. Tutti gli individui analizzati sono stati genotipizzati con 654 marcatori molecolari spaziati
adeguatamente su tutto il genoma. Abbiamo seguito un metodo multi-step basato sull’aumento della
complessità dei pedigree e sulla massimizzazione dell’ereditabilità del tratto all’interno delle
famiglie. Nella prima fase l’analisi di associazione delle variance components è stata condotta
usando 5 grandi famiglie (la più piccola di 32 individui, la più grande di 206 individui) per un totale
di 261 campioni fenotipizzati. I valori di ereditabilità erano tutti altamente significativi (0,40 per
CT; 0,37 per LDL e 0,41 per HDL). Il valore di lod multipoint più elevato è stato localizzato sul
cromosoma 18 (Z=2.07) in associazione con le LDL. La stessa regione è stata trovata egualmente in
associazione colesterolo totale (Z=1.84). Altre regioni che mostravano valori di LOD multipoint > 1
sono stati riscontrati per il cromosoma 2, 4 e 18 per TC e LDL, e per il cromosoma 1, 3 e 19 per
HDL. Fra le regioni candidate descritte in letteratura in associazione con i livelli lipidici il valore
più significativo è stato riscontrato utilizzando una famiglia di 936 individui che ha permesso
l’associazione del colesterolo totale con il cromosoma 19p13.2. In questa regione genomica è
presente il gene candidato LDLR che potrebbe avere un ruolo preponderante nella nostra
popolazione.
Genetica del gusto amaro
L'incapacità di percepire il sapore amaro della feniltiocarbamide (PTC) è una delle caratteristiche
ereditate più studiate in varie popolazioni, ma gli studi genetici sinora pubblicati hanno fornito
risultati inconsistenti e contraddittori. Recentemente, uno studio di linkage ha identificato un nuovo
locus in associazione con la sensibilità al gusto amaro sul cromosoma 5p15 e alcune evidenze
dell’esistenza di un secondo locus sul cromosoma 7q. Abbiamo intrapreso lo studio di questo tratto
ereditabile nella popolazione di Talana. I fenotipi sono stati determinati usando 6 soluzioni scalari
di PTC. L'assegnazione qualitativa della soglia di gusto per la PTC in 280 individui analizzati ha
identificato in questa popolazione il 75% di taster (persone sensibili al gusto amaro) e il 25% di
non-taster (individui insensibili). Abbiamo utilizzato circa 400 marcatori molecolari (microsatellite)
adeguatamente spaziati su tutto il genoma per analizzare 110 individui selezionati. L'analisi
parametrica e non parametrica effettuata con i programmi LINKAGE e GENEHUNTER, ha
mostrato valori statisticamente significativi nella regione cromosomica 7q35 (un peak lodscore per
l’analisi a due punti di 3,03 per il marcatore D7S661 e un lodscore multipoint di 2,77). Il fenotipo è
stato analizzato inoltre come tratto quantitativo utilizzando il programma SOLAR che ha
confermato la localizzazione sul cromosoma 7. Abbiamo ottenuto valori significativi di
associazione con il tratto studiato nella stessa regione del cromosoma 7 precedentemente
identificata con l’analisi parametrica e non parametrica. Questo risultato conferma precedenti studi
che hanno assegnato una associazione tra la sensibilità al gusto amaro della PTC e la regione
cromosomica che circonda l’antigene del gruppo sanguigno Kel.
Bibliografia
Bourgain C, Genin E, Holopainen P, Mustalahti K, Maki M, Partanen J, Clerget-Darpoux F
(2001) Use of Closely Related Affected Individuals for the Genetic Study of Complex
Diseases in Founder Populations. Am J Hum Genet 68:154-159.
de la Chapelle A, Wright FA (1998) Linkage disequilibrium mapping in isolated
populations: the example of Finland revisited. Proc Natl Acad Sci U S A 95:12416-12423
Freimer NB, Service SK, Slatkin M (1997) Expanding on population studies. Nat Genet
17:371-373
Gillian CLJ and Todd JA (2000) Strategies in complex disease mapping. Curr Opin Genet
Develop 10:330-334
Hastbacka J, de la Chapelle A, Kaitila I, Sistonen P, Weaver A, Lander E (1992) Linkage
disequilibrium mapping in isolated founder populations: diastrophic dysplasia in Finland.
Nat Genet 2:204-211
Houwen RH, Baharloo S, Blankenship K, Raeymaekers P, Juyn J, Sandkuijl LA, Freimer
NB (1994) Genome screening by searching for shared segments: mapping a gene for benign
recurrent intrahepatic cholestasis. Nat Genet 8:380-386
Kruglyak L (1999) Prospects for whole-genome linkage disequilibrium mapping of common
disease genes. Nat Genet 22:139 144
Nikali K, Soumalainen A, Terwilliger JD, Koskinen T, Weis-senbach J, Peltonen L (1995)
Random search for shared chromosomal regions in four affected individuals: the assignment of a new hereditary ataxia locus. Am J Hum Genet 56:1088–1095
Peltonen L (2000) Positional cloning of disease genes: advantages of genetic isolates. Hum
Hered. 50:66-75.
Peltonen L, Jalanko A, Varilo T (1999) Molecular genetics of the Finnish disease heritage.
Hum Mol Genet 8:1913-1923
Peltonen L, Palotie A, Lange K (2000) Use of population isolates for mapping complex
traits. Nat Genet 1:182-190
Puffenberger EG, Hosoda K, Washington SS, Nakao K, deWit D, Yanagisawa M,
Chakravart A (1994) A missense mutation of the endothelin-B receptor gene in multigenic
Hirschsprung's disease. Cell 79:1257-1266
Puffenberger EG, Hosoda K, Washington SS, Nakao K, deWit D, Yanagisawa M,
Chakravart A (1994) A missense mutation of the endothelin-B receptor gene in multigenic
Hirschsprung's disease. Cell 79:1257-1266
Risch NJ (2000) Searching for genetic determinants in the new millennium. Nature 405:847856
Terwilliger JD, Goring HH (2000) Gene mapping in the 20th and 21st centuries: statistical
methods, data analysis, and experimental design. Hum Biol 72:63-132
Terwilliger JD, Weiss KM (1998) Linkage disequilibrium mapping of complex disease:
fantasy or reality? Curr Opin Biotechnol 9(6):578-94.
Wright AF, Carothers AD, Pirastu M (1999) Population choice in mapping genes for
complex diseases. Nat Genet 23: 397-404