scienza&medicina
SNPs, GWAS e diagnostica
Nell’ultimo decennio, è diventata sempre più chiara l’esistenza di una stretta associazione tra varianti genetiche e
fattori di rischio per molte malattie complesse. Le varianti genetiche più frequenti sono i polimorfismi a singolo
nucleotide (single nucleotide polymorphism o SNP), ossia
variazioni di singoli nucleotidi in determinati tratti di DNA.
Scoperti negli anni ’80 del secolo scorso, gli SNP non sono
sequenze ripetute (come gli RFLP usati per fare le mappe
fisiche, (vedi Unità 5) e possono trovarsi sia nelle regioni
codificanti del genoma sia in quelle non-codificanti. Se
sono nella sequenza di un gene, non sempre determinano una modificazione della sequenza amminoacidica: dal
momento che il codice genetico è degenerato, gli SNP
sinonimi (synonymous), ossia quelli in cui la sostituzione
del nucleotide codifica per lo stesso amminoacido, porteranno alla sintesi della stessa proteina; solo nel caso di SNP
non-sinonimi (non-synonymous) si avrà sostituzione amminoacidica e perdita di struttura-funzione della proteina.
al rischio di una specifica malattia. In questo modo sono
state trovate, infatti, associazioni tra la frequenza di alcuni
SNP e la degenerazione maculare, il diabete mellito di tipo
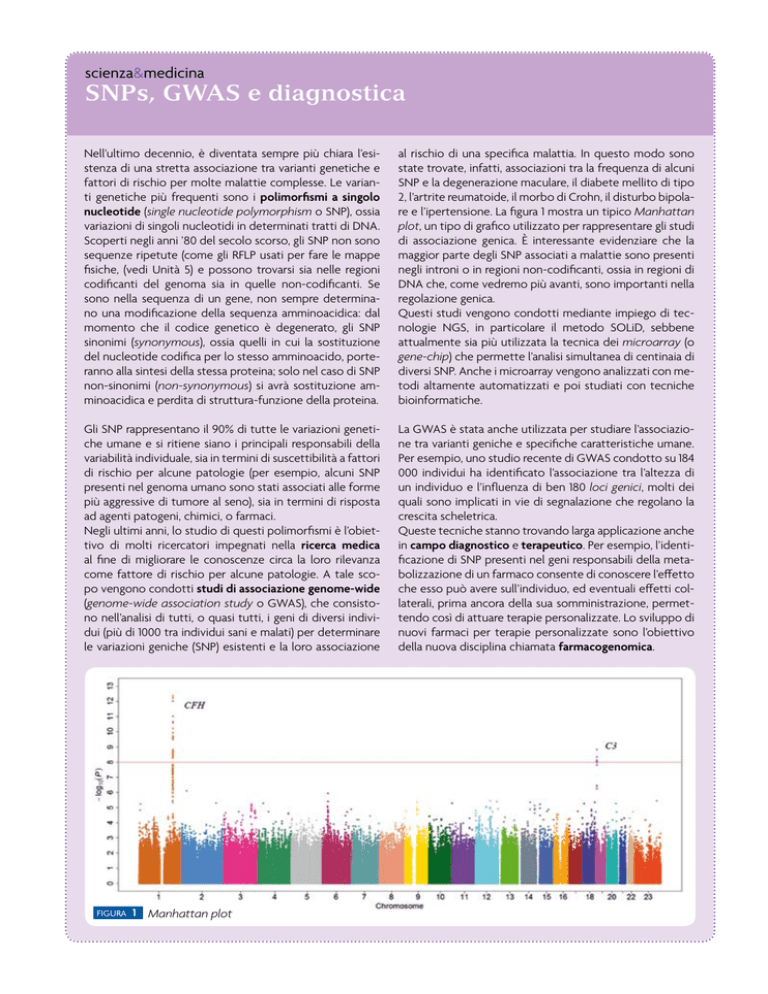
2, l’artrite reumatoide, il morbo di Crohn, il disturbo bipolare e l’ipertensione. La figura 1 mostra un tipico Manhattan
plot, un tipo di grafico utilizzato per rappresentare gli studi
di associazione genica. È interessante evidenziare che la
maggior parte degli SNP associati a malattie sono presenti
negli introni o in regioni non-codificanti, ossia in regioni di
DNA che, come vedremo più avanti, sono importanti nella
regolazione genica.
Questi studi vengono condotti mediante impiego di tecnologie NGS, in particolare il metodo SOLiD, sebbene
attualmente sia più utilizzata la tecnica dei microarray (o
gene-chip) che permette l’analisi simultanea di centinaia di
diversi SNP. Anche i microarray vengono analizzati con metodi altamente automatizzati e poi studiati con tecniche
bioinformatiche.
Gli SNP rappresentano il 90% di tutte le variazioni genetiche umane e si ritiene siano i principali responsabili della
variabilità individuale, sia in termini di suscettibilità a fattori
di rischio per alcune patologie (per esempio, alcuni SNP
presenti nel genoma umano sono stati associati alle forme
più aggressive di tumore al seno), sia in termini di risposta
ad agenti patogeni, chimici, o farmaci.
Negli ultimi anni, lo studio di questi polimorfismi è l’obiettivo di molti ricercatori impegnati nella ricerca medica
al fine di migliorare le conoscenze circa la loro rilevanza
come fattore di rischio per alcune patologie. A tale scopo vengono condotti studi di associazione genome-wide
(genome-wide association study o GWAS), che consistono nell’analisi di tutti, o quasi tutti, i geni di diversi individui (più di 1000 tra individui sani e malati) per determinare
le variazioni geniche (SNP) esistenti e la loro associazione
La GWAS è stata anche utilizzata per studiare l’associazione tra varianti geniche e specifiche caratteristiche umane.
Per esempio, uno studio recente di GWAS condotto su 184
000 individui ha identificato l’associazione tra l’altezza di
un individuo e l’influenza di ben 180 loci genici, molti dei
quali sono implicati in vie di segnalazione che regolano la
crescita scheletrica.
Queste tecniche stanno trovando larga applicazione anche
in campo diagnostico e terapeutico. Per esempio, l’identificazione di SNP presenti nel geni responsabili della metabolizzazione di un farmaco consente di conoscere l’effetto
che esso può avere sull’individuo, ed eventuali effetti collaterali, prima ancora della sua somministrazione, permettendo così di attuare terapie personalizzate. Lo sviluppo di
nuovi farmaci per terapie personalizzate sono l’obiettivo
della nuova disciplina chiamata farmacogenomica.
figura
1
Manhattan plot












