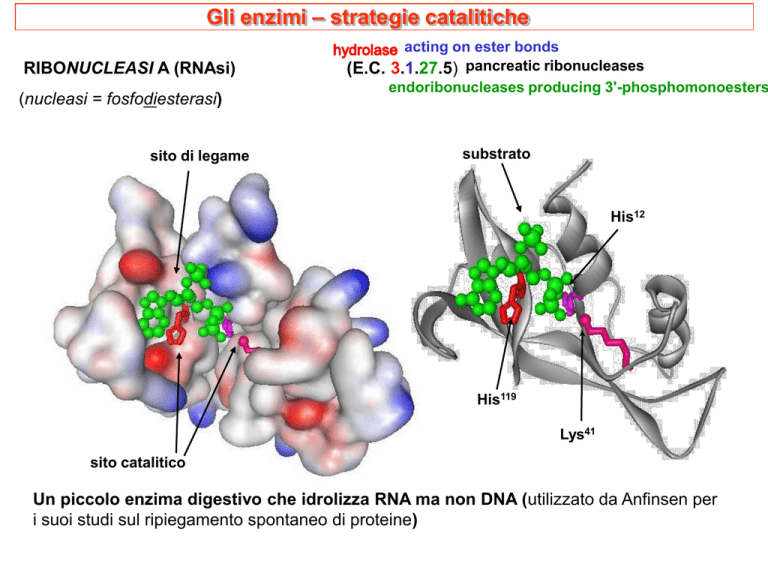
Gli enzimi – strategie catalitiche
RIBONUCLEASI A (RNAsi)
(nucleasi = fosfodiesterasi)
sito di legame
acting on ester bonds
(E.C. 3.1.27.5) pancreatic ribonucleases
endoribonucleases producing 3'-phosphomonoesters
substrato
His12
His119
Lys41
sito catalitico
Un piccolo enzima digestivo che idrolizza RNA ma non DNA (utilizzato da Anfinsen per
i suoi studi sul ripiegamento spontaneo di proteine)
Ribonucleasi - meccanismo d’azione
5’
OC
Lys41
3’
+
OC
2’
H
+
+H
His12
+
(deprotonato)
His119
(protonato)
HO
2'
-
O
O P
O
5'
3'
OC
+
O
-
O
H
LISOZIMA
(una glicosidasi)
(enzima protettivo)
glycosilase
(E.C. 3.2.1.17) lisozyme
glycosidase hydrolysing O- and S-glycosyl bond
Glu35
Asp52
Peptidoglicano batterico
sito di legame
-Nag-Nam-Nag-Nam-Nag-Nam-Nag-
legame
-(1-4)glicosidico
Meccanismo d’azione del lisozima
Meccanismo proposto originalmente
sito catalitico
H
H
H
HO
H2O
O
O
O
O
substrato
O
substrato
distorto
O
O
O +H
O
stato di
transizione (TS)
O
O
analogo stabile del TS
(inibitore)
Meccanismo alternativo
Meccanismo SN2
Meccanismo SN1
CHIMOTRIPSINA
(serina proteasi, endo peptidasi)
acting on peptide bonds (peptidases)
(EC: 3.4.21.1) chymotripsin
serine endopeptidases
(enzima digestivo)
His57
Asp102
triade catalitica
Ser195
Meccanismo d’azione della chimotripsina
sito di legame
Gly193
Gly193
NH
NH
Ser195
cavità
ossianionica
tasca per il legame
specifico del substrato
C
O C
O–H
NH
R-C
NH
O
Ser195
O
HN
Asp102 C
R-C
H
N+
N
O
C
C NH
O
HN
Asp102 C
O
His57
triade catalitica
O
His57
FASE DI ACILAZIONE
nella chimotripsina
la tasca è profonda
ed idrofobica, adatta
ad accettare un residuo
di fenilalanina
FASE DI DEACILAZIONE
Gly193
NH
O
C
C
O
NH
H
NH
C
OH
Ser195
HN
C
His57
R
|
C
O
HN
C
O
C
O C
OH
OH
N
+
Asp102
Asp102
C
H
N
N
O
-O
O
Ser195
O
H
O
NH
NH
His57
Asp102
NH
Ser195
Gly193
Gly193
O
HN
C
O
His57
Le serina proteasi tagliano il legame peptidico dopo specifici residui
Tripsina:
Chimotripsina:
Elastasi:
dopo Arg o Lys
dopo Phe (Trp, Tyr, Leu, Met)
dopo Ala (Val > Ile, Leu, Ser)
Questa specificità deriva dalle caratteristiche delle tasche per il
riconoscimento specifico del substrato
Arg/Lys
AA idrofobici
elastasi
chimotripsina
tripsina
Asp
Ala
Phe
Penicillina - meccanismi d’azione e di resistenza
amoxicillina
penicilline G e V
H20
meccanismo di resistenza basato
sulla degradazione del antibiotico
Cisteina o tiol proteasi
(EC:
acting on peptide bonds (peptidases)
3.4.22.2) papain
cysteine endopeptidases
(endo peptidasi)
PAPAINA
CATEPSINA (digestione intracellulare
di proteine)
Acilazione
Deacilazione
Carbossipeptidasi A
(EC:
acting on peptide bonds (peptidases)
3.4.17.1) carboxypeptidase A
metallocarboxypeptidases
(metallo proteasi, esopeptidasi )
(enzima digestivo)
His196
Zn
His69
Glu72
aromatico o
alifatico grande carbossipeptidasi A
stacca il residuo
C-terminale
basico
carbossipeptidasi B
Meccanismo d’azione della carbossipeptidasi
Asn144
Asn144
O
O
C
C
H2N
Glu270
O
C
C
O-
H2N
Glu270 O
O
C
- +
C
O
HN
OH
HO
C O
H2O
++
Ser197
Zn
His196
Ser197
His69
Glu72
C
HN
His196
O- +
O
HO
HO C
C O
C O
C
-O
Zn++
His69
Glu72
acting on peptide bonds (peptidases)
Proteasi di HIV-1
EC: 3.4.23.16
HIV-1 retropepsin
aspartic endopeptidases
(aspartico proteasi virale, endopeptidasi)
Asp A25
Integrasi
virale
trascrittasi
inversa virale
RNA
virale
DNA
virale
Enzimi
cellulari
DNA GENOMICO
Asp
B25
Proteasi
virale
POLIPEPTIDI
virali
PROTEINE
virali
-NH- -NHH H
\ /
O
Meccanismo d’azione
dell’aspartico proteasi
di HIV
P1 spesso è Phe
P1’ spesso è Pro
P2’ spesso è Glu
H2O
N
HN
HN
O
H
O O
N
H
O
H
HO
O
O O
O O
OH
HN
OH
HN
O
O
O
H
O O
(stato di transizione = gem diolo)
H
O O
O O
Meccanismi d’azione a confronto
serina proteasi
aspartico proteasi
cisteina proteasi
metallo proteasi
Inibitori della HIV proteasi - esempi
Isosteri del legame peptidico
statina
diidrossietilene
ammide ridotto
monoidrossietilene
Saquinavir (1995)
Amprenavir (1999)
Ritonavir (1996)
Fosamprenavir (2003)
Indinavir (1996)
Lopinavir (ABT-378)
Nelfinavir (1997)
Tipranavir (2005)
La progettazione di inibitori di proteasi si basa sulle caratteristiche
P3-P2-P1-P1’-P2’-P3’
1) del sito di legame
(
2) del meccanismo d’azione
( Asp— COOH / Asp—COO -)
3) dello stato di transizione
(
gemdiolo
TS
HN
HO
)
analogo del TS
N
HN
)
OH
HN
O
NH
HO
HN
HO
idrossietilene
1) Criteri per la progettazione di inibitori
- Sostituire il legame peptidico idrolizzato con un isostere non-idrolizzabile
- Modellare la struttura dell’isostere su quella dello stato di transizione
- Ridurre le dimensioni ( < 600 Da)
} aumentare la biodisponibilità
- Ridurre la natura peptidica
2) Criteri per la progettazione di inibitori come farmaci
- ADME (Assorbimento, Distribuzione, Metabolismo, Eliminazione)
- Lipinski’s rule of five (RO5) 1) non più di 5 proton donatori (OH e/o NH)
2) non più di 10 proton accettori (N e/o O)
3) MW nel intervallo 500 - 600 Da
4) Coefficiente di partizione (logPo/w) > 5
octanol
[I]o
Po/w = w
[I]
water
3) STRATEGIE per identificare nuovi inibitori:
- metodi basati sul substrato - utile quando non si conosce meccanismo e struttura
- High Troughput Screening - saggi rapidi e robotizzati su larga scala
- Metodi CADD (Computer Aided Drug Design) - progettazione basata sulla struttura
- Librerie molecolari - sintesi combinatoriale/parallela di molte varianti
TIPI DI COENZIMA
Coenzima
stechiometrico
E1
Coenzima
catalitico
ADP
ATP
CE1
S -P
CE1
-OH
+S
CE1
-OH
+SP
E2
ADP + Pi
ATP
• Un catalizzatore partecipa nella reazione ma non ne viene modificato
• È presente a concentrazioni anche molto inferiori a quelle del substrato
• Il cosubstrato invece deve avere una concentrazione almeno uguale a quella del substrato
• Viene ricostituito ad opera di un altro enzima
Vitamine e Coenzimi – trasportatori attivati
Vitamine idrosolubili trasportatori attivati
Vitamina
coenzima
B3 (niacina)
B2 (riboflavina)
B5 (acido pantotenico)
acido lipoico
B1
B6
biotina
B12
folato
C
NADH, NADPH
FADH2, FMNH2
coenzima A (CoA)
lipoammide
tiammina pirofosfato
piridossal fosfato
biocitina
cobalammina
tetraidrofolato
acido ascorbico
Vitamine liposolubili altri ruoli
Vitamina
forma attiva
A (retinolo)
trans-retinale
K(naftochinone) fillo- e menachinone
D (calciferolo)
ergo- e colecalciferolo
-tocoferolo
E (tocoferolo)
gruppi trasportati/
fonti
modificati
2e- (H-)
carne, veg., uova, lattic.
1 o 2 elatte, uova, verd., fegato
acili
tutti i cibi naturali
Acili
carne, spinaci
aldeidi
carne, veg.,
amminici
carne, veg., uova, latticini
CO2
legumi, cereali, latte, lievito
H, alchilici
carne, pesce, pollame, lattic.
unità monoC
verdure, succhi, lenticchie
riduzione FeIII verdura, frutta
funzione
visione, crescita
coagulazione
pro-ormone
antiossidante
Fonti: http://njms.umdnj.edu/biochemistry/education/bioweb/EnzymeCofactors.htm
http://web.indstate.edu/thcme/mwking/vitamins.html
lipidi animali e vegetali
lipidi animali e vegetali
" e produzione endogena
lipidi animali e vegetali
CN
Co
acido lipoico
dimetilimidazolo
cianocobalamina
tiammina pirofosfato
acido folico
piridossalfosfato
CH3
biotina
retinolo
ascorbato
nella pelle:
luce UV
vitamina D3
deidrocolesterolo
luce UV
nella piante:
ergosterolo
vitamina E
vitamina D2
vitamina K1
Coenzimi – il piridossalfosfato
Il PLP è un coenzima molto versatile che partecipa a diverse reazioni
enzimatiche che spesso coinvolgono amino acidi:
• - e -decarbossilazioni
• reazioni aldol
• transaminazioni
• racemizzazioni
• -eliminazioni
Transaminazione
O
R´
NH2
C
COO - +
H
amminoacido
R C C OO -chetoacido
R´
O C COO - +
-chetoacido
NH 2
R C C OO H
amminoacido
La funzione del PLP è di
1) formare basi di Schiff (aldimine) stabili con gli -ammino gruppi degli AA
2) agire da trappola di elettroni per stabilizzare gli intermedi della reazione
Meccanismo d’azione del piridossal fosfato
aminotrasferasi
E (sito attivo)
Lys
PLP C HO
=
NH 2
OH
P O 4C H 2
+
=
N
C H3
N
R’ C
H
OH
P O 4 C H2
N
O
+
C H2
OH
+ Lys
+ (l’amminoacido
AA1
Lys
- sostituisce
Lys
nel sito attivo )
CH
=
C H3
+ H2O -
H+
C H3
COO R CH
N
- H2O + H+
piridossammina fosfato
(PMP)
( -chetoacido, CA1)
- AA2
H+
H2O
NH2
+
N
H
N
H2O H+
CA1
=
P O 4C H 2
OH
C H3
(viceversa)
+ PMP
O
C OO R C H
N
+ H
C
AA2
C OO -
E
Base di Schiff
CH
AA1
CA2
E-Lys-PLP
E
OH
P O 4C H 2
+
N
H
C H3
Intermedio aldimmina
(resta legato all’enzima mediante
legami non-covalenti)
Coenzimi – la tiammina pirofosfato
decarbossilazione
condensazione
acetaldeide
acetolattato
TPP è un coenzima spesso coinvolto in reazioni del metabolismo dei
carboidrati in cui vengono sintetizzati o scissi legami con atomi di
carbonio carbonilico (aldeidi o chetoni).