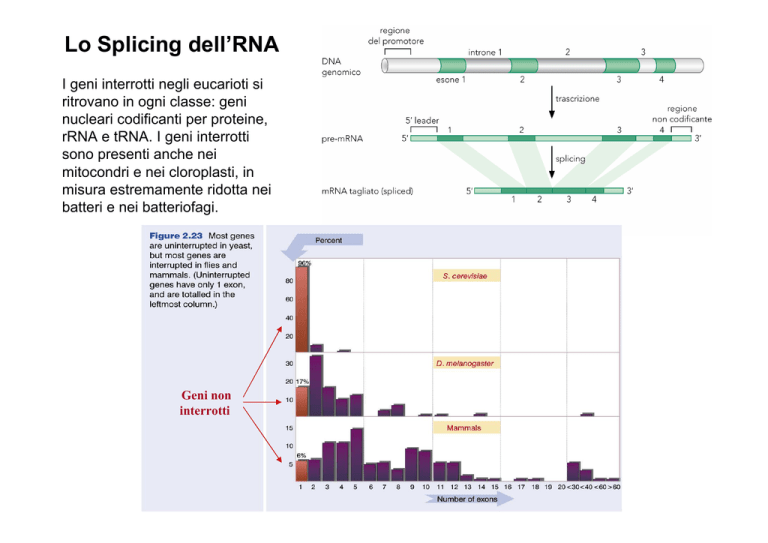
Lo Splicing dell’RNA
I geni interrotti negli eucarioti si
ritrovano in ogni classe: geni
nucleari codificanti per proteine,
rRNA e tRNA. I geni interrotti
sono presenti anche nei
mitocondri e nei cloroplasti, in
misura estremamente ridotta nei
batteri e nei batteriofagi.
Geni non
interrotti
La scoperta dello splicing
Lo splicing fu scoperto nel 1977 attraverso studi di ibridazione DNA-RNA nell’adenovirus.
La comparazione della mappa di restrizione eseguita sul cDNA e quella sul DNA genomico
confermò l’esistenza principalmente nei eucariotici dei geni interrotti.
Caratteristiche generali
1) I Geni sono spaccati più che dispersi
2) Stessa struttura in tutti i tessuti
3) Gli introni presentano codoni di stop in tutti e tre i frames di
lettura (UAA-UGA-UAG)
Gli esoni sono mediamente circa 150 nucleotidi
Gli introni vanno da 200 a 800.000 nucleotidi,
In media circa 1000-3000 basi
Il paradosso del valore-C
Il DNA genomico totale (aploide) caratteristico di
un organismo è detto volore-C. Una relazione
diretta tra contenuto di DNA e complessità
dell’organismo si riscontra fino ai nematodi.
In alcuni phyla evolutivi si hanno grosse
variazioni del contenuto di DNA tra organismi che
mostrano la stessa complessità.
Comparazione mRNA – DNA genomico
Geni molto complessi sono il risultato di numerosi e lunghi introni
e non di prodotti proteici molto lunghi
Species
Average exon N°
Average gene length (Kb)
Average mRNA length (kb)
S. cerevisiae
1
1,6
1,6
Fungi
3
1,5
1,5
C. elegans
4
4.0
D. melanogaster
4
11,3
2,7
Chicken
9
13,9
2,4
Mammals
7
16,6
2,2
10
times
3,0
Il rimescolamento degli esoni
Modello degli introni precoci: perdita degli introni nei batteri
Modello degli introni tardivi: comparsa degli introni
durante l’evoluzione
Probabilmente proteine formate da subunità ripetute come
le immunoglobuline si sono evolute per duplicazione e
divergenza degli esoni
Gli esoni codificano per
domini funzionali
LDL (plasma low density lipoprotein)
EGF (epidermal growth factor).
Organizzazione dei geni per l’actina
Probabilmente alcuni introni sono andati perduti. Il gene primordiale aveva circa 20
introni.
19 differenti posizioni degli
introni in differenti organismi
La Chimica dello Splicing
Lo splicing avviene mediante 2 reazioni di transesterificazione in cui
un gruppo OH libero attacca un legame fosfodiesterico. Le sequenze
più conservate sono: GU (sito 5’), AG (sito 3’) e la A della branch site.
1°Attacco nucleofilo
2°
ofilo
e
l
c
nu
cco
a
t
t
A
degradazione
Teoricamente ogni sito di splicing 5’
potrebbe reagire con qualunque sito
di splicing 3’ ma normalmente lo
splicing avviene tra i siti 5’ e 3’ dello
stesso introne.
Struttura della giunzione a tre vie che si forma durante la reazione di splicing
Punto di
ramificazione
C-2’
Sito 5’ (GU)
C-3’
Nelle due reazioni di
esterificazione non vi è
dispendio di energia.
L’ATP viene poi consumata
nell’assemblaggio e nel
funzionamento del
complesso macchinario di
splicing.
Il Macchinario dello Spliceosoma
U1
snRNA
(Fattori di
splicing)
U2
U5
Un singolo small nuclear RNA (100-300 basi) +
diverse proteine formano lo small nuclear ribonuclear
protein (snRNP,”snurp”). Lo spliceosoma è formato
da circa 150 proteine.
U4/U6
Struttura secondaria del U1 snRNA umano
I domini identificati dalle strutture
stem-loop A,B,C e D forniscono i
siti di legame per proteine
specifiche del U1 snRNP.
Il dominio Sm è richiesto
per l’interazione con
proteine comuni.
La regione a singolo
filamento è
complementare al sito 5’
di splicing dell’introne.
Struttura dell’ snRNA U1
Le interazioni RNA-RNA e RNA-proteina sono fondamentali per il funzionamento
dello spliceosoma
U1 e U6 legano il
sito 5’ di splicing
U2 riconosce il punto di
ramificazione (branch
site)
Branch-point binding protein
Interazione U2-U6
Le fasi della reazione di splicing
U1 lega il sito 5’ mentre il fattore ausiliario
2AF65 contatta il tratto di pirimidine e il sito 3’
di splicing
Complesso E (early)
Legame di U2 con il
sito di ramificazione
(rimozione di BBP)
Complesso A
La A della branch site viene spinta
fuori dal RNA e resa disponibile per la
reazione con il sito 5’ di splicing
Tripla
U6 lega il sito 5’ di
splicing
U4 viene rilasciato,
U6 interagisce con
U2 formando il sito
attivo in cui avviene
la I°reazione di
transesterificazione
(formazione della
lariat)
Complesso C
Complesso B
Avvicinamento dei siti di splicing
U1 viene rilasciato
U5 promuove la II°reazione
di transesterificazione tra i
siti 5’ e 3’ con conseguente
unione dei 2 esoni
Degradazione
dell ’introne
Formazione del centro catalitico
L’appaiamento U6-U4 nella tripla è incompatibile con l’appaiamento U6-U2. U5 è invece legato
alla tripla mediante interazioni proteina-proteina. Il rilascio di U4 permette un cambiamento
conformazionale di U6 dove una parte della sequenza rilasciata forma una forcina mentre
l’altra parte si appaia con una sequenza complementare di U2. Il fattore U2 lega la branch site.
Rilascio U4
↓
Spliceosoma di mammifero contenente U1, U2,
U4, U5, U6 e proteine addizionali (25 nm x 50
nm). Lo spliceosona è equivalente per
grandezza ad una subunità del ribosoma.
Self- Splicing (introni del gruppo I e II)
Splicing nucleare:richiede il complesso apparato di
splicing ed è tipico degli eucarioti.
Introni del gruppo II: la chimica e gli intermedi di
reazione sono simili ai pre-mRNA nucleari. La reazione
produce una lariat (introne a cappio). Si ritrova nei geni
mitoncondriali ed in alcuni geni procariotici.
Introni del gruppo I: un nucleoside libero G con il suo
3’-OH attacca il sito 5’ di splicing. La seconda reazione
procede normalmente ed il sito 3’ dell’esone 1 attacca
sito 3’ di spicing il (5’ dell’ esone 2). L’introne rilasciato è
lineare. Si ritrova nei geni mitoncondriali ed in alcuni geni
procariotici.
La struttura secondaria dell’RNA negli introni di gruppo I
e II è cruciale (non puo’ essere mutata) per la loro
funzione (400-1000 nt). In vitro ad opportune
concentrazione di ioni positivi si osserva self-splicing.
Gli introni del gruppo I possono
formare riboenzimi
La struttura del sito catalitico formata da U6
ed U2 nello spliceosoma è molto simile alla
struttura secondaria intramolecolare che si
forma negli introni di gruppo II.
Affidabilità del sistema di splicing
Meccanismo 1
La subunità maggiore della RNA Polimerasi II
ha una coda (CTD) formata dall’esapeptide
Tyr-Ser-Pro-Thr-Ser-Pro-Ser che va incontro a
fosforilazione. Il grado di fosforilazione
controlla il richiamo di fattori di splicing e della
maturazione dell’RNA.
Un gene umano contiene circa 9 esoni.
Gli esoni sono in media 150 nucleotidi mentre
gli introni (circa 3000 nt) possono arrivare a
molte migliaia di nucleotidi. Di conseguenza gli
esoni sono dispersi tra un oceano di sequenze
introniche.
La RNA Pol II trasferisce i fattori di splicing sul
sito 5’ che poi interagiscono con quelli al sito
3’ successivo appena questo viene trascritto.
Questo meccanismo diminuisce la possibilità
di salto degli esoni.
Meccanismo 2
Le proteine SR (ricche in Ser ed Arg) si legano agli esoni alle sequenze dette ESE (exonic
splicing enhancer) e reclutano alcuni componenti (U1 e U2AF) dello spliceosoma alle
giunzioni 5’ e 3’. Questo meccanismo evita l’uso di siti di splicing non corretti.
Lo splicing alternativo
(troponina T)
Lo splicing alternativo
può generare centinaia
o migliaia di prodotti
proteici differenti (gene
slo di ratto o DSCAM
di Drosophila).
Splicing alternativo
della troponina T,
proteina muscolare di
mammifero.
Le due forme
differiscono per un
esone (3 o 4).
Cinque modi differenti di splicing
alternativo per un mRNA
Lo splicing alternativo costitutivo
Il gene per l’antigene T del
virus SV40 di scimmia
codifica per due proteine:
Large T e Small t che sono
il risultato dell’uso di 2 siti
5’ differenti . Rappresenta
un caso di estensione
dell’ esone.
Si osserva l’estensione dell’esone 1.
Vengono usati i siti di splicing 5’ sst e 3’ SST.
L’ esone 1 viene fuso con l’esone 2.
Vengono usati i siti di splicing 5’ SST e 3’ SST.
Entrambe le forme vengono prodotte in una cellula infettata da SV40 ma elevati livelli di SF2/ASF (SR protein)
favoriscono l’uso dei siti di splicing più vicini e quindi la formazione dello small t
Lo splicing alternativo regolato da attivatori e repressori
Splicing regolato
- Momenti diversi
- Condizioni diverse
- Diversi tessuti o tipi cellulari
Regolatori:
ESE o ISE → exonic-intronic splicing
enhancers
ESS o ISS → exonic-intronic splicing
silencers
Proteine SR
- Il dominio C-terminale
(SR) interagisce con il
macchinario di splicing
- Il dominio RRM
riconosce l’RNA
Determinazione del sesso in Drosophila
Regolazione del gene Sxl
Pm → Promotore mantenimento
Pe → Promotore embrionale
Nei maschi se la proteina Sxl precoce
venisse sintetizzata risulterebbe abortiva
per la presenza di un codone di stop
Nel moscerino il sesso è determinato dal rapporto tra cromosomi X ed autosomi.
Due attivatori SisA e SisB trascrizionali codificati dal cromosoma X stimolano la trascrizione
del gene Sxl (Sex-Lethal).
Un repressore Dpn (Deadpan), codificato da cromosoma 2, inibisce la trascrizione del gene
Sxl.
Negli embrioni future femmine si ha un ‘espressione piu’ elevata degli attivatori SisA-SisB e
quindi della proteina Sxl.
Inizialmente la proteina Sxl precoce
promuove lo splicing alternativo del suo
stesso gene (rimozione dell’introne che
contiene il sito di stop) determinando la
formazione di una proteina Sxl attiva.
Altri due regolatorari Tra e Dsx sono
coinvolti nella cascata regolativa dello
splicing alternativo che determina il
sesso nella Drosophila.
Sxl promuove l’ eliminazione dell’esone
che contiene il sito di stop e favorisce lo
splicing alternativo del gene tra.
A sua volta il regolatore Tra con Tra-2
stimolano lo splicing alternativo del gene
dsx (Double sex).
150 a.a. al CTD
30 a.a. al CTD
Le proteine Dsx che differiscono per il Cterminale nel maschio e nella femmina
controllano i geni del differenziamento
del sesso.