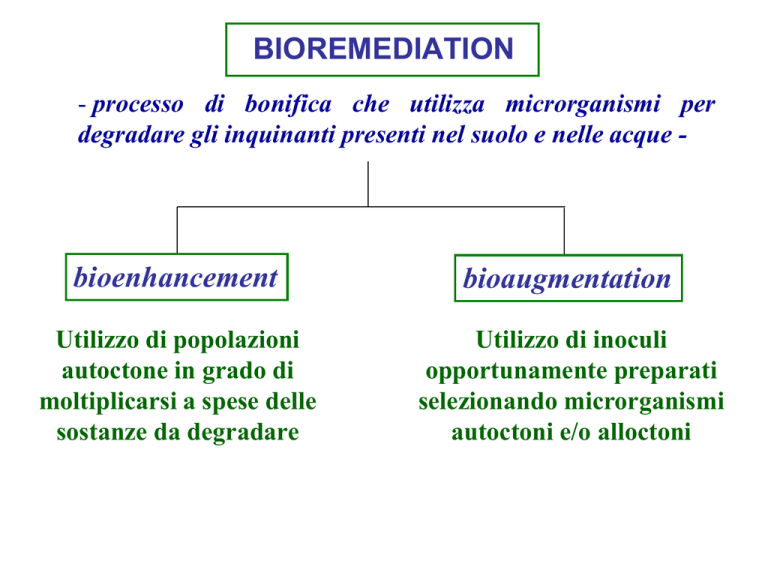
BIOREMEDIATION
- processo di bonifica che utilizza microrganismi per
degradare gli inquinanti presenti nel suolo e nelle acque -
bioenhancement
bioaugmentation
Utilizzo di popolazioni
autoctone in grado di
moltiplicarsi a spese delle
sostanze da degradare
Utilizzo di inoculi
opportunamente preparati
selezionando microrganismi
autoctoni e/o alloctoni
Quando si ricorre alla
bioaugmentation?
1. La popolazione autoctona non è in grado di degradare
composti xenobiotici;
2. gli inquinanti comprendono complesse miscele
contenenti un’ampia varietà di substrati;
3. necesità di aumentare la velocità di biodegradazione
La selezione del microrganismo piu
“adatto”
- molecular breeding
- genetically engineered microrganisms
molecular breeding
- Si applica una pressione selettiva su colture di arricchimento
con lo scopo di accelerare il processo evolutivo che determina
l’istaurarsi di nuovi pathways biochimici ESEMPIO (1):
Coltura di arricchimento in chemostato fornendo piccole
concentrazioni di un composto xenobiotico e alte concentrazioni
di un substrato ad esso correlato ma utilizzabile
Graduale diminuzione del substrato utilizzabile e incremento
del composto xenobiotico
(settimane o mesi)
Selezione di mutanti spontanei con incrementata capacità di
utilizzare il composto xenobiotico
molecular breeding
ESEMPIO (2):
Coltura di arricchimento in chemostato con aggiunta di
microrganismi contenenti plasmidi o trasposoni di
resistenza o di biodegradazione
Lo scambio, la ricombinazione e l’amplificazione
dell’informazione genetica in condizioni di pressione
selettiva, assieme alle mutazioni spontanee o indotte,
favorisce significativamente il processo di evoluzione di nuovi
pathways degradativi
-genetically engineered microrganisms
Ai sensi del D.lgs. 206/01 deve considerarsi geneticamente modificato
qualunque microrganismo (batterio, fungo o virus) il cui materiale genetico
sia stato modificato in modo che non avviene in natura.
Rientrano in questa definizione tutti gli organismi ottenuti tramite
l’introduzione, diretta o indiretta, di materiale genetico preparato in vitro e
che porta alla formazione di nuove combinazioni genetiche ereditabili.
Non sono invece MGM i microrganismi ottenuti mediante mutagenesi,
processi di ricombinazione che utilizzano microrganismi non modificati in
vitro, o fusione di protoplasti purchè ciò avvenga tra microrganismi che,
anche in natura possono scambiare materiale genetico.
-genetically engineered microrganisms
La scelta del microrganismo :
•Caratteristiche metaboliche naturali;
•La capacità di crescere nell’ambiente in cui si vuole
intervenire;
•Disponibilità di opportuni strumenti molecolari che ne
consentono la manipolazione genetica;
•Non patogeno (ovviamente!!)
Il miglioramento genetico di un microrganismo da
utilizzare nei processi di biorisanamento
•trasferimento di geni già esistenti in un ospite eterologo;
•costruzione di nuove vie cataboliche
•modificazione di geni codificanti gli enzimi biodegradativi;
•modificazioni delle proteine regolatrici
•incremento della biodisponibilità degli inquinanti idrofobici.
•Trasferimento di geni già esistenti in un ospite eterologo
ES.1: Pseudomonas putida F1
PROPRIETA’: - degrada benzene, toluene ed etilbenzene
- non patogeno
- utilizzato in numerose applicazioni ambientali
- genoma disponibile in banca dati
OBIETTIVO:
conferire al ceppo biodegradatore la capacità di degradare anche lo
xilene
METODO:
Trasformazione di P. putida F1 con il plasmide catabolico TOL pWW0
oriV
rep
tra
116.580/0
oriT
87.435
pWW0 TOL
29.145
58.290
IS1246
xylR
xylS
IS1246
xyl
xyl
Figura 6.15: Mappa genetica del plasmide pWW0 TOL, isolato da Pseudomonas putida mt-2. Il cerchio interno indica le
dimensoni del plasmide in kilobasi. Il cerchio esterno mostra la posizione di alcuni locus. In verde è mostrata la regione
catabolica coinvolta nella biodegradazione di toluene, xilene e dei loro prodotti di ossidazione. I geni codificanti gli enzimi
coinvolti nella via di degradazione sono organizzati in due operoni, xylUWCMABN e xylXYZLTEGFJQKIH, che sono
positivamente regolati dai prodotti dei geni xylS e xylR. In blu: sequenze di inserzione IS1246. In rosso: i geni coinvolti nel
trasferimento coniugativo del plasmide (traABC) e l’origine di trasferimento (oriT). In verde scuro: il sito di origine della
replicazione (oriV) e un gene coinvolto nel processo replicativo (rep). Le frecce indicano la direzione della trascrizione.
•Trasferimento di geni già esistenti in un ospite eterologo
ES.2: Pseudomonas putida F1
OBIETTIVO DEL MIGLIORAMENTO:
Costruire un ceppo di Pseudomonas capace di degradare
contemporaneamente miscele di BTEX e stirene (feniletene)
METODO:
Trasformazione di P. putida F1 con due plasmidi:
- pWW0 TOL (degradazione dello xilene)
- pVAD (degradazione dello stirene)
A
B
styA
//
styB
styC
styD
//
Figura 6.16: A. Degradazione dello stirene attraverso la via dell’acido fenilacetico. Il processo di degradazione è diviso in
due fasi. Nella prima fase (upper pathway), lo stirene è convertito in acido fenilacetico. Sotto ad ogni reazione è indicato
l’enzima che la catalizza: StyAB, stirene monoossigenasi; StyC, epossistirene isomerasi; StyD, fenilacetaldeide
deidrogenasi. Gli enzimi Paa sono responsabili del catabolismo dell’acido fenilacetico ad intermedi del ciclo di Krebs
(lower pathway). B. Organizzazione genetica dell’operone styABCD in Pseudomonas sp., ceppo Y2.
Referenza :
Paloma Lorenzo, Sergio Alonso, Ana Velasco, Eduardo Díaz, José L. García and Julián Perera.
Design of catabolic cassettes for styrene biodegradation.
Antonie Van Leeuwenhoek. 2003;84(1):17-24
XbaI
HindIII
Ptac
XbaI styA
styB styC
styD
HindIII
XbaI
styB
Ptac
TcR
lacIq
pVLT31
(10 kb)
styA
XbaI + HindIII
styC
styD
HindIII
lacIq
pVAD
(14.1 kb)
TcR
Figura 6.17: Schema di costruzione del plasmide pVAD. Una cassetta “sty”, amplificata per PCR dall’operone styABCD
di Pseudomonas sp. Y2 e contenente all’estremità i siti di riconoscimento degli enzimi di restrizione XbaI e HindIII, è stata
clonata nel plasmide pVLT31 digerito con gli stessi enzimi di restrizione. Nel plasmide risultante, pVAD, l’perone
styABCD è sotto il controllo del promotore Ptac, un promotore ibrido, derivante dalla combinazione dei promotori trp
(triptofano) e lac (lattosio) di E. coli, con caratteristiche di promotore forte e regolabile (represso dal repressore di lac e
derepresso dall’ isopropil-β-tiogalattoside, IPTG, molecola sintetica con struttura simile al lattosio, induttore dell’operone
lac); lacIq: forma mutante del gene lacI di E. coli, che produce livelli elevati del repressore di lac; TcR: gene conferente
resistenza alla tetraciclina.
A
cromosoma
pVAD
E. coli S17-1 pir (pVAD)
E. coli S17-1 pir (pVAD)
cromosoma
P. putida F1
B
cromosoma
P. putida F1 (pVAD)
pWW0
P. putida KT2442 (pWW0 TOL)
P. putida KT2442 (pWW0 TOL)
cromosoma
pVAD
P. putida F1 (pVAD)
P. putida F1 (pVAD, pWW0 TOL)
Figura 6.18: Isolamento del ceppo di P. putida F1 (pVAD, pWW0 TOL). A. Il plasmide pVAD (Figura. 6.17) è stato
trasferito, mediante coniugazione, dal ceppo donatore E. coli S17-1 pir (pVAD) al ceppo ricevete P. putida F1. I
transconiuganti, riceventi il plasmide pVAD, sono stati selezionati in un terreno di coltura contenente tetraciclina. B. Il
plasmide coniugativo pWW0 TOL (Figura. 6.15) è stato trasferito, mediante coniugazione, dal ceppo donatore P. putida
KT2442 (pWW0 TOL) al ceppo ricevente P. putida F1 (pVAD). I transconiuganti sono stati selezionati per la loro
capacità di crescere su terreno contenente tetraciclina (resistenza conferitagli dal plasmide pVAD) e m-xilene come unica
fonte di carbonio (capacità catabolica conferitagli dal plasmide pWW0 TOL).
Il miglioramento genetico di un microrganismo da
utilizzare nei processi di biorisanamento
•trasferimento di geni già esistenti in un ospite eterologo;
•costruzione di nuove vie cataboliche
•modificazione di geni codificanti gli enzimi biodegradativi;
•modificazioni delle proteine regolatrici
•incremento della biodisponibilità degli inquinanti idrofobici.
•Modificazione di geni codificanti enzimi biodegradativi
OBIETTIVI DEL MIGLIORAMENTO:
- aumentare la specificità di substrato;
- migliorarne l’attività enzimatica (aumento della velocità di
degradazione)
- aumentare il livello di espressione dei geni codificanti le
attività enzimatiche
METODI MOLECOLARI:
- scambio di geni codificanti subunità omologhe dello stesso
enzima, provenienti da specie correlate;
- mescolamento di porzioni di geni correlati;
- mutagenesi sito-diretta
•Modificazione di geni codificanti enzimi biodegradativi
ES.1: citocromo P450CAM di Psedomonas putida
-Monossigenasi con elevato potere ossidoriduttivo;
-Catalizzano idrossilazione di composti alifatici,dealchilazione
e dealogenazione;
-Distribuzione ubiquitaria;
-Identificate diverse centinaia;
-CAM: substrato naturale è la “canfora”.
OBIETTIVO DEL MIGLIORAMENTO:
- aumentare la specificità di substrato;
METODO:
- mutagenesi sito diretta: sostituzione, nel sito attivo, della
tirosina in posizione 96 con un’alanina
•Modificazione di geni codificanti enzimi biodegradativi
“La nuova variante dell’enzima è
in grado di ossidare il dimetilmetano, un substrato non
riconosciuto
dalla
forma
selvatica”
Figura 6.19: Struttura del citocromo P450CAM. Sono mostrati i residui aminoacidici del sito attivo: V, valina; T, treonina; I,
isoleucina; F, fenilalanina; G, glicina; L, leucina. Il numero vicino a ciascun aminoacido indica la sua posizione nella
sequenza aminoacidica. La sostituzione del residuo di tirosina in posizione 96 (Y96, evidenziato in rosso) con un’alanina,
cambia la specificità di substrato dell’enzima.
Referenza: Kellner DG, Maves SA, Sligar SG.
Engineering cytochrome P450s for bioremediation.
Curr Opin Biotechnol. 1997 Jun;8(3):274-8. Review.
PMID: 9206006
•Modificazione di geni codificanti enzimi biodegradativi
ES.2: operone bph di Pseudomonas sp. KKS102
- degradazione dei composti fenilici
OBIETTIVO DEL MIGLIORAMENTO:
- aumentarne la capacità di degradare il bifenile e alcuni
policlorobifenili (PCB);
METODO:
- sostituzione del promotore regolato (pE) con un promotore
costitutivo
•Modificazione di geni codificanti enzimi biodegradativi
B. Presenza di bifenile
A. Assenza di bifenile
pE O
bphS
tpnBPH
X
bphE
pE O
//
bphS
tpnBPH
bphE
operone bph
//
operone bph
X
BphS
BphS
HOPDA
Figura 6.20: Regolazione trascrizionale dell’operone bph. A. In assenza di bifenile, il prodotto del gene bphS, la proteina
regolativa BphS, lega il sito O (in giallo) immediatamente a valle promotore pE (in blu), bloccando la trascrizione. B. In
presenza di bifenile, l’ HOPDA (2-hydroxy-6-oxo-6-phenilhexa-2,4-dienoic acid), un intermedio catabolico del bifenile,
blocca l’attività del repressore BphS, permettendo la trascrizione dell’operone. bphE: il primo gene dell’operone bph;
tnpBPH: gene codificante una trasposasi.
Il miglioramento genetico di un microrganismo da
utilizzare nei processi di biorisanamento
•trasferimento di geni già esistenti in un ospite eterologo;
•costruzione di nuove vie cataboliche
•modificazione di geni codificanti gli enzimi biodegradativi;
•modificazioni delle proteine regolatrici
•incremento della biodisponibilità degli inquinanti idrofobici.
Biosintesi dei ramnolipidi in Pseudomonas aeruginosa
Fig 6.8: Biosintesi dei ramnolipidi in P. aeruginosa. La timidina.difosfo ramnosio (TDP-ramnosio) agisce da donatore del ramnosio in due
reazioni sequenziali catalizzate dalla ramnosiltransferasi 1 e ramnosiltransferasi 2. Nelle due reazioni prima il 3-idrossidecanoil-3idrossidecanoato, e poi l’ L-ramnosil-3-idrossidecanoil-3-idrossidecanoato, agiscono da accettori del ramnosio.
Ref.: Ochsner UA, Fiechter A, Reiser J.
Isolation, characterization, and expression in Escherichia coli of the Pseudomonas aeruginosa rhlAB genes encoding a rhamnosyltransferase
involved in rhamnolipid biosurfactant synthesis.
J Biol Chem. 1994 Aug 5;269(31):19787-95.
•Incremento della biodisponibilità degli inquinanti idrofobici
ES.1: cluster genico rhlABRI di Pseudomonas aeruginosa PG201
-Biosintesi del ramnolipide 2
-rhlAB: ramnosiltransferasi 1, complesso enzimatico di membrana
(RhlA, localizzazione periplasmatica; RhlB, componente catalitica
nello strato fosfolipidico)
-rhlRI: regolazione trascrizionale (RhlR, regolatore trascrizionale;
RhlI, sintesi dell’acilomoserinlattone);
-Regolato da segnali ambientali: carenza di azoto e ferro,
incremento della concentrazione cellulare (quorum sensing)
OBIETTIVO DEL MIGLIORAMENTO:
- superare i limiti della complessa regolazione ambientale;
METODO:
- clonaggio dei geni rhlAB in un plasmide ricombinante, sotto il
controllo del promotore Ptac
•Incremento della biodisponibilità degli inquinanti idrofobici
Figura 6.23: Mappa genetica del plasmide pUO98. I geni coinvolti nella biosintesi del ramnolipide 2 (rhlAB) sono sotto il
controllo del promotore inducibile Ptac. lacIq: forma mutante del gene lacI di E. coli, che produce livelli elevati del
repressore di lac; Sm, resistenza alla streptomicina; rep/mob: regione coinvolta nei processi di replicazione e mobilizzazione
del plasmide. Sotto la mappa è indicata l’esatta fusione del promotore tac con i geni rhlAB. S/B, sequenza Shine-Dalgarno.
Referenza:
Ochsner UA, Reiser J, Fiechter A, Witholt B.
Production
of
Pseudomonas
aeruginosa
Rhamnolipid
Biosurfactants
in
Heterologous
Hosts.
Appl Environ Microbiol. 1995 Sep;61(9):3503-3506.
PMID: 16535135
Tabella 1: Attività ramnosiltrasferasica e formazione di ramnolipidi nei ceppi ricombinanti che contengono i geni
rhlAB nel plasmide pUO98 sotto il controllo del promotore tac.
Referenza: Ochsner UA, Reiser J, Fiechter A, Witholt B.
Production
of
Pseudomonas
aeruginosa
Rhamnolipid
Biosurfactants
in
Heterologous
Hosts.
Appl Environ Microbiol. 1995 Sep;61(9):3503-3506.
PMID: 16535135












