Trasporto degli elettroni e
fosforilazione ossidativa
La fosforilazione ossidativa rappresenta il culmine del metabolismo energetico negli
organismi aerobici
Tutte le tappe enzimatiche della degradazione ossidativa dei carboidrati, degli acidi
grassi e degli amminoacidi convergono nella tappa finale della respirazione cellulare,
in cui l’energia prodotta da tali ossidazioni viene utilizzata per la sintesi dell’ATP
Negli eucarioti la fosforilazione ossidativa avviene nei mitocondri e comporta la
riduzione dell’O2 ad H2O. Gli elettroni vengono forniti dal “potere riducente” di
NADH + H+ e FADH2
La trasduzione energetica operante nella fosforilazione ossidativa si spiega con la
formazione di gradienti protonici transmembrana, secondo la teoria
chemiosmotica di Mitchell (Peter Mitchell, 1961)
Il flusso di elettroni “accoppiato” con la sintesi di ATP avviene nella membrana
mitocondriale interna, che può contenere anche più di 10000 gruppi di trasportatori
di elettroni (o catene respiratorie) e di complessi di ATP sintasi
Anatomia biochimica di un mitocondrio
Le
involuzioni
(creste)
aumentano
considerevolmente l’area della superficie della
membrana interna
La membrana mitocondriale esterna è facilmente
permeabile a piccole molecole (Mr < 5000)
ed agli ioni, che l’attraversano grazie a canali
denominati “porine”
La membrana mitocondriale interna, invece, è
impermeabile alle piccole molecole ad agli ioni,
compresi i protoni (H+)
Gli elettroni entrano in una “catena respiratoria
mitocondriale”, nella quale passano tra proteine
(per lo più integrali) di membrana, mediante: 1)
trasferimento diretto (come nella riduzione di
Fe3+ a Fe2+); 2) trasferimento di un atomo
d’idrogeno (H+ + e-); 3) trasferimento di uno
ione idruro (:H-)
La maggior parte degli elettroni che entrano nella catena respiratoria deriva
dall’azione delle deidrogenasi dipendenti da nucleotidi nicotinammidici
(NAD(P)H + H+) e delle flavoproteine contenenti un cofattore flavinico
(FMNH2 o FADH2)
Coenzima Q
Gli elettroni passano poi attraverso una serie di trasportatori legati alla membrana, tra
cui l’ubichinone (detto anche coenzima Q o Q) e due tipi diversi di proteine contenenti
ferro (i citocromi e le proteine ferro-zolfo)
L’ubichinone è un benzochinone liposolubile con una lunga catena laterale isoprenoide
Nei cloroplasti delle piante si trova il plastochinone, mentre nei batteri si trova il
menachinone
La riduzione completa dell’ubichinone richiede due elettroni e due protoni e può
avvenire con un radicale intermedio semichinonico
I Citocromi
I citocromi sono proteine con un’elevata capacità di assorbire la luce visibile, dovuta ai loro gruppi prostetici eme
(contenenti ferro). Nei mitocondri vi sono citocromi a, b e c
Ogni eme possiede
quattro anelli a cinque
membri, contenenti
azoto, disposti in una
struttura ciclica
detta porfirina
La ferro
protoporfirina IX
si trova nei citocromi
b ed anche nella
mioglobina e
nell’emoglobina
L’eme A ha una lunga
coda isoprenoide legata
ad un anello
L’eme C è legato covalentemente
alla proteina
del citocromo c mediante ponti
tioetere con due residui di Cys
I quattro atomi di azoto sono
coordinati con un atomo centrale
di Fe, che può essere nel suo stato
ridotto (Fe2+) od ossidato (Fe3+)
I legami doppi coniugati della porfirina (in
rosa nelle strutture precedenti) sono
responsabili dell’assorbimento della
luce visibile
Ogni citocromo ha tre bande di
assorbimento della luce visibile nel suo
stato ridotto (Fe2+)
La banda γ, nella regione blu dello spettro,
è anche detta “banda di Soret” ed è
utilizzata spesso per descrivere
l'assorbimento di molecole contenenti
eme
I citocromi a e b sono proteine integrali
della membrana mitocondriale interna,
mentre il citocromo c è solubile e si
lega alla superficie esterna della stessa
membrana mediante interazioni
elettrostatiche
Le proteine ferro-zolfo
Nelle proteine ferro-zolfo il metallo è associato ad atomi di zolfo inorganico
o ad atomi di zolfo di residui di Cys della proteina
Tali centri ferro-zolfo possono essere molto semplici (a) o molto complessi (b, c)
Le proteine ferro-zolfo di Rieske vedono il ferro coordinato con
due residui di His e non di Cys
Tutte le proteine Fe-S partecipano a reazioni redox in cui si trasferisce un elettrone alla
volta, sfruttando cambiamenti dello stato di ossidazione del Fe
Potenziali di riduzione standard della catena
respiratoria e dei suoi trasportatori di elettroni
I supercomplessi o respirasomi
Inibitori del trasporto e- e della fosforilazione ox
Complessi proteici della catena di trasporto
Vi sono quattro componenti proteici distinti (I-IV) nella catena di trasporto
degli elettroni mitocondriale
I: NADH-Coenzima Q reduttasi
II: Succinato-coenzima Q reduttasi
III: Coenzima Q-citocromo c reduttasi
IV: Citocromo c ossidasi
Panoramica dei complessi e delle vie nella catena
di trasporto elettronico mitocondriale
Il complesso I ossida il NADH e riduce il Coenzima Q
Il complesso II ossida il succinato e riduce il coenzima Q
La prima tappa della β ossidazione degli acidi grassi è catalizzata dalla flavoproteina acil-CoA
deidrogenasi, che non cede elettroni al Complesso II, ma alla flavoproteina ETF, che
a sua volta li cede alla EFT:Q ossidoreduttasi, la quale riduce il coenzima Q
Anche il glicerolo 3-fosfato “salta” il Complesso II, cedendo elettroni direttamente al coenzima Q
attraverso la glicerolo 3-fosfato deidrogenasi
Il complesso III media il trasporto
degli e- dal coenzima Q al citocromo c
Il complesso III media il trasporto
degli e- dal coenzima Q al citocromo c
Il complesso IV trasferisce gli e- dal cit c
per ridurre l’O2 sul lato della matrice
La citocromo ossidasi trasferisce in
modo ordinato gli elettroni da 4
molecole
di
citocromo
c
all’ossigeno molecolare (O2), che
viene ridotto a 2 molecole di H2O
utilizzando 4 e- e prelevando 4H+
“substrato”
dalla
matrice
mitocondriale
Contestualmente, l’enzima pompa
4H+ dalla matrice verso lo spazio
intermembrana,
aumentando
ulteriormente
il
potenziale
elettrochimico
generato
dal
trasporto elettronico attraverso i
Complessi I e III
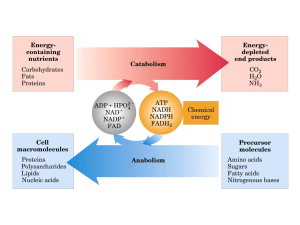
L’energia associata al trasporto elettronico viene efficientemente conservata sotto forma di
gradiente di protoni, che si genera a livello dei Complessi I, III e IV
(per quest’ultimo viene rappresentata una semi-seazione)
L’energia elettrochimica contenuta nel gradiente di concentrazione protonica e nella
separazione delle cariche elettriche genera una forza protomotrice (o forza motrice
protonica, ΔG), che è formata da due componenti: energia potenziale chimica
(gradiente protonico, ΔpH) ed energia potenziale elettrica (gradiente elettrico, Δψ)
[R, costante universale dei gas
T, temepratura assoluta
Z, carica elettrica dello ione
F, costante di Faraday
Δψ, potenziale di membrana]
Modello chemiosmotico di Mitchell
[Reazione generale: ADP + Pi + nHP+ → ATP + H2O + nHN+]
L’accoppiamento del trasferimento
elettronico con la sintesi di ATP nei
mitocondri richiede la presenza di
ADP + Pi e di un substrato
ossidabile (come il succinato)
L’accoppiamento è bloccato da
inibitori della citocromo ossidasi
(come il cianuro, CN-) o dell’ATP
sintasi (come la venturicidina e
l’oligomicina)
Gli “agenti disaccoppianti” (come il
dinitrofenolo, DNP) bloccano la
sintesi di ATP, pur consentendo la
respirazione
I disaccoppianti chimici della
fosforilazione
ossidativa
possiedono
un
protone
dissociabile (sono acidi deboli) e
sono molto idrofobici
Essi
agiscono
protoni attraverso
mitocondriale
dissipando così
protonico (ΔpH)
trasportando
la membrana
interna
e
il gradiente
ATP sintasi
La reazione ADP + Pi ↔ ATP + H2O è facilmente reversibile sulla superficie di
F1, dove l’ATP viene stabilizzato più dell’ADP ma viene rilasciato grazie al
gradiente protonico
Il meccanismo catalitico di F1 è stato in gran parte chiarito, anche grazie ad
esperimenti di scambio di 18O in presenza di acqua marcata (a) Lo stato di
transizione più probabile per l’idrolisi e la sintesi di ATP è stato identificato (b)
Diagrammi delle coordinate di reazione dell’ATP sintasi e di un enzima modello
Anche se la variazione di energia libera per la formazione di ATP da ADP e Pi in una soluzione
acquosa è grande e di segno positivo, il legame molto saldo dell’ATP all’enzima
F1 fornisce una quantità di energia di legame che abbassa l’energia libera dell’ATP
legato a valori simili a quella dell’ADP + Pi
L’energia libera richiesta per il rilascio dell’ATP è fornita dalla forza protomotrice
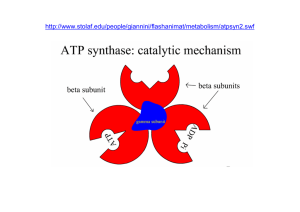
Meccanismo di catalisi rotazionale
Il modello della “modificazione del
legame” dell’ATP sintasi si basa sul
fatto che il complesso F1 possiede tre
siti non equivalenti per il legame dei
nucleotidi adenilici, uno per ciascuna
coppia di subunità α e β. In ogni
momento, uno di questi siti si trova
nella conformazione β-ATP (che lega
saldamente l’ATP), il secondo in
conformazione β-ADP (che lega
debolmente l’ATP) ed il terzo in
conformazione β-vuota (che lega molto
debolmente l’ATP)
La forza protomotrice provoca la
rotazione dello stelo centrale (la
subunità γ, in verde) che, toccando un
sito alla volta, ne modifica la
conformazione, permettendo la sintesi
ed il rilascio di ATP
La teoria chemiosmotica supporta stechiometrie frazionarie per la sintesi di ATP
ed il consumo di ossigeno (rapporto P/O)
xADP + xPi + 1/2O2 + NADH + H+ → xATP + H2O + NAD+
dove il valore di x rappresenta il “rapporto P/O” (o rapporto P/2e-)
Moltissimi studi sperimentali hanno prodotto rapporti P/O compresi tra 2 e 3, quando il
donatore di e- è il NADH, e tra 1 e 2, quando il donatore è il succinato. Presumendo che
P/O dovesse essere un numero intero, per anni si è convenuto che esso valesse 3 a partire
dal NADH e 2 a partire dal succinato
Tuttavia, l’accettazione universale della teoria chemiosmotica ha permesso d’ipotizzare
anche valori frazionari per il rapporto P/O. Stabilito concordemente che:
i protoni pompati fuori dal mitocondrio per ogni coppia di elettroni sono 10 per il
NADH e 6 per il succinato;
servono 4H+ per la sintesi di un ATP (di cui uno è necessario per trasportare Pi, ADP ed
ATP attraverso la membrana mitocondriale)
si è arrivati a concludere che il rapporto P/O è pari a 2.5 (10/4) per il NADH e 1.5 (6/4)
per il succinato
Queste stechiometrie sono largamente accettate, ma diventeranno definitive solo quando i
dettagli del meccanismo di reazione di F0F1 saranno completamente definiti
ATP sintasoma
Shuttle del malato-aspartato
Shuttle del glicerolo 3-fosfato
Regolazione della fosforilazione ossidativa
La completa ossidazione di una molecola di glucosio a CO2 rende 30 o 32 molecole di ATP (a
seconda dello shuttle utilizzato), mentre la glicolisi si ferma a 2 molecole soltanto
La fosforilazione ossidativa è regolata dal fabbisogno energetico cellulare,
secondo vari meccanismi:
a) Controllo da accettore della respirazione, vale a dire dipendenza della velocità di
consumo di O2 dalla concentrazione di ADP, l’accettore del gruppo fosforico.
Si definisce rapporto di controllo da accettore quello tra la velocità massima
di consumo di O2 in presenza di concentrazioni ottimali di ADP e la velocità
basale in assenza di ADP (con valori anche superiori a 10)
b) Rapporto di azione di massa del sistema ATP-ADP, vale a dire [ATP]/([ADP][Pi])
La sintesi di ATP continua fino a che il rapporto di azione di massa non
raggiunge il suo valore massimo, un evento che fa rallentare
la respirazione cellulare
c) Proteine regolatrici contribuiscono a regolare i livelli di ATP. Per esempio,
un inibitore proteico (IF1) impedisce l’idrolisi dell’ATP durante l’ipossia
(per esempio, per un attacco cardiaco od un colpo apoplettico)
IF1 (84 amminoacidi) si lega contemporaneamente a due molecole
di ATP sintasi, inibendone l’attività ATPasica
L’inibitore è attivo solo nella sua forma dimerica, che può esistere
a pH < 6.5 (metabolismo anaerobico!)
Un problema rilevante della fosforilazione ossidativa è la formazione di ROS, a causa di
“perdite” di elettroni lungo la catena respiratoria. Servono, quindi, meccanismi di
detossificazione efficienti (in blu), per difendere il mitocondrio e la cellula
Il fattore inducibile dell’ipossia (HIF-1) regola l’espressione genica
per ridurre la formazione di ROS
I meccanismi di regolazione delle vie
che producono ATP sono coordinati
Le interconnessioni delle regolazioni della
glicolisi, dell’ossidazione del piruvato, del ciclo
dell’acido citrico e della fosforilazione ossidativa
sono dipendenti dalle concentrazioni relative di
ATP, ADP, AMP, NADH e NAD+
L’azione del citrato nell’inibire la glicolisi
ed il ciclo dell’acido citrico è coordinata
con quella dei nucleotidi adeninici
Il tessuto adiposo bruno
Le P-450 ossigenasi (o citocromi P-450) mitocondriali catalizzano l’ossidrilazione
degli steroidi nella ghiandola surrenale, specializzata appunto nella sintesi degli
ormoni steroidei
Le P-450 ossigenasi del reticolo endoplasmatico degli epatociti catalizzano
reazioni simili a quelle degli enzimi P-450 mitocondriali, ma i loro substrati
sono composti idrofobici, tra cui molti xenobiotici (sostanze non presenti in
natura, ma sintetizzati industrialmente)
I mitocondri hanno un ruolo primario anche nella
fase iniziale dell’apoptosi
All’inizio di questo processo, la membrana
mitocondriale esterna aumenta notevolmente la sua
permeabilità, a causa dell’apertura del complesso del
poro di transizione della permeabilità (PTPC), una
proteina multimerica
Il citocromo c può così uscire nel citosol, dove
interagisce con i monomeri della proteina Apaf-1
(fattore-1 di attivazione della proteasi dell’apoptosi),
causando la formazione di un apoptosoma
(composto da 7 molecole di Apaf-1 e 7 molecole di
citocromo c)
Questo complesso costituisce una piattaforma sulla
quale la proteasi procaspasi-9 viene attivata a caspasi9, che porta avanti una cascata proteolitica nel
processo apoptotico
[Tutte le caspasi sono proteasi a cisteina, che tagliano
i legami peptidici dal lato carbossilico di un acido
aspartico (da cui il nome)]