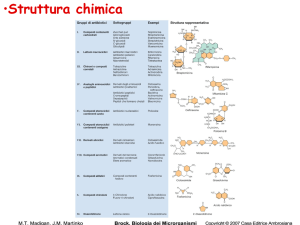
Farmaci antiparassitari
I
farmaci
antiprotozoari
selettivamente sul parassita:
agiscono
– inibendo la sintesi degli acidi nucleici (chinina,
clorochina,
meflochina,
metronidazolo,
pentamidina, etc.)
– inibendo la sintesi dei cofattori (trimethoprim,
sulfadossina, dapsone, etc.)
– inibendo la sintesi delle proteine (tetraciclina)
– destabilizzando
la
membrana
cellulare
(amfotericina B)
– interferendo con il metabolismo energetico
(primachina)
Farmaci antiparassitari
I farmaci antiprotozoari presentano
tossicità relativa per l’ospite in quanto:
– si concentrano nel parassita (chinina,
clorochina,
meflochina,
pentamidina,
tetraciclina)
– sono attivati dal parassita (metronidazolo)
– il bersaglio d’azione è presente solo nel
parassita (sulfadossina, dapsone)
Farmaci antiparassitari
I
farmaci
antielmintici
selettivamente sul parassita:
agiscono
– interferendo con la funzione dei microtubuli
(benzimidazoli)
– inibendo le funzioni neuromuscolari (ivermectina)
– interferendo con il metabolismo energetico
(diclorofene)
I farmaci antielmintici presentano tossicità
relativa per l’ospite in quanto:
– sono in grado di discriminare tra bersaglio
nell’ospite
e
bersaglio
nel
parassita
(benzimidazoli)
Farmaci antiprotozoari
Classe del
Farmaco
Meccanismo
d’azione
Esempi
Indicazione
clinica
Metalli pesanti:
arsenicali e
antimonici
Inattivano gruppi
sulfidrilici
Melarsoprol
Stibogluconato di Na
Tripanosomiasi
Leishmaniosi
Analoghi della
amminochinolina
Si accumulano nelle cellule
parassitate; bloccano la
replicazione del DNA; legano
la ferroprotoporfirina IX;
causano aumento del pH
intravescicolare; bloccano la
digestione dell’emoglobina
Clorochina
Meflochina
Chinina
Primachina
Antagonisti
dell’acido folico
Bloccano la sintesi di
acido folico
Sulfamidici
Trimethoprim
Pirimetamina
Toxoplasmosi
Malaria
Ciclosporidiasi
Inibitori sintesi
proteica
Bloccano la sintesi
proteica a livello dei
ribosomi
Clindamicina,
Spiramicina,
Paromomicina,
Tetraciclina, Doxiciclina
Malaria,
Babesiasi,
Amebiasi,
Criptosporidiosi
Diammidine
Legano il DNA
Bloccano la funzione delle
poliammine
Pentamidina
Pneumocistosi
Tripanosomiasi
Leishmaniosi
Antimoniato di meglumina
Profilassi e
terapia della
malaria
Cura radicale
(solo primachina)
Farmaci antiprotozoari
Classe del
Farmaco
Meccanismo
d’azione
Esempi
Indicazione
clinica
Nitroimidazoli
Non chiarito; danni al DNA
Inibizione del metabolismo
del glucosio; interferenza
con funzione mitocondriale
Metronidazolo
Benzimidazolo
Tinidazolo
Amebiasi
Giardiasi
Tricomoniasi
Chinoloni
Inibizione di DNA girasi
Ciprofloxacina
Malaria
Sesquiterpeni
Reagiscono con eme,
formando radicali liberi,
con danno alle membrane
Artemisinina
Malaria
Analogo
dell’ornitina
Blocco dell’ornitindecarbossilasi e del
metabolismo di poliammine
Difluorometilornitina
Tripanosomiasi
africana
Inibitori della sintesi
di acidi nucleici
Blocco di enzimi per il
recupero delle purine
Allopurinolo
Leishmaniosi
Acetanilide
Sconosciuto
Dilaxanide fuorato
Naftilammina
solfato
Ridotta sintesi di ATP; Blocco
di glicerolfosfato ossidasi e
glicerol 3-fosfato deidrogenasi
Suramina
Amebiasi
intestinale
Tripanosomiasi
africana
Fenantrenemetanoli
Blocco dei mitocondri
Alofantrina
Malaria
Farmaci antivirali
Esempi di bersagli di farmaci antivirali
Terapie con farmaci antivirali approvati
dalla FDA americana
Analoghi
nucleosidici
sono analoghi strutturali dei
nucleotidi, che fungono da
terminatori della catena
Vengono riconosciuti da
DNA-polimerasi, RNApolimerasi, reversotrascrittasi.
Aggiunti alla catena
nucleotidica, durante la
sintesi degli acidi nucleici,
non permettono il legame del
nucleotide successivo,
bloccando la sintesi
Inibitori non nucleosidici della
reverso-trascrittasi o della DNApolimerasi
acido fosfonoformico
(sale trisodico: Foscarnet)
si legano all’enzima
bloccandolo nel sito
di scambio del
pirofosfato
acido fosfonoacetico
si legano
all’enzima,
bloccandolo, in
diversi siti
Nevirapina (diazepina)
Delavirdina (piperazina)
Inibitori delle proteasi
nelle fasi conclusive del ciclo moltiplicativo, la proteasi taglia alcune
proteine che assumono la struttura definitiva (HIV: maturazione
della gp160); il blocco della proteasi porta alla produzione di
virioni immaturi non infettivi
Saquinavir
Indinavir
Ritonavir
Nelfinavir
Farmaci antivirali anti-influenzali
amine attive
solo nei
confronti del
virus della
influenza A
Amantadina
Rimantadina
Analoghi dell’acido sialico:
bloccano la neuraminidasi dei virus influenzali
Resistenza agli antibiotici
Resistenza intrinseca (naturale)
insensibilità naturale di alcune specie batteriche nei
confronti di alcuni antibiotici e chemioterapici
- mancata penetrazione nella cellula
- sistemi di efflusso specifici ed aspecifici
- enzimi inattivanti l’antibiotico (codificati da geni
cromosomici)
- assenza del bersaglio
Resistenza agli antibiotici
Resistenza acquisita
endogena o cromosomica
acquisita per mutazione (1×106 - 1×109)
* alterazione del bersaglio
* alterazione della permeabilità (es. alterazione porine)
di solito nei confronti di un solo antibiotico o di antibiotici
della stessa classe (resistenza crociata)
i mutanti di solito sono meno vitali, l'antibiotico li seleziona,
la resistenza è reversibile in assenza di pressione selettiva
esogena
acquisita da plasmidi, trasposoni o per ricombinazione
genetica
Fattori R
di solito resistenza multipla per più antibiotici contemporaneamente
epidemiologicamente molto importante per la diffusione di ceppi
multiresistenti
Esempi di fattori R identificati in alcuni microrganismi
Microrganismo
Enterobacteriaceae
Pseudomonas
Streptococcus faecalis
Staphylococcus aureus
Plasmide
R1
R64
R386
R388
RP4
TP116
R1033
RP1
pMG30
pJH1
pIP800
pSH2
pSJ19
Resistenza conferita
Ap,Cm,Km,Sm,Su
Tc,Sm
Tc
Su,Tp
Ap,Tc,Km
Cm,Sm,Su
Cb,Cm,Gm,Km,Sm,Su,Tc,Hg
Cb,Km,Tc
Gm,Sm,Su,Tc,Hg,Pmr
Em,Tc,Km
Km,Gm,Cm
Nm
Km,Tm,Gm,An
An: amikacina, Ap: ampicillina, Cb: carbenicillina, Cm: cloramfenicolo, Em: eritromicina, Gm: gentamicina, Hg: cloruro di mercurio,
Km: kanamicina, Nm: neomicina, Pmr: acetato fenilmercurico, Sm: streptomicina, Su: sulfonamidi, Tc: tetraciclina, Tp: trimetoprim
Trasposoni codificanti per la resistenza ad
antibiotici
Meccanismi molecolari della resistenza
Ridotta permeabilità cellulare
- alterazione delle porine
- Alterazione del lipopolisaccaride (Gram negativi)
(β-lattamici, cicloserina, fosfomicina, chinoloni, aminoglicosidi, cloramfenicolo,
tetracicline)
Aumentato efflusso
sistemi di efflusso simili a quelli operanti
nelle resistenze intrinseche
(l'antibiotico viene espulso dalla cellula)
(β-lattamici, fluorochinoloni, rifampicina, macrolidi, cloramfenicolo,
tetracicline)
Sistemi di efflusso
Esempi di sistemi di efflusso
Rappresentazione schematica di sistemi
di efflusso presenti in batteri Gram negativi
Il sistema di efflusso è localizzato sulla faccia interna della membrana citoplasmatica ed
espelle l’antibiotico nello spazio periplasmico, dove possono essere presenti enzimi
inattivanti (es. β-lattamasi). In alcuni casi la pompa di efflusso è accoppiata con
proteine accessorie e proteine canale presenti nella membrana esterna che provvedono
all’espulsione dell’antibiotico all’esterno della cellula
Alterazione del bersaglio
Ridotta affinità del bersaglio per l'antibiotico
proteina bersaglio mutata, ma ancora biologicamente attiva
(mutazione cromosomica o ricombinazione genetica)
- alterata RNA polimerasi → resistenza a rifampicina
- PBP mutate → resistenza a β–lattamici
- DNA girasi mutata → resistenza a chinoloni
(β-lattamici, chinoloni, rifampicina, aminoglicosidi, macrolidi,
cloramfenicolo, tetracicline, sulfamidici, trimetoprim)
Iperproduzione del bersaglio
mutazione a livello di controllo genico dell'operone
- iperproduzione PBP → resistenza a β–lattamici
- iperproduzione diidropteroato sintetasi e deidrofolato reduttasi →
→ resistenza ai sulfamidici e trimetoprim
(β -lattamici, cicloserina, sulfamidici, trimetoprim)
Alterazione del bersaglio
Sostituzione del bersaglio
acquisizione di un nuovo gene che codifica per un bersaglio
con ridotta affinità per l'antibiotico
- gene mecA in S.aureus (PBP2') → resistenza a tutti i
β–lattamici (ceppi meticillino-resistenti)
- resistenza a Glicopeptidi (Vancomicina e Teicoplanina):
Tn 1546: 9 geni → sostituzione di D-alanil-D-alanina
del peptidoglicano con D-alanil-D-lattato, che non lega tali
antibiotici
(β -lattamici, glicopeptidi, sulfamidici, trimetoprim)
Modificazione enzimatica del bersaglio
unico esempio: resistenza ai macrolidi
gene plasmidico codificante per un enzima che metila RNA
ribosomale 23S → l'antibiotico non si lega più al ribosoma
Produzione di enzimi inattivanti
geni cromosomici, plasmidici, trasposoni
- β-lattamasi (penicilline, cefalosporine): molto diffuse
- glutatione S-transferasi (cicloserina)
- acetiltransferasi, adeniltransferasi, fosfotransferasi
(aminoglicosidi)
- acetiltransferasi (cloramfenicolo)
(β-lattamici, fosfomicina, cicloserina, aminoglicosidi,
cloramfenicolo)
Ridotta attività di funzioni enzimatiche secondarie
alcuni antibiotici vengono attivati in vivo dal metabolismo batterico
alterazioni degli enzimi coinvolti determinano resistenza
- nitrofurani: attivati da nitrato reduttasi batterica
- nitroimidazoli: attivati da piruvato deidrogenasi
Resistenza agli antibiotici
Conseguenze
Necessità di adottare strategie per la prevenzione delle
resistenze
• uso razionale degli antibiotici in terapia
• utilizzazione di associazioni di antibiotici in terapia
• ricerca di nuovi antibiotici:
- in grado di inibire i meccanismi molecolari della resistenza
- insensibili ai meccanismi di resistenza
- in grado di colpire nuovi bersagli molecolari
Necessità di saggiare la sensibilità agli antibiotici e
chemioterapici (Antibiogramma) dei ceppi batterici agenti
eziologici di malattie da infezione per l’adozione di una
terapia “mirata”
Potenziali meccanismi molecolari di
resistenza ai farmaci antifungini
Alterazioni dell’entrata del farmaco
Alterazioni della processazione intracellulare del farmaco
Modificazione
Degradazione
Alterazioni dell’enzima bersaglio
Mutazioni puntiformi
Iperproduzione
Amplificazione genica
Alterazioni di enzimi della via biosintetica dell’ergosterolo
Alterazioni delle pompe di efflusso
ABC transporters
Major facilitators
Resistenze ai farmaci antivirali
I virus diventano facilmente resistenti per mutazione
Necessità di utilizzare combinazioni di farmaci
(almeno 1 per classe)
Mutazioni primarie
Mutazioni secondarie
Sono le prime a comparire
sotto la pressione selettiva del
farmaco
Si manifestano in genomi in
cui sono già presenti mutazioni
primarie
Sono farmaco-specifiche
Sono farmaco-aspecifiche
Determinano la
farmaco-resistenza
Migliorano la capacità
replicativa del virus
Influiscono sul legame
farmaco-enzima
Influiscono sul sito catalitico
dell'enzima
Antibiogramma
Saggio di sensibilità agli antibiotici in vitro
– Minima concentrazione inibente (MCI)
La più bassa concentrazione in cui c’è inibizione
visibile della crescita
– Minima concentrazione battericida (MCB)
La più bassa concentrazione che uccide il 99.9%
delle cellule dell’inoculo originale
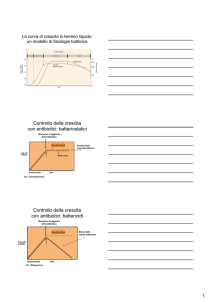
Antibiogramma
mediante metodo di diluizione in brodo
Determinazione della concentrazione minima inibente (CMI o
MIC) e della concentrazione minima battericida (CMB o MBC)
mediante metodo di diluizione in brodo
A. Una serie di provette, contenenti un terreno di coltura liquido,
trasparente ed idoneo alla crescita del batterio, e quantità
decrescenti di antibiotico, viene inoculata con una quantità fissa
molto piccola di batteri.
B. Dopo opportuna incubazione (18-24 ore, a 37°C), viene valutata
visivamente la comparsa di torbidità. La prima provetta l
impida equivale alla CMI.
C. Per determinare la CMB, aliquote di brodo vengono prelevate
dalle provette B limpide e seminate su piastre dello stesso terreno
agarizzato, ma privo di antibiotico. La mancata comparsa di
colonie, dopo opportuna incubazione, indica la CMB.
Metodo di diluizione in brodo per la
determinazione di MCI e MCB
Determinazione della sensibilità mediante metodo
di diffusione in agar (metodo Kirby-Bauer)
Dischetti imbevuti di una quantità predefinita di antibiotico vengono applicati
manualmente (A) o per mezzo di un distributore multiplo (C) sulla superficie di
una piastra contenente il terreno di saggio, preventivamente inoculata con il
batterio in esame (B). Durante l’opportuna incubazione (es. 18-24 ore, a 37°C),
l’antibiotico diffonde dal dischetto ed eventualmente inibisce la crescita del
batterio nell’area circostante (alone di inibizione della crescita).
Si misura il diametro degli aloni in mm. (D)
Assenza di alone: resistenza certa
Presenza di alone: interpretazione
(Sensibile, Intermedio, Resistente)
A
B
C
D
Correlazione tra CMI e diametro
dell’alone di inibizione
Per ogni antibiotico viene
costruita una retta di
regressione: molti ceppi
con CMI nota vengono
saggiati con il metodo
Kirby-Bauer e viene
costruito
un
grafico
simile a quello mostrato
(CMI in ordinata; alone
di inibizione in mm in
ascissa).
Per ogni nuovo ceppo basterà riportare nel grafico il diametro in
mm per ottenere la corrispondente CMI.
Interpretazione dei saggi di sensibilità
Concentrazione limite (breakpoint): valore di CMI che permette
di discriminare i ceppi sensibili dai ceppi resistenti. Per ogni
antibiotico sono fissati due breakpoints: uno più basso che
definisce la sensibilità (il ceppo è inibito a concentrazioni ≤ al
breakpoint) e uno più alto che definisce la resistenza (il ceppo
cresce a concentrazioni ≥ al breakpoint). Se il ceppo batterico è
inibito dalla seconda concentrazione ma non dalla prima viene
definito intermedio.
Picco ematico: la più elevata concentrazione di un farmaco
raggiungibile nel sangue dopo somministrazione a dosi
terapeutiche (secondo la posologia e la via di somministrazione).
I ceppi batterici vengono definiti:
Sensibile (S):
CMI = a ¼ del valore del picco ematico
Resistente (R):
CMI = o > del valore del picco ematico
Intermedio (I):
CMI compresa tra le precedenti
Diffusione da disco
E-test®
Consiste in una combinazione dei saggi di diffusione e di
diluizione e consente la determinazione del valore preciso di CMI