Corsi di laurea magistrale in:
Biotecnologie agrarie e ambientali (LM-7)
Biologia cellulare e molecolare (LM-6)
Sicurezza e qualità agroalimentare (LM-69 & LM-70)
insegnamento di
Basi Teoriche e Applicazioni delle
Nuove Tecnologie Genomiche
Aula 16
Orario, venerdi 8:50-11:20
Silvia Bongiorni:
tel.:
0761-357429
email:
[email protected]
recapito: DIBAF stanza 23A (seminterrato ex facoltà agraria)
GENOMICA STRUTTURALE:
1.
2.
3.
4.
5.
6.
7.
GENOMICA FUNZIONALE:
Anatomia dei genomi
8. Funzionamento dei genomi
Il genoma dei procarioti
Modificazioni della cromatina e l’espressione del genoma
Il genoma degli eucarioti
Microarray e RNA-seq
La mappatura dei genomi
Metil-seq
Mappatura genetica
Chip-seq
Mappatura fisica
Il sequenziamento automatico del DNA
Il principio del sequenziamento secondo Sanger
Il sequenziamento su larga scala
La lettura dei tracciati di sequenziamento
Il sequenziamento del genoma
I nuovi metodi di sequenziamento
Il sequenziamento gerarchico
Il sequenziamento shogun
Piattaforme di sequenziamento di interi genomi
La verifica delle sequenze e assemblaggio dei contig
L’annotazione del genoma
Il sequenziamento delle EST
Le caratteristiche funzionali delle sequenze genomiche
I Progetti Genoma
Il Progetto Genoma Umano
Il Progetto Genoma Animali
Il Progetto Genoma Piante
Il Progetto Genoma Microrganismi
Genotipizzazione
Gli SNP e la variazione
La genotipizzazione degli SNP
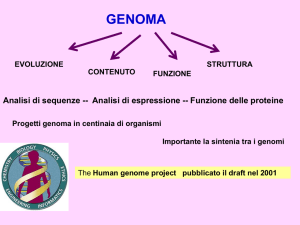
Il GENOMA è l’insieme di tutti i geni di un organismo. L’insieme di tutte
le sequenze nucleotidiche codificanti e non di un organismo.
La sequenza completa del suo DNA
La scienza dei genomi, la GENOMICA è lo studio della struttura, del
contenuto e dell’evoluzione dei genomi
La genomica non è più limitata alla determinazione delle sequenze ma
si estende anche all’analisi dell’espressione e delle funzioni dei geni e
delle proteine
Le analisi sperimentali sono integrate strettamente da
Bioinformatica
Biologia computazionale
GENOMICA STRUTTURALE:
1. Anatomia dei genomi
Il genoma dei procarioti
Il genoma degli eucarioti
Organismo
Numero bp
Numero di geni
ΦX-174
5836
10
Mycoplasma
pneumoniae
816394
680
Helicobacter pylori
1667867 (1,66 Mb)
1589
Escherichia coli
4639221 (4,64 Mb)
4377
Saccharomyces
cerevisiae
12495682 (12,5 Mb)
5770
Caenorhabditis
elegans
100258171 (100 Mb)
19099
Drosophila
melanogaster
122653977 (122,6 Mb)
13472
Homo sapiens
3,3 x 109 (3289 Mb o 3,3 Gb)
25000
Il genoma dei procarioti
DNA batterico
DNA plasmidico
Il genoma degli eucarioti
Compattazione della cromatina
1,70m in 10µ
µm
146bp
54bp
2-6 µm
genoma
cromosoma
gene
gene
regione intergenica
Perché alcuni genomi sono così grandi?
La quantità di DNA contenuta nel
genoma aploide è una
caratteristica propria di ciascuna
specie vivente ed è definita come
valore C
Il valore C varia enormemente da
<106 bp per un micoplasma a >1011
bp per anfibi ed alcune piante
La dimensione del genoma cresce
con la complessità dell’organismo.
Ma in alcuni gruppi c’è un’enorme
variabilità nel contenuto di DNA di
specie simili fra loro
Il paradosso del valore C si riferisce alla mancanza di correlazione tra le dimensioni del
genoma e la complessità biologica
I genomi eucariotici contengono
sia sequenze uniche che DNA
ripetuto
Il DNA non ripetuto è formato da
sequenze uniche: una copia nel
genoma aploide
Il DNA ripetuto è formato da
sequenze che sono presenti in più
copie nel genoma aploide
• DNA moderatamente ripetuto
(sequenze >100bp ripetute tra le
10 e le 1000 volte)
• DNA altamente ripetuto
(sequenze <100bp ripetute
migliaia di volte)
Quanti geni ci sono nei vari genomi?
Organismo
Numero bp
Numero di geni (% codificante)
ΦX-174
5836
10
Mycoplasma
pneumoniae
816394
680
Helicobacter pylori
1667867
1589
Escherichia coli
4639221 (4,64 Mb)
4377 (88)
Saccharomyces
cerevisiae
12495682 (12,5 Mb)
5770 (70)
Caenorhabditis
elegans
100258171 (100 Mb)
19099
Drosophila
melanogaster
122653977 (122,6 MMb)
13472
Homo sapiens
3,3 x 109 (3289 Mb)
25000 (1,3)
non-coding DNA
Junk-DNA
Junk-DNA:
ruolo nel sileziamento genico
Silenziamento post-trascrizionale di geni attivi:
ad opera di micro-RNA (miRNA) e small
interfering RNA (siRNA) (21-23bp)
complementari
miRNA e siRNA legano l’mRNA complementare,
richiamano il complesso RISC e degradano
l’mRNA
Junk-DNA: ruolo nell’evoluzione
geni umani vs gene di scimpanzè:
•differiscono dello 0,1% del genoma (circa 20 geni)
•differenze nelle regioni non-coding sono 40 volte superiori (4%)
– il ncDNA può fare la differenza tra uomo e scimpanzè
Qual è la funzione dei vari geni?
Struttura dei geni
esone
exon
expressed region
introne
intron
intervening region
Splicing
Splicing alternativo
A
C
D
A B C D E
F
B
mRNA 1
AAAAAAA
A B C E
F
mRNA 2
AAAAAAA
A B C D
F
AAAAAAA
mRNA 3
A B C
mRNA 4
E
F
AAAAAAA
F
Analisi di sequenze genomiche
Gli introni sono abbondanti
94% uomo, 85% moscerini, 95% nematode, 95% piante
Genoma umano (25000 geni)
solo 2 - 5 volte più geni del moscerino (13472) o del nematode (19099)
Lo splicing alternativo è frequente
~ il 75% dei geni umani trascritti subiscono splicing alternativo
Geni sovrapposti
Alcune sequenze di DNA codificano per più proteine
GENOMICA STRUTTURALE:
2.
La mappatura dei genomi
Mappatura genetica
Mappatura fisica
Le mappe indicano la localizzazione
delle cose in relazione ad altre cose
Le mappe genomiche rivelano l’organizzazione del materiale ereditario
e possono essere suddivise in 2 tipi:
1. Mappe genetiche: posizionano i geni sui cromosomi sulla base della
frequenza di ricombinazione
2. Mappe fisiche: usano marcatori all’interno della sequenza, che
vanno dai siti di restrizione all’analisi diretta del DNA genomico
Mappe Genetiche
descrivono l’ordine relativo dei marcatori genetici nei gruppi di associazione o LINKAGE
Marcatori = brevi sequenze ripetute di DNA (microsatelliti, Short Tandem Repeats),
polimorfismi (Single Nucleotide Polymorphism), siti di restrizione, loci genici
Le mappe genetiche sono costruite partendo dai dati di segregazione dei marcatori e dalla
loro frequenza di ricombinazione nella progenie
perciò la distanza tra marcatori è espressa in centi-Morgan (cM), unità di misura della
frequenza di ricombinazione
1cM = frequenza di ricombinazione dell’1% (circa 1000bp nell’uomo, 2000bp nel topo)
Per fare una mappa genetica o di associazione (linkage) non necessariamente serve
conoscere la sequenza
Limiti:
1. la distanza genetica non corrisponde direttamente alla distanza fisica
(eu/eterocromatina, sequenze ripetute)
2. non tutti i geni hanno un fenotipo ovvio
Mappe Genetiche
MAPMAKER
A program for genetic linkage
analysis
MAPMAKER/EXP was developed
to be a linkage analysis package
designed to help construct
primary linkage maps of markers
segregating
in
experimental
crosses
MAPMAKER/QTL is a companion
program to MAPMAKER/EXP
which allows one to map
genes
controlling
polygenic
quantitative
traits
in
F2
intercrosses and BC1 backcrosses
relative to a genetic linkage map.
More
information
on
MAPMAKER/QTL can be found in
the technical report.
Mappe Fisiche
è una costellazione di frammenti di
DNA sovrapposti, ordinati e
orientati, che coprono ciascun
cromosoma di un genoma.
Le mappe fisiche sono basate
sull’analisi diretta del DNA genomico
ed indicano l’effettivo numero di
coppie di basi (bp), kilobasi (kb) o
megabasi (Mb) che separano un
marcatore
(locus)
da
quello
adiacente.
Ci sono diverse strategie per allineare i contig:
• Ibridazione (chromosome walking)
• Sequenziamento delle estremità (STS, sequence-tagged sites)
• Profili di restrizione
Mappe Fisiche
Nello sviluppare una mappa
fisica dell’intero genoma è
importante considerare la
lunghezza degli inserti dei
cloni e l’ordine dei cloni lungo
il genoma
La costruzione delle mappe fisiche di interi genomi, cioè l’assemblaggio di
tratti contigui (contigs) di DNA cromosomico sarà discusso più avanti
La mappatura dei genomi
Le mappe genetiche e fisiche possono essere correlate
Qualsiasi gene clonato
può essere mappato
geneticamente e
posizionato fisicamente
nel genoma
IL METODO PRINCIPALE PER ALLINEARE TRA LORO MAPPE GENETICHE E
MAPPE FISICHE E’ RAPPRESENTATO DALLE MAPPE CITOLOGICHE
Mappe Cromosomiche
bandeggio
Ibridazione in situ
Le Mappe
Tutti i dati di sequenza, ricombinazione derivanti da queste mappe
sono immagazzinati in banche dati (database)
The National Center for Biotechnology Information
http://www.ncbi.nlm.nih.gov/
The European Molecular Biology Laboratory
http://www.embl.de/
DNA Data Bank of Japan
http://www.ddbj.nig.ac.jp/index-e.html