Quantificazione di acidi nucleici mediante coloranti fluorescenti
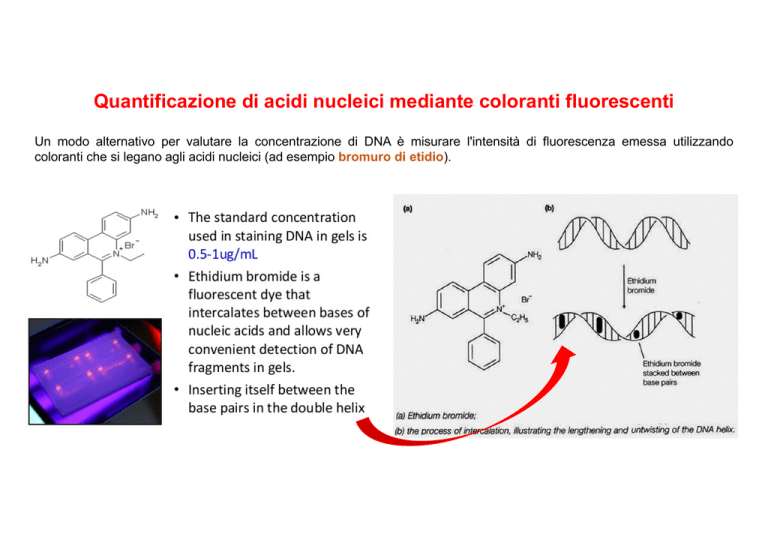
Un modo alternativo per valutare la concentrazione di DNA è misurare l'intensità di fluorescenza emessa utilizzando
coloranti che si legano agli acidi nucleici (ad esempio bromuro di etidio).
Quantificazione di acidi nucleici usando l’elettroforesi in gel di Agarosio
L’elettroforesi su gel di Agarosio è un metodo di elettroforesi molto utilizzato in biochimica, biologia molecolare e chimica
clinica per separare DNA, RNA (o proteine) in una matrice di agarosio. Il DNA o frammenti di DNA e l’RNA sono separati
applicando un campo elettrico per far migrare in base alle loro dimensioni dell molecole cariche attraverso una matrice di
agarosio.
β-(1-4)-(3,6)-anhydro-L-galactose
α-(1-3)-D-galactose
Quantificazione di acidi nucleici usando l’elettroforesi in gel di Agarosio
Proprietà del gel di Agarosio. Il gel di Agarosio è una matrice tridimensionale formata da molecole di agarosio
elicoidali strutturate in fasci superavvolti, raggruppati in strutture tridimensionali formanti canali e pori attraverso i
quali possono passare delle biomolecole. La struttura 3-D è tenuta insieme da legami idrogeno e può quindi essere
interrotta mediante riscaldamento del gel, ri-portandolo allo stato liquido. La temperatura di fusione è diversa dalla
temperatura di gelificazione, a seconda delle fonti, il gel di agarosio ha una temperatura di gelificazione di 35-42 °C e una
temperatura di fusione di 85-95 °C. Sono inoltre commercialmente disponibili agar con basso punto di fusione e bassa
proprietà gelificante, ottenute tramite modificazioni chimiche.
Quantificazione di acidi nucleici usando l’elettroforesi in gel di Agarosio
I gel di agarosio sono facili da allestire e sono particolarmente adatti per la separazione di DNA di varie dimensioni e spesso utilizzati nei
laboratori.
Il DNA separato può essere visualizzato come una banda, più frequentemente illuminandolo con una luce UV, ed i frammenti di DNA possono
essere estratti facilmente dal gel. La maggior parte dei gel di agarosio sono usati a concentrazioni tra 0.7-2% e sciolti in un opportuno
tampone di elettroforesi.
Il polimero di agarosio contiene gruppi carichi, in particolare piruvato e solfato. Durante la corsa elettroforetica si può assistere
all'insorgenza di un fenomeno è la conseguenza di una differenza di carica tra le molecole di acqua del tampone e la superficie del gel. Ciò
genera una forza motrice che provoca il movimento verso il catodo degli ioni del tampone che, per un effetto di trascinamento del solvente,
portano con se anche molecole prive di carica. Questo fenomeno accelera il movimento dei cationi e ritarda quello degli anioni essendo
contrario alla loro migrazione. L'effetto elettro-endosmotico (EEO), può essere vantaggioso o dannoso. E‘ vantaggioso nell'elettroforesi
capillare, mentre è dannoso nell'isoelettrofocusing. L’elettroendoosmosi può quindi ritardare il movimento di DNA e causare sfocatura di
bande. Una maggiore concentrazione di gel avrebbe un più alto flusso elettro-endosmotico. Un basso EEO è quindi generalmente preferito per
elettroforesi di acidi nucleici, ma agarosio ad alta EEO può essere utilizzato per scopi specifici. Un agaroso con minore contenuto in solfati, con
un punto di fusione particolarmente basso (LMP), è anche utile nei casi in cui il DNA estratto dal gel deve essere utilizzato per ulteriori
manipolazioni dove la presenza di solfati contaminanti possono influenzare alcune procedure, come reazioni di ligasi o di PCR.
L’Agarasi: un enzima usato per digerire
frammenti di DNA “ritagliati” (excisi) da gel d’agarosio
Quantificazione di acidi nucleici usando l’elettroforesi in gel di Agarosio
Il gel di agarosio per le dimensioni dei pori e buona resistenza risulta particolarmente adatto per l'elettroforesi sia di DNA, che
di molecole proteiche. La dimensione dei pori di un gel di agarosio all’1% è stato stimato da 100 nm a 200-500 nm.
Gel a bassa concentrazione (0.1 - 0.2%) sono fragili e quindi più difficili da maneggiare.
Il gel di agarosio ha un potere di risoluzione inferiore rispetto gel di poliacrilammide per il DNA, ma presenta una gamma più
ampia di separazione, ed è quindi usato per frammenti di DNA di dimensioni che possono variare da 50-20.000 bp. Il
limite superiore di risoluzione è di circa 750 kb, ma la risoluzione di oltre 6 Mb è possibile usando pulsed field gel
electrophoresis (PFGE).
Migrazione di acidi nucleici in gel di Agarosio
Fattori che possono influenzare la migrazione degli acidi nucleici:
la dimensione dei pori del gel (concentrazione di gel = % di
agarosio)
dimensioni del DNA da analizzare per elettroforesi
la tensione utilizzata (Voltaggio)
la forza ionica del tampone
la concentrazione di colorante intercalante (ad esempio bromuro di
etidio, se utilizzato durante l'elettroforesi)
Elettroforesi di acidi nucleici in gel di Agarosio
Preparazione del gel
La concentrazione di gel influisce sulla risoluzione e la separazione del DNA. Per una elettroforesi su gel di agarosio standard, una
concentrazione dello 0,8% dà una buona separazione e risoluzione di grandi frammenti di DNA 5-10 kb, mentre il 2% dà una buona
risoluzione per i piccoli frammenti 0.2-1 kb. 1% gel è usato spesso per un elettroforesi standard. La concentrazione del gel è misurata
come percentuale % (in peso di agarosio su volume di tampone utilizzato g/100 ml). Alte percentuali di Agarosio produce gel rigidi e
spesso l’Agarosio non si scioglie in modo uniforme, al contrario i gel con basse percentuali (0,1-0,2%) sono molto molli e non facili da
maneggiare.
Il gel viene preparato sciogliendo la polvere di agarosio in un tampone appropriato, ad esempio Tris/acetato/EDTA (TAE) e Tris/borato/EDTA
(TBE), da utilizzare per l’elettroforesi. I tamponi utilizzati contengono EDTA per inattivare molte nucleasi che richiedono cationi bivalenti per la
loro funzione. L'agarosio viene sciolto nel buffer prima di riscaldarlo fino al punto vicino al punto di ebollizione, ma evitando di farlo bollire.
L'agarosio fuso viene lasciato raffreddare prima di versarlo in un cast per non deformare o rompere il cast, qualora la soluzione di agarosio
fosse troppo calda. Un pettine è posto nel gel per creare pozzetti («spazi vuolti») per il caricamento del campione. l gel deve essere
completamente solidificato prima dell'utilizzo.
Tamponi (buffers)
In generale, il buffer ideale dovrebbe avere una buona conducibilità e produrre poco calore, non surriscaldarsi troppo. Oltre ai buffers di TAE
o TBE, sono stati proposti molti altri tipi di buffer, ad esempio litio borato (LB), poco utilizzato, ecc.; nella maggior parte dei casi il
presupposto logico è applicare una più bassa corrente (=meno calore) e/o mobilità di ioni abbinati, che consente anche una «vita» più lunga
del buffer. Il TAE ha la capacità di tamponamento più basso ma fornisce la migliore risoluzione per il DNA più grandi dimensioni. Ciò
significa una tensione inferiore e un maggiore tempo di migrazione, ma un prodotto migliore. Il tampone borato TBE potrebbe dare più
problemi nella polimerizzazione, e/o interagire con l’RNA. Un tampone Tris-fosfato ha elevata capacità tamponante, ma non può essere usato
se il DNA estratto dalla banda del gel deve successivamente essere utilizzato in reazioni sensibili al fosfato LB è relativamente nuovo ed è
inefficace nel risolvere i frammenti più grandi di 5 kb; tuttavia, per la sua bassa conduttività, potrebbe essere utilizzata una tensione molto più
alta (fino a 35 V / cm), che significa un tempo di analisi più breve per elettroforesi di routine.
Elettroforesi di acidi nucleici in gel di Agarosio
Analisi di acidi nucleici usando elettroforesi in gel di Agarosio
Caricamento di campioni
Una volta che il gel è solidificato, il pettine viene rimosso,
lasciando pozzetti (buchi) vuoti in cui i campioni di DNA
possono essere caricati.
Il buffer di caricamento contiene anche un composto denso,
come il glicerolo (maggiore gravità del glicerolo in soluzioni
acquose), saccarosio, o Ficoll, che aumentano la densità del
campione, in modo che il campione di DNA può depositarsi sul
fondo del pozzetto. Se il campione di DNA contiene etanolo
residuo dall’estrazione, ne può causare la fuoriuscita dal
pozzetto per galleggiamento.
Poiché il DNA non è visibile alla luce naturale, l'avanzamento
della elettroforesi del campione è controllata mediante
l’addizione di coloranti, come Xilene cianolo, Cresol Red,
Orange G, e il blu di bromofenolo utilizzato per monitorare
l'andamento della elettroforesi. Xilene cianolo (colore azzurro)
co-migra con grandi frammenti di DNA, mentre il blu di
bromofenolo (blu scuro) co-migra con i frammenti più piccoli.
Meno usati includono Cresol Rosso e Orange G.
I campioni di DNA vengono caricati con una pipetta.
Elettroforesi di acidi nucleici in gel di Agarosio
Elettroforesi
L’elettroforesi su gel di Agarosio è comunemente eseguita
orizzontalmente in immersione nel tampone. E‘ anche
possibile, ma meno comuni, eseguire l'elettroforesi verticale,
utilizzando una apparecchiatura appropriata. Il buffer usato
per diluire l’Agarosio che costituisce il gel deve essere
lo stesso del tampone di corsa nel serbatoio elettroforesi.
Per una risoluzione ottimale di DNA di dimensioni maggiori
di 2 kb in elettroforesi su gel di norma è da 5 a 8 V/distanza
in cm tra gli elettrodi. La tensione (Voltaggio) può essere
limitata dal fatto che riscalda il gel e può causarne la
fusione, se viene la migrazione viene eseguita ad alta
tensione per un periodo prolungato, soprattutto se il gel
utilizzato è gel di agarosio LMP. Una tensione troppo
elevata può anche ridurre la risoluzione, oltre a causare
striature nella banda quando si analizzano grandi molecole
di DNA (DNA genomico). Un voltaggio troppo basso può
portare ad allargamento (diffusione) della banda quando si
analizzano piccoli frammenti di DNA.
Elettroforesi di acidi nucleici in gel di Agarosio
La velocità di migrazione del DNA è proporzionale alla
tensione applicata: maggiore è la tensione, più velocemente
il DNA si muove. La risoluzione di grandi frammenti di DNA è
comunque inferiore se si applica alta tensione.
La mobilità del DNA può anche cambiare in un campo
instabile, in un campo che è periodicamente invertito (PFGE),
causando field inversion gel electrophoresis (FIGE), per cui
grandi frammenti di DNA si muovono più velocemente di
quelli piccoli.
Anomalie nella migrazione
Un sovraccarico (troppo concentrato) di DNA rallenta la
migrazione di frammenti di DNA, ridurrà la migrazione
elettroforetica DNA in modo dose-dipendente producendo
sbavature.
Gel "smiley»: questo effetto distorto è causato quando la
tensione applicata è troppo alta rispetto alla concentrazione di gel
utilizzato.
Contaminazione - presenza di impurità, come sali o proteine può
influenzare il movimento del DNA.
A
Analisi di acidi nucleici usando coloranti fluorescenti
La colorazione e la visualizzazione
DNA e RNA sono normalmente visualizzati mediante colorazione con bromuro di etidio, che si intercala nel solco maggiore del DNA ed è
fluorescente quando sottoposto a luce UV. Il bromuro di etidio può essere aggiunto alla soluzione di agarosio prima della sua gelificazione, o
il gel DNA può essere colorato più tardi dopo elettroforesi. SYBR Green, GelRed e altri prodotti commerciali simili sono venduti come
alternative meno pericolose rispetto all’etidio bromuro, che è stato dimostrato essere mutageno nei test di Ames, anche se non è stata
effettivamente stabilita la sua cancerogenicità. Il SYBR Green richiede l'uso di un transilluminatore luce blu, ma il DNA colorato con crystal
violet può essere visualizzato con luce naturale, senza l'uso di un transilluminatore UV, ciò costituisce un vantaggio, ma tuttavia non può
produrre una banda intensa.
ethidium bromide
Syber Green
Analisi di acidi nucleici usando coloranti fluorescenti
La colorazione e la visualizzazione
Quando colorato con bromuro di etidio, il gel viene visto con un (UV) transilluminatore a luce ultravioletta. I transilluminatori standard
utilizzano lunghezze d'onda di 302/312-nm (UV-B), tuttavia l'esposizione del DNA alle radiazioni UV per un minimo di 45 secondi è in grado di
produrre danni al DNA e influenzare le procedure successive, ad esempio riducendo l'efficienza di reazione come la trasfezione, la
trascrizione in vitro e PCR. L'esposizione del DNA a radiazioni UV, pertanto deve essere limitata. Usando una lunghezza d'onda
maggiore, di 365 nm (UV-A) si provocano meno danni al DNA, ma produce una fluorescenza molto più debole.
L'apparato transilluminatore può anche contenere dispositivi di cattura delle immagini, come ad esempio una fotocamera digitale o polaroid,
che permettono di avere un’immagine del gel e/o stamparla.
Ethidium Bromide
Quantificazione di DNA usando gel di Agarosio
Un marcatore DNA è caricato vicino ai campioni nel gel per misurare
la lunghezza kb (lunghezza) e ngr (peso) dei frammenti di DNA
Quantificazione di DNA usando gel di Agarosio
Le molecole di DNA più piccole molecole viaggiano più velocemente rispetto a molecole più grandi in gel, e si muovono ad un tasso che è
inversamente proporzionale al log10 del numero di paia di basi (bp). Molecole più grandi vengono risolte meglio utilizzando gel a bassa
concentrazione, mentre le molecole più piccole sono separate meglio in gel ad alta concentrazione.
Kb e ngr
Quantificazione di DNA usando gel di Agarosio
Quantificazione di DNA usando gel di Agarosio
Caricati 100 ng di DNA standard
La concentrazione può essere determinata quando
l’elettroforesi su gel è completata, confrontando
l'intensità del campione di DNA con quella di uno
standard di quantificazione.
Ad esempio, se 2 μl di un campione di DNA diluito
caricato sul gel (PCR) ha la stessa intensità
approssimata dello standard 100 ng, quindi la
concentrazione della soluzione è 50 ng/µl (100 ng
diviso per 2 μl.
Gli Standard usati per la quantificazione devono
essere costituiti da bande di cui una avente la
stessa dimensione del campione di DNA
analizzato e intensità simile. Per visualizzare il
DNA nel gel, si usa una colorazione con un
colorante intercalante come il bromuro di etidio o
SYBR® Verde.
Applicazioni: analisi di DNA plasmidico
Il movimento del DNA può essere influenzato dalla conformazione della
molecola di DNA, per esempio, DNA superavvolto di solito si muove più
velocemente di DNA «rilassata», perché è strettamente avvolto e quindi più
compatto. In una normale preparazione di DNA plasmidico, possono essere
presenti molteplici forme di DNA.
L’elettroforesi su gel dei plasmidi normalmente mostra la forma
«superavvolta» come la banda principale, mentre le altre forme di DNA
forma circolare aperta («lineare») e la forma circolare chiuso («rilassata»)
appaiono come bande minori. La velocità con cui le varie forme si muovono
tuttavia può cambiare usando differenti condizioni di elettroforesi, e la
mobilità del DNA circolare può essere maggiormente influenzata rispetto al
DNA lineare dalle dimensione dei pori del gel.
L’etidio bromuro che si intercala nel DNA circolare può cambiare la carica,
la lunghezza, così come la superhelicity della molecola di DNA, quindi la
sua presenza nel gel durante l'elettroforesi ne può influenzare il movimento.
L’ elettroforesi in gel di Agarosio può essere utilizzata per analizzare il DNA
circolare con diversa tipologia di avvolgimento.
* Nota: la dimensione di un DNA circolare plasmidico non può essere
accuratamente quantificata utilizzando marcatori standard, se non è stato
prima linearizzato con digestrione usando enzimi di restrizione.
Applicazioni
Separazione di frammenti di DNA per l'estrazione e
purificazione
L'analisi dei prodotti di PCR (in diagnosi genetica
molecolare o fingerprinting genetico)
Stima della dimensione delle molecole di DNA tagliati con
enzimi di restrizione (presenti nella mappatura di
restrizione del vettore di clonaggio del DNA)
Southern blotting o Northern blotting (trasferimento di
RNA)
Nucleic acid analized by Polyacrylamide gel electrophoresis (PAGE)
Polyacrylamide gel electrophoresis (PAGE), describes a technique widely used in biochemistry, forensics, genetics, molecular biology and
biotechnology to separate biological macromolecules, usually proteins or nucleic acids, according to their electrophoretic mobility. Mobility is a
function of the length, conformation and charge of the molecule.
As with all forms of gel electrophoresis, molecules may be run in their native state, preserving the molecules' higher-order structure, or a
chemical denaturant may be added to remove this structure and turn the molecule into an unstructured linear chain whose mobility depends
only on its length and mass-to-charge ratio. For nucleic acids (small fragments of dsDNA and oligonucleotides), urea is the most
commonly used denaturant (for ssDNA analysis). For proteins, sodium dodecyl sulfate (SDS) is an anionic detergent applied to protein
sample to linearize proteins and to impart a negative charge to linearized proteins, this procedure is called SDS-PAGE.
Polyacrylamide matrix
Preparing Polyacrylamide gels
The gels typically consist of acrylamide, bisacrylamide, (named native gel) the optional denaturant (SDS or urea), and a buffer with an
adjusted pH. The solution may be degassed under a vacuum to prevent the formation of air bubbles during polymerization. Alternatively,
butanol may be added to the resolving gel (for proteins) after it is poured, as butanol removes bubbles and makes the surface smooth. A
source of free radicals Ammonium persulfate and a stabilizer, such as tetramethylethyldiamine (TEMED), are added to initiate
polymerization. The polymerization reaction creates a gel because of the added bisacrylamide, which can form cross-links between two
acrylamide molecules. The ratio of bisacrylamide to acrylamide can be varied for special purposes, but is generally about 1 part in
35. The acrylamide concentration of the gel can also be varied, generally in the range from 5% to 25%. Lower percentage gels are
better for resolving very high molecular weight molecules, while much higher percentages are needed to resolve smaller proteins. Gels are
usually polymerized between two glass plates in a gel caster, with a comb inserted at the top to create the sample wells. After the gel is
polymerized the comb can be removed and the gel is ready for electrophoresis.
Nucleic acid analized by Polyacrylamide gel electrophoresis (PAGE)
Various buffer systems are used in PAGE depending on the nature of the sample and the experimental objective. The buffers used at the
anode and cathode may be the same or different.
An electric field is applied across the gel, causing the negatively charged proteins or nucleic acids to migrate across the gel away from the
negative electrode (which is the cathode being that this is an electrolytic rather than galvanic cell) and towards the positive electrode (the
anode). Depending on their size, each biomolecule moves differently through the gel matrix: small molecules more easily fit through the pores
in the gel, while larger ones have more difficulty. The gel is run usually for a few hours, though this depends on the voltage applied across the
gel; migration occurs more quickly at higher voltages, but these results are typically less accurate than at those at lower voltages. After the set
amount of time, the biomolecules have migrated different distances based on their size. Smaller biomolecules travel farther down the gel,
while larger ones remain closer to the point of origin. Biomolecules may therefore be separated roughly according to size, which depends
mainly on molecular weight under denaturing conditions, but also depends on higher-order conformation under native conditions.
Chemicals for processing and visualization
The following chemicals and procedures are used for processing of the gel and samples visualized in it:
•
•
•
•
•
•
Tracking dye. As proteins and nucleic acids are mostly colorless, their progress through the gel during electrophoresis cannot be easily
followed. Anionic dyes of a known electrophoretic mobility are therefore usually included in the PAGE sample buffer. A very common
tracking dye is Bromophenol blue (BPB, 3',3",5',5" tetrabromophenolsulfonphthalein). This dye is colored at alkali and neutral pH and is a
small negatively charged molecule that moves towards the anode. Being a highly mobile molecule it moves ahead of most proteins. As it
reaches the anodic end of the electrophoresis medium electrophoresis is stopped. It can weakly bind to some proteins and impart a blue
color. Other common tracking dyes are xylene cyanol, which has lower mobility, and Orange G, which has a higher mobility.
Loading aids. Most PAGE systems are loaded from the top into wells within the gel. To ensure that the sample sinks to the bottom of the
gel, sample buffer is supplemented with additives that increase the density of the sample. These additives should be non-ionic and nonreactive towards proteins to avoid interfering with electrophoresis. Common additives are glycerol and sucrose.
Coomassie Brilliant Blue R-250 (CBB)(C45H44N3NaO7S2; mW: 825.97). CBB is the most popular protein stain.
Ethidium bromide (EtBr) is the traditionally most popular nucleic acid stain.
Silver staining. Silver staining is used when more sensitive method for detection is needed, as classical Coomassie Brilliant Blue
staining can usually detect a 50 ng protein band, Silver staining increases the sensitivity typically 50 times.
Western Blotting is a process by which proteins separated in the acrylamide gel are electrophoretically transferred to a stable,
manipulable membrane such as a nitrocellulose, nylon, or PVDF membrane. It is then possible to apply immunochemical techniques to
visualise the transferred proteins.
DNA/RNA Purification from PAGE
Electroelution












