31/08/2009
Replicazione e genetica virale
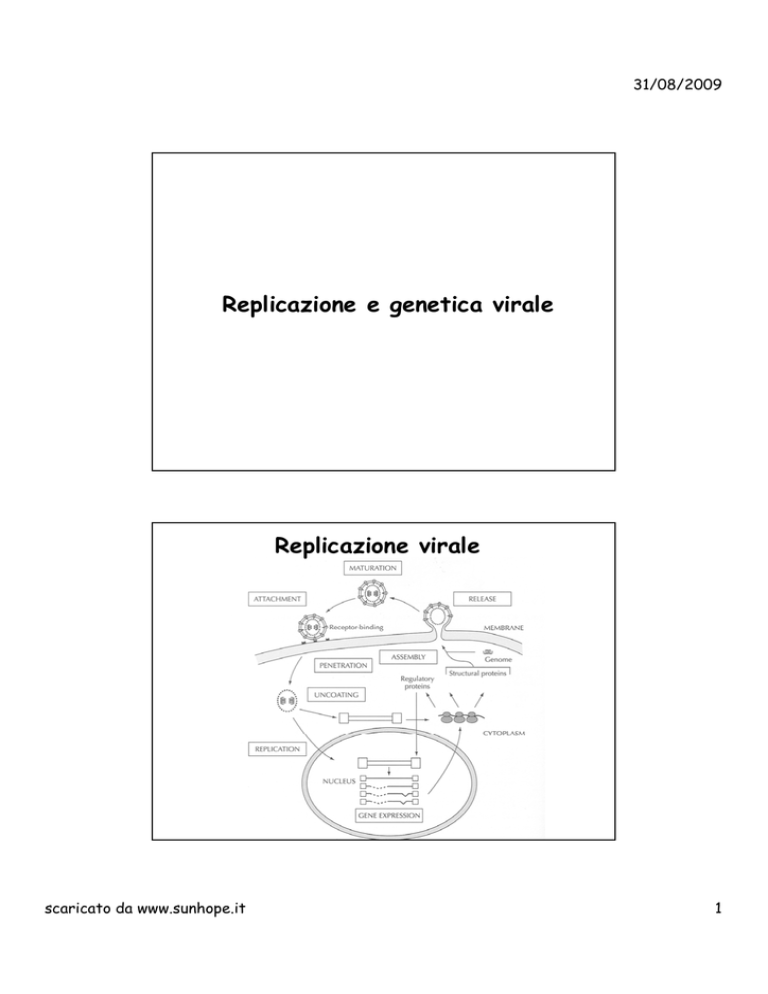
Replicazione virale
scaricato da www.sunhope.it
1
31/08/2009
L’infezione avviene attraverso tre fasi:
1) fase primaria che comprende:
adsorbimento: ancoraggio del virione a specifici recettori
sulla
ll superficie
fi i cellullare
ll ll
per mezzo di siti
i i
stereochimicamente complementari (detti
"antirecettori"). Richiede solo una partecipazione passiva
della cellula;
penetrazione: nel caso dei virus batterici interessa solo
l’acido nucleico genomico (ed alcune proteine interne),
mentre nel caso dei virus dei vertebrati questo fenomeno
implica sempre la partecipazione di tutto il virione;
scapsulamento: esposizione dell’acido nucleico virale, che
nei virus batterici é contemporaneo alla penetrazione,
mentre nei virus dei vertebrati avviene successivamente.
Tre differenti strategie per lo svestimento (uncoating) dei virioni
scaricato da www.sunhope.it
2
31/08/2009
2) fase replicativa che comprende:
- sintesi degli acidi nucleici virali;
- sintesi dell’RNA messaggero;
- sintesi delle proteine virali.
3) fase di rilascio che comprende
assemblaggio con un processo morfogenetico dei vari componenti
virali neosintetizzati;
m t
maturazione:
i n : fuoriuscita
f
i s it d
deii virioni
i i ni d
della
ll p
progenie
ni all
ll’esterno
st n d
della
ll
cellula ospite (a seconda dei virus può avvenire con la lisi della cellula
oppure per gemmazione dalla membrana citoplasmatica, cioé in modo
non traumatico per la cellula).
Subito dopo, o contemporaneamente alla penetrazione, inizia la
cosiddetta "fase di eclisse" che dura per un periodo che va da minuti
a ore, a seconda del tipo di virus. In questa fase i virioni, perdendo la
loro integrità
g
strutturale,, sembrano scomparire
p
alle comuni indagini
g
immunologiche e virologiche. Queste proprietà, tipiche dei virioni
completi, ricompaiono solo in presenza della progenie virale, che può
essere dimostrata anche prima della liberazione dalla cellula
infettata.
scaricato da www.sunhope.it
3
31/08/2009
Genomi Virali.
I genomi virali variano in grandezza da circa 3,500 nucleotidi (nt) e.s.
batteriophago della famiglia Leviviridae (MS2) a circa 280 coppie di
kilobasi (kbp) e.s. la famiglia Herpesviridae (CMV)
Il genoma dei virus (1)
30% dei virus animali ha un
genoma a DNA
5-300 kb
3 – 100 proteine
L’acido nucleico di tutti i virus a
DNA, eccetto i Parvovirus, è a
doppio filamento (ma il DNA degli
Hepadnavirus è parzialmente a
g
filamento quando
q
non è in
singolo
fase di replicazione)
scaricato da www.sunhope.it
4
31/08/2009
Il genoma dei virus (2)
70% dei virus animali ha un genoma
ad RNA
7 – 30 kb
proteine
4 – 18 p
Sempre a singolo filamento,
tranne nei Reovirus
Genomi virali.
• I genomi a RNA sono più piccoli: massimo 30Kb.
Perchè? Le RNA polimerasi virali sono prone
all’errore se paragonate alle DNA polimerasi. La
fedeltà di replicazione potrebbe limitarne la
taglia.
• I genomi a DNA? Fino a 300kb
• Il tipo di genoma e la sua grandezza dettano I
vari step della replicazione virale.
scaricato da www.sunhope.it
5
31/08/2009
Organizzazione dei Genomi
• Genomi molto condensati
• Un’infinità
U ’i fi ità di strategie
t t i per massimizzare
i i
lla
capacità codificante
!
!
!
! !
!
!
!
!
!
!
!
!
Differenti strategie per codificare proteine nei genomi virali
scaricato da www.sunhope.it
6
31/08/2009
Differenti strategie per codificare proteine nei genomi virali
Differenti strategie per codificare proteine nei genomi virali
scaricato da www.sunhope.it
7
31/08/2009
Differenti strategie per codificare proteine nei genomi virali
Diagramma schematico delle diverse strade che i virus devono seguire
per giungere alla sintesi dei propri RNA-messaggeri specifici
scaricato da www.sunhope.it
8
31/08/2009
Tutti i virus sono parassiti del sistema di traduzione
dell’RNA messaggero delle cellule
Classificazione di Baltimore
scaricato da www.sunhope.it
9
31/08/2009
Classe I
La prima classe comprende i virus a DNA bicatenario, cioé la quasi
t t lità dei
totalità
d i virus
i
a DNA.
DNA In
I questo
t caso la
l sintesi
i t i di mRNA
RNA virale
i l può
ò
essere realizzata dalla RNA-polimerasi della cellula ospite, che ha
sede nel nucleo, come avviene per Herpesvirus, Adenovirus e
Papovavirus. Questi generi costituiscono la sottoclasse 1°a e svolgono
la maggior parte degli eventi replicativi nel nucleo della cellula. Gli
altri virus, con genoma a DNA bicatenario, come i Poxvirus e gli
Iridovirus, possiedono una propria RNA-polimerasi associata al
virione, che consente loro di replicarsi a livello del citoplasma. Questi
costituiscono la sottoclasse 1°b.
Strategie replicative dei genomi
Genomi a doppio filamento di DNA (dsDNA)
Classe I
2 fasi: early e late
RNApol cellulare
scaricato da www.sunhope.it
10
31/08/2009
Virus con genomi a DNA (ss/dsDNA)
che usano un passaggio intermedio a singolo filamento di RNA (ssRNA)
DNApol virale nel virione
RNApol cellulare
Transcriptasi inversa virale
Classe II
La seconda classe comprende i virus con genoma a DNA a singolo
filamento positivo (+) come i Parvovirus. La replicazione avviene nel
nucleo e prevede la sintesi del filamento complementare del DNA; la
doppia elica di DNA viene poi usata come stampo per la sintesi degli
RNA messaggeri e dei nuovi genomi virali. A differenza dei virus a
DNA a doppia elica, quelli a filamento singolo codificano soltanto le
proteine del capside.
capside Essi si affidano ai sistemi cellulari per ciò che
concerne la loro replicazione e trascrizione.
scaricato da www.sunhope.it
11
31/08/2009
Herpesviridae
Penetrazione per fusione dell’envelope con la
membrana citoplasmatica. I recettori cellulari
sono rappresentati dall’eparano solfato che si
lega a gC, in seguito un legame più stabile viene
fornito dal legame di gD con diversi recettori
(a seconda del tipo di cellula) che appartengono
alla famiglia del TNF. La fusione avviene a pH
neutro sulla superficie della cellula mediante
interazione con le proteine gH/gL e gB.
D l tegumento sii liberano
Dal
lib
due
d proteine
i
strutturali: 1) VHS (Virino Host Shut-off
protein) che inibisce la sintesi delle proteine
cellulari,
2) α-TIF (α-gene Trans Inducine Factor) che
serve ad avviare la prima fase della
trascrizione del DNA una volta che questo è
stato portato, ancora all’interno del capside, a
livello del nucleo e qui rilasciato.
La trascrizione del DNA in RNAm avviene nel nucleo, in tre fasi distinte, con produzione di RNAm (e proteine) di tipo α
(immediate early), β (early) e γ (late). L’estressione di questi 3 gruppi di geni è finemente controllata da una serie di
meccanismi a feedback. Le proteine α attivano la trascrizione dei geni β e contemporaneamente regolano la propria
espressione.
i
L
Le proteine
t i α e β,
β insieme
i i
sono iin grado
d di regolare
l
l’l’espressione
i
d
deii genii g. Il ciclo
i l è completato
l t t d
dalla
ll
sintesi delle proteine g, alcune delle quali bloccano l’ulteriore sintesi di proteine α e β. La maggior parte dei prodotti dei
geni β sono coinvolti nella replicazione del DNA virale (DNA polimerasi, elicasi/primasi, timidino-chinasi, etc). Questi
enzimi rendono la replicazione del virus del tutto indipendente dall’attività degli enzimi cellulari.
L’acido nucleico sembra replicarsi con un meccanismo a circolo rotante, che prevede la circolarizzazione del DNA ed è
caratterizzato dalla formazione di concatameri, lunghe molecole di dsDNA costituite dall’insieme di monomeri di
lunghezza gnomica. La processazione dei concatameri e la formazione dei genomi virali avvengono al momento
dell’inserzione del DNA nei capsidi in via di formazione.
La maggior parte delle proteine γ sono componenti strutturali che, una volta prodotti nel citoplasma,, rientrano nel nucleo,
dove si assemblano.
L’acquisizione dell’envelope avviene adurante la fuoriuscita del capside dal nucleo: acquisizione dalla membrana interna del
nucleo, vestimento in seguito a fusione con la membrana esterna del nucleo e acquisizione finale nell’apparato del Golgi.
Genomi a singolo filamento di DNA (ssDNA), tipici dei piccoli genomi.
Classe II
RNApol cellulare
DNApol cellulare
scaricato da www.sunhope.it
12
31/08/2009
Classe III
La terza classe comprende i virus con genoma ad RNA bicatenario (es.
Reovirus). Il genoma di questi virus è suddiviso in 10-12 segmenti
contenuti in un doppio capside isometrico. Ogni segmento è detto
"minicromosoma", perché é trascritto separatamente e dà luogo ad un
singolo RNA messaggero monocistronico. Poiché non esistono enzimi
cellulari per la trascrizione diretta di RNA in RNA, sono necessari
enzimi virali associati ai virioni che, dopo la penetrazione e lo
scapsulamento trascrivono il filamento genomico negativo (-) per
produrre
d
mRNA.
RNA
Genomi a doppio filamento di RNA (dsRNA)
Classe III
Più piccoli dei genomi a DNA.
Tutti i virus a dsRNA hanno un genoma costituito da 2
a 12 molecole diverse di RNA (“genoma segmentato”).
RNApol virale associata al capside
scaricato da www.sunhope.it
13
31/08/2009
Genomi a singolo filamento di RNA (ssRNA)
Possono essere divisi in 2 gruppi:
• quelli che funzionano come l’ mRNA (senso positivo)
Picorna, Calici, Corona, Flavi, Togaviridae.
• quelli che sono complementari all’ mRNA prodotto da loro
stessi (senso negativo)
Orthomyxo Paramyxo,
Orthomyxo,
Paramyxo Rhabdo
Rhabdo, Filoviridae
Filoviridae.
• Polarità positiva AUGGCACGA
met-ala-arg
met
ala arg
• Polarità negativa
g
UACCGUGCU
scaricato da www.sunhope.it
14
31/08/2009
Classe IV
La quarta classe comprende i virus con genoma ad RNA a singolo filamento
positivo(+). Di questa classe fanno parte i Poliovirus, i Picornavirus e il virus
dell’epatite
dell
epatite A (sottoclasse IVa).
IVa) All
All’estremitá
estremitá 3
3’ il genoma é poliadenilato
mentre all’estremitá 5’ é legato covalentemente ad una piccola molecola
proteica. Dopo lo scapsidamento, questa proteina viene rimossa da una
proteasi cellulare ed il genoma può quindi legarsi ai ribosomi e funzionare
come RNA messaggero. La trascrizione origina un’unica proteina,
corrispondente alla capacità codificativa totale del virus. Questa molecola
proteica viene detta "poliproteina", perchè in seguito a tagli operati da
proteasi cellulari vengono generate diverse proteine funzionali più piccole.
Una di qu
queste
st è l’RNA-polimerasi
N po m ras RNA-dipendente,
N
p n nt , n
necessaria
c ssar a p
perr la
a
replicazione del genoma, che avviene all’interno di un "complesso
replicativo". Viene sintetizzato un filamento ad RNA negativo(-) che
funziona da stampo. Alla sottoclasse IVb appartengono i Togavirus che
necessitano di due o più cicli di traduzione per produrre l’RNA genomico.
Genomi a singolo filamento di RNA con
polarità positiva (ssRNA)
Classe IV
scaricato da www.sunhope.it
15
31/08/2009
Classe VI
La sesta classe comprende i virus con genoma "diploide" cioé formato da
due identiche molecole di RNA monocatenario tenute insieme in un dimero
speculare a livello delle rispettive estremità 5’ da legami a idrogeno,
mentre le estremità 3’ delle due molecole sono libere. A questa classe
pp
g
i Retrovirus.. In q
questo caso il g
genoma
m ha la stessa p
polarità
appartengono
dell’RNA messaggero, ciò nonostante non funziona da mRNA, ma viene
trascritto in un filamento complementare di DNA da una DNA-polimerasi
RNA-dipendente associata al virione, detta "trascrittasi inversa".
Successivamente una delle subunità di questo enzima funziona da RNasi,
depolarizzando il filamento di RNA genomico presente nell'ibrido
RNA/DNA. Lo stesso enzima agisce poi da DNA-polimerasi DNAdipendente e duplica il filamento di DNA negativo (-). Infine, il DNA a
doppio filamento migra nel nucleo della cellula dove viene integrato in uno
d cromosomi (legame
dei
(l
covalente
l
all DNA
DN cellulare).
ll l
) Questo DNA
DN a doppio
d
filamento che contiene l’informazione genetica virale viene detto "provirus"
per analogia con il profago dei batteri e si comporta come un insieme di
geni cellulari. In particolare, il suo filamento negativo (-) può essere
trascritto in molecole di mRNA ad opera della RNA-polimerasi cellulare.
Virus con genomi a singolo filamento di RNA (ssRNA) che
usano un passaggio intermedio a doppio filamento di DNA
(dsDNA, un provirus)
Classe VI
Transcriptasi inversa virale
RNApol cellulare
scaricato da www.sunhope.it
16
31/08/2009
Classe V
La quinta classe comprende i virus con genoma a RNA a singolo
filamento negativo (-). Di questa classe fanno parte Orthomyxovirus,
Paramyxovirus e Rhabdovirus. In questo caso il genoma non può legarsi
ai ribosomi ma deve prima essere trascritto in molecole complementari
ad opera di una RNA-polimerasi RNA-dipendente associata al virione.
La sottoclasse Va (Orthomyxovirus) presenta un genoma segmentato.
La sottoclasse Vb (Rhabdovirus) presenta un genoma non segmentato.
Genomi a singolo filamento di RNA con polarità negativa (ssRNA)
Classe V
RNApol virale associata al virione
scaricato da www.sunhope.it
17
31/08/2009
Assemblaggio
• L’ assemblaggio implica la raccolta di tutti i
componenti necessari per la formazione del
vvirione
r one maturo in
n un punto part
particolare
colare della
cellula (viroplasma) dove si forma la struttura di
base della particella virale
• Il sito di assemblaggio dipende dal sito di
replicazione e dal meccanismo usato dal virus per
uscire dalla cellula
– Nella maggior parte dei virus a RNA l’assemblaggio
avviene nel citoplasma
– Nella maggior parte dei virus a DNA l’assemblaggio
avviene nel nucleo
scaricato da www.sunhope.it
18
31/08/2009
scaricato da www.sunhope.it
19
31/08/2009
Esempi di Assemblaggio
Assemblaggio dei poliovirus nel
citoplasma della cellula infetta
scaricato da www.sunhope.it
20
31/08/2009
Assemblaggio dell’HSV-1 nel nucleo
Assemblaggio del virus dell’influenza
scaricato da www.sunhope.it
21
31/08/2009
Assemblaggio di un retrovirus da una poliproteina precursore
Assemblaggio di adenovirus nel nucleo
scaricato da www.sunhope.it
22
31/08/2009
Viral assembly at cellular membranes
Impacchettamento del DNA
scaricato da www.sunhope.it
23
31/08/2009
Maturazione
• La maturazione rappresenta lo stadio del ciclo replicativo in cui il
virus diventa infettivo
• La maturazione, generalmente, consiste in cambiamenti strutturali
della particella virale che possono essere conseguenti a tagli
proteolitici specifici delle proteine del capside per formare prodotti
maturi o cambiamenti conformazionali delle proteine durante
l’assemblaggio
• Le proteasi virali sono frequentemente coinvolte nella maturazione
anche se enzimi cellulari o una miscela di enzimi cellulari e virali può
essere utilizzata
• La maturaz
maturazione
one può avven
avvenire
re all
all’ interno
nterno della cellula durante ll’
assemblaggio e la furiuscita o anche all’ esterno dopo che i virus della
progenie sono usciti dalla cellula
Quando i virus sono sprovvisti di
envelope, i virioni neoformati si
accumulano
nella
sede
di
montaggio,
formando
ammassi
talora
cospiqui
che
possono
assumere un ordinato aspetto
tridimensionale
di
formazioni
“cristalline” (fattorie). In questi
casi la liberazione del virus
neoformato nell’ambiente esterno,
si verifica solo per lisi della cellula
scaricato da www.sunhope.it
24
31/08/2009
Microscopia elettronica di fattorie citoplasmatiche in una
cellula infetta con un reovirus
scaricato da www.sunhope.it
25
31/08/2009
Rilascio
Liberazione della progenie virale
a) Virus con pericapside
gemmazione dalla
membrana plasmatica
esocitosi
A
golgi
B
C
D
scaricato da www.sunhope.it
26
31/08/2009
Gemmazione del virus VSV dalla superficie di cellule CHO
Gemmazione di un retrovirus alla
superficie di una cellula infetta
Cellula
C
ll l infetta
i f
da
d virus
i
dell’herpes
d ll’h
simplex osservata al microscopio
elettronico:
dx: nucleocapsidi icosaedrici nel nucleo
sx: virioni extracellulari provvisti di
envelope
scaricato da www.sunhope.it
27
31/08/2009
Poxvirus
scaricato da www.sunhope.it
28
31/08/2009
Assemblaggio e rilascio
Herpesvirus
La genetica interviene in tutti gli aspetti della virologia
L’evoluzione naturale dei virus
Il management clinico delle infezioni virali
La virologia sperimentale
scaricato da www.sunhope.it
29
31/08/2009
Virologia sperimentale
Goal
Comprendere l’organizzazione funzionale del genoma di un virus
Determinazione della struttura di un genoma virale a livello nucleotidico
Isolamento di varianti mutate del virus alterate in ciascun gene
Analisi degli effetti di ciascuna mutazione
sulla replicazione e/o la patogenesi
I primi metodi per identificare, mappare, e caratterizzare i geni virali
1940s, 1950s, e 1960s
L’analisi genetica dei virus consisteva in:
1) casuale, forzato isolamento di numerosi virus individuali mutati
seguito da analisi di complementazione per raggruppare i diversi mutanti
2) ricombinazione
i
bi
i
per d
determinare
t
i
l’l’ordine
di fisi
fisico d
deii genii nell genoma virale
i l
3) analisi fenotipica dei mutanti per determinare la funzione dei geni
Colture cellulari
Inizio dell’era moderna dell’analisi genetica dei virus
1970s
Nuove tecniche
mappatura con enzimi di restrizione
marker rescue
sequenziamento del DNA
ma
Le tecniche classiche continuano
ad essere utilizzate
Tecnologia del DNA ricombinante
Reverse Genetics
scaricato da www.sunhope.it
La struttura del genoma virale viene determinata
e dopo la funzione dei singoli geni viene analizzata
attraverso mutanti specificamente costruiti
30
31/08/2009
Mappatura dei siti di restrizione sul genoma virale
0
2
4
6
8
DNA lineare
tagliato con enzimi
di restrizione A e B
(-)
A
B
5.0
4.5
3.0
2.5
(+)
1.0
“What genes do you need to survive,
and why do you need them?”
scaricato da www.sunhope.it
31
31/08/2009
VARIABILITA’ VIRALE
• C’è p
più diversità tra i virus che in tutti
gli altri esseri biologici esistenti in
natura. Nuove varianti si formano in
continuazione
• E’ impossibile descrivere tutti i gruppi di
virus non solo per la loro quantità,
quantità ma
anche perché in continuo cambiamento
Wild-Type Virus
(ceppo selvatico)
I virus definiti come wild type possono essere molto diversi da quelli
realmente presenti in natura
La genetica virale si fonda sulla coltivazione e l’analisi dei virus in colture cellulari
Isolati virali di campo possono essere soggetti a diverse modificazioni
genetiche durante l’adattamento alla coltivazione in vitro su colture cellulari
In più
I virus
i
d
designati
i
ti wild
ild ttype d
devono essere purificati
ifi ti mediante
di t selezione
l i
di
placca prima di iniziare uno studio genetico
unico background genetico
scaricato da www.sunhope.it
32
31/08/2009
Concetti fondamentali di genetica
Genotipo/Fenotipo
Genotipo
Reale modificazione genetica rispetto al wt
Fenotipo
Manifestazione misurabile della modificazione in
un sistema di analisi specifico
Singolo genotipo
Diversi fenotipi
Secondo il sistema utilizzato
ESEMPIO
Mutazione di un
gene virale
Mutante temperatura
sensibile su
es.: VERO
SI
es.: BHK21 NO
Concetti fondamentali di genetica
Selezione e screen genetico si riferiscono a due metodi fondamentalmente
diversi per identificare singole varianti virali contenute in una popolazione
mista di virus
La selezione implica la presenza di una condizione in cui solo il virus
desiderato è in grado di crescere mentre gli altri vengono soppressi
ESEMPIO
Identificazione di un virus resistente ad un farmaco da una popolazione
mista contenente anche virus sensibili
Infettare un monostrato di cellule e trattarlo con il farmaco
- drug
+ drug
scaricato da www.sunhope.it
33
31/08/2009
Concetti fondamentali di genetica
Screen genetico
Mutante desiderato (es.: tm) + molti altri virus
Coltivati con le stesse condizioni permissive (temperatura ottimale)
Analisi delle singole placche (a temperature più elevate)
Introduzione di un marker fenotipico
Inserimento del gene della betagalattosidasi al posto del gene
da inattivare
Virus wt (placche bianche)
In presenza dell’appropriato
cromogeno (X-gal)
Virus mutato (placche blu)
Concetti fondamentali di genetica
Essenziale/Nonessenziale
Essenziale
Nonessenziale
Necessariamente richiesto per la replicazione in specifiche
condizioni
Non richiesto p
per la replicazione
p
in specifiche
p
condizioni
Eventualmente alcuni geni possono essere essenziali per la replicazione
nell’ospite naturale, ma non per la replicazione in vitro
Gene mutato
Isolamento
Analisi funzione
Gene
nonessenziale
Facile
(il gene può essere deleto)
Difficile (no fenotipo)
Gene
essenziale
Condizioni difficili da
trovare
Caratterizzazione precisa
della mutazione causa del
difetto replicativo
scaricato da www.sunhope.it
34
31/08/2009
Mutazioni
Mutazioni Spontanee
Mutazioni Indotte
Mutazioni doppie
pp
Mutanti doppi (o multipli) possono contenere più di una
mutazione per portare un fenotipo
Mutazioni Spontanee
Sia i virus a DNA che ad RNA possono andare incontro a mutazioni spontanee
RNA viruses > DNA viruses
103 - 106 per ciclo replicativo
Trascrittasi inversa/
RNA polimerasi RNA-dipendente
108 - 1011 per ciclo replicativo
DNA polimerasi DNA-dipendente
funzione proofreading
No proofreading
Differenze nel tasso di frequenza di mutazioni spontanee
Biologia del virus
Analisi genetica dei virus
I virus RNA in natura esistono come: “quasi-species
scaricato da www.sunhope.it
35
31/08/2009
Mutazioni indotte
La frequenza di mutazioni può comunque essere bassa che indurre mutazioni
è necessario per isolare virus mutati
normalmente
single-base changes
Mutageni chimici
in vivo
in vitro
Analoghi nucleosidici
Alterazione dell acido nucleico
trattando i virioni in assenza di
replicazione
idrossilamina
acido nitroso
agenti alchilanti
Devono essere incorporati
durante la replicazione del
virus
Modificazioni chimiche di
specifiche
p
basi causano
disaccoppiamento che porta
ad mutazioni missenso
nitrosoguanidine
g
mutazioni missenso
L’uso di mutageni chimici porta ad un aumento della frequenza delle
mutazioni di varie centinaia di volte
0.5% della popolazione totale di virus
Genotipo mutato
Sostituzione di base
GCG
GCA
Ala
Ala
Silente
GAC
GAG
Asp
Glu
Missenso
Delezione/inserzione
CUC-AGC-GUU
DEL
CUC-GUU
Leu Val
UAA
Val
INS
CUC-AGC-CAU-GUU
Leu Ser His
Val
Del/Ins di triplette o multipli
CUC-AGG-UU
UAG
Leu Ser
Leu Arg
CUC-AAG-CGU-U
Leu Lys Arg
Trp
Stop
Del/Ins di singole basi
Non-senso
Mutazioni non-senso, frameshift, grosse delezioni/inserzioni in frame:
inattivazione del gene
Mutazioni missenso: cambiamenti nel fenotipo (es.: resistenza farmaci)
scaricato da www.sunhope.it
36
31/08/2009
Fenotipo mutato
Goal
Funzione di ciascun gene virale
Inibiscono la replicazione
p
inattivando uno specifico gene
Mutanti utili
Necessità di coltivazione
Mutazione non letale
Fenotipi
Spettro
p
d’ospite
p
Nonsense
Temperatura-sensibili
Dipendenti dai farmaci
Morfologia di placca
Mutanti virali per spettro d’ospite
Virus mutante
Virus wt
Cellula A
Cellula B
Cellula A
Cellula B
SI
NO
SI
SI
Crescita
Naturale o ingegnerizzato
Raro
Delezione di un gene essenziale
Creazione di una linea cellulare (su cui coltivare il virus)
che esprime in modo costitutivo o transiente il gene deleto
in modo da fornire al virus la funzione mancante
scaricato da www.sunhope.it
37
31/08/2009
Mutanti temperatura-sensibili
Virus mutante
Bassa Temp.
Crescita
SI
Alta Temp.
NO
Virus wt
Bassa Temp. Alta Temp.
SI
SI
Generalmente mutazioni in un singolo aminoacido che
rendono la proteina target instabile a temperature più
alte rendendo il gene non funzionale
Metodo di selezione
Mutagenesi random indotta e poi screening
alle due temperature
p
Raccolta e test delle singole placche
Lo screening può essere reso più facile dall’utilizzo ddella tecnica
dell’allargamento delle placche: si coltivano i virus a temperatura
permissiva, poi si spostano a temperatura più elevata (non permissiva
per i mutanti) e si selezionano le placche che restano della stessa taglia
Farmaco resistenza/dipendenza
Diverse sostanze ad attività antivirale
Maggiore utilità
(inibiscono diverse famiglie virali)
sono state descritte
Antivirali specifici per un singolo prodotto genico
ESEMPI
G nidin
Guanidine
p li 2C NTPase
polio
NTP s
Acyclovir
herpes simplex virus thymidine
kinase e DNA polymerase
Amantidine
influenza virus M2 (proteina integrale di membrana)
IBT (Isatin-βthiosemicarbazone)
due geni dei poxviruses coinvolti nella trascrizione virale
If
farmacii più
iù utili
ili sono quelli
lli che
h inibscono
i ib
lla crescita
i su colture
l
cellulari
ll l i (trattate
(
col farmaco) del wt permettendo la selezione delle placche formate da particelle
virali resistenti
Gli effetti tossici devono essere minimali per consentire la sopravvivenza delle cellule
Utilizzo:
scaricato da www.sunhope.it
a) marker di selezione
b) identificazione del target o meccanismo
d’azione di un farmaco antivirale
38
31/08/2009
Diluzioni seriali in base 10 del virus wt
o
del mutante temperatura sensibile ts56
40°C temperature non-permissiva per ts56
Wt virus forma placche sia a 31°C
sia a 40°C, ma la formazione di
placche è inibita dal’IBT
ts56 contiene una mutazione missenso
che lo rende incapace di crescere a
40°C, ma è anche dipendente dalla
presenza di IBT, infatti cresce a
40°C solo in presenza di IBT
Mutanti spontanei resistenti all’IBT
nel wt virus stock (placche a 10–3 e
10–4 in presenza di IBT
Morfologia di placca
Mutanti con diversa morfologia di placca sono facilmente distinguibili dal wt
Le placche provocate dai virus mutati saranno di maggiori o minori
dimensioni rispetto al wt
Esempio
Mutanti sinciziali
Virus erpetici
Difetto nelle
Glicoproteine
di membrana
wt
scaricato da www.sunhope.it
Estesa fusione
delle cellule infette
Gruppi di cellule arrotondate
Con scarsa fusione
39
31/08/2009
Reversion
Gene
V
A
mutante ts
Gene
A
V
Mutante del mutante ts
Reversione: il risultato di una mutazione spontanea che porta
il genotipo del virus al genotipo originale
Frequenza
Riflette la frequenza degli errori degli enzimi
di replicazione
Mutazioni missenso
Delezioni
Estremamente rare (intera sequenza di nucleotidi da rimpiazzare)
Analisi genetica dei mutanti
Complementazione
Serve a determinare se due mutanti virali hanno subito
mutazione nello stesso gene o in geni differenti
A
A
Virus mutato A
B
Virus mutato B
p
Condizioni permissive
np
Condizioni non-permissive
A+ B
B
2
3
np
1
Raccolta del virus
A
A+ B
B
np
p
Indice di Complementazione (CI)
Titolo (A+B)p – Titolo (A+B)np
Titolo (A)p + Titolo (B)np
scaricato da www.sunhope.it
=
Titolazione
CI
40
31/08/2009
Condizioni di crescita non permissive
Complementazione?
Mutante A
Gene X
Mutante B
Gene Y
A fornisce il gene Y a B
B fornisce il gene X ad A
SI
Mutante A
Gene X
Mutante B
Gene X
Il gene X viene sempre a mancare
NO
Complementazione: CI maggiore di 1
Complementazione qualitativa
ts12, ts15, ts28, ts54, ts61
Stesso gruppo di complementazione?
Esperimento
Monostrati di African green
monkey kidney cell line BSC40
Infezione (0.03 pfu/cell) con
virus mutati singoli o in coppie
Controllo ts7 appartenente ad un
diverso gruppo di complementazione
Risultati
Singoli virus
NO
Infezioni doppie
NO
Infezioni miste con ts7
SI
ts12, ts15, ts28, ts54, ts61 appartengono allo stesso gruppo di complementazione
scaricato da www.sunhope.it
41
31/08/2009
Complementazione
Non solo gruppi di complementazione
Fondamentale per: crescita di virus deleti ingegnerizzati mediante infezione
di cellule (a loro volta ingegnerizzate) che forniscono il gene mancante
Interazione genetica:
Due tipi di complementazione
• Complementazione allelica (intragenica):
quando differenti mutanti hanno difetti
complementanti nello stesso gene
• Complementazione non allelica
((intergenica):
g
) risultante da da difetti in
geni diversi
scaricato da www.sunhope.it
42
31/08/2009
Ricombinazione
Sequenze di acido nucleico da due virus
Parentali genotipicamente diversi vengono
scambiate
Ricombinazione
La progenie contiene sequenze di entrambi i virus
Nei virus esistono tre diversi meccanismi di ricombinazione
DNA
Rottura fisica e ricongiunzione tra le due molecole
di DNA attraverso regioni contenenti sequenze omologhe
RNA non-segmentato
Picornavirus,, coronavirus,,
Togavirus e retrovirus
(efficiente ricombinazione)
RNA segmentato
Ricombinazione durante la replicazione dove
attraverso il meccanismo (copy choice) viene
alternativamente scelto il template
Riassortimento dei vari segmenti di RNA
COPY CHOICE
RECOMBINATION
scaricato da www.sunhope.it
43
31/08/2009
COPY CHOICE
RECOMBINATION
COPY CHOICE RECOMBINATION
scaricato da www.sunhope.it
44
31/08/2009
RIASSORTIMENTO
Riassortimento dei segmenti di RNA del virus dell’influenza
Polyacrylamide gel degli
RNA marcati con 32P
e autoradiografia
scaricato da www.sunhope.it
45
31/08/2009
RICOMBINAZIONE
Ricombinazione classica
Comune nei virus a DNA
RICOMBINAZIONE - SOME USES
scaricato da www.sunhope.it
46
31/08/2009
Ricombinazione mediante trasfezione di DNA plasmidico ed infezione con HSV-1
di cellule CR1 (cellule Vero che esprimono stabilmente gH)
HSV - SynK∆gH
UL21
UL23
UL53
LacZ
A
V
Mutazione in gH da analizzare una volta inserita in HSV-1
UL22 - gH
aa 280
CMVp
pIMPLgHMut280
fOri
AmpR
p
HSV – SynKgHMut280
UL21
UL23
UL53
UL22 - gH
A
Fenotipo: Syn/White
HSV – SynKgHM280
V
UL53
UL22
A
HSV - ∆gH
V
Syn mutant
LacZ
flank seq.
UL21
1 Kb
UL22
315 bp
UL22
flank seq.
UL23
Fenotipo: Non-Syn/Blue
HSV - SynK
UL53
UL22
A
HSV - ∆gHpA
V
Syn mutant
LacZ
flank seq.
UL21
scaricato da www.sunhope.it
flank seq.
UL23
47
31/08/2009
Selezione
Selezione
Co-infezione a 10 MOI (HSV-1∆gHpA e HSV-1 SynK) su cellule CR1
24h
Raccolta progenie viruale e preparazione di diverse aliquote
Titolazione su CR1
Risultato
3.3 x 107 pfu/ml
7,2%
(% di ricombinazione)
Syn/Blu
Infezione di cellule CR1 con progenie virale
24h
Ricoprire le cellule con agarosio +X-gal
6-8h
Raccolta delle placche blu sinciziali
Risultato
10 placche blu sinciziali raccolte
1 selezionata
scaricato da www.sunhope.it
48
31/08/2009
Selezione
Infezione di cellule CR1 con placca selezionata
24h
Ricoprire le cellule con agarosio +X-gal
6-8h
Raccolta delle placche blu sinciziali
Risultato
1 selezionata
10 placche blu sinciziali raccolte
2.1 x
104
pfu/ml
20% Blu/Syn
La raccolta delle placche viene effettuata almeno 3-4 volte
Infezione di cellule CR1 con placca selezionata
24h
Ricoprire le cellule con agarosio +X-gal
6-8h
Raccolta delle placche blu sinciziali
Risultato
10 placche blu sinciziali raccolte
8 x 104 pfu/ml
HSV – SynK∆gH
1 selezionata
100% Blu/Syn
108 pfu/ml
Southern blotting di ∆gHpASynK
scaricato da www.sunhope.it
49
31/08/2009
Costruzione di virus mutato
Trasfezione delle cellule
con il DNA plasmidico
contenente il gene mutato
da sostituire
DNA
Infezione con il
virus ricombinante
Virus
HSV - SynK∆gH
UL23
UL21
LacZ
UL53
A
V
UL22-gH
CMVp
aa 280
pIMPLgHMut280
Selezione delle placche
sinciziali bianche (il gene
LacZ viene sostituito da gH)
fO i
fOri
AmpR
HSV – SynKgHMut280
UL23
UL21
UL22-gH
UL53
A
V
Southern blotting di
HSV-1SynKpAgHM280
scaricato da www.sunhope.it
50


![Lezione 15 Virus [modalità compatibilità]](http://s1.studylibit.com/store/data/000771737_1-84b1cca561c5813066d1b76125338a98-300x300.png)









