Regolazione
dell’espressione genica
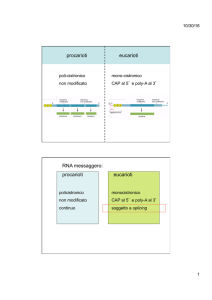
I promotori batterici hanno due sequenza consenso
distinte
Trascrizione nei
procarioti
Regolazione dell’espressione genica nei
procarioti
Il modello dell’operone di Jacob-Monod per la regolazione
dei geni:
Proposto nel 1959, questo modello introduceva il concetto
di geni regolatori che codificano per prodotti (proteine) che
controllano altri geni.
L’operone lac è un esempio di controllo negativo degli
enzimi coinvolti nel metabolismo del lattosio. Tuttavia,
questo operone viene anche regolato da un controllo
positivo.
Il modello dell’operone ha portato alla scoperta degli
mRNA.
Modello del’operone di Jacob e Monod
Esperimento (1966)
di Gilbert e MüllerHill che dimostra che
il repressore lac, una
proteina, si lega al
DNA dell’operatore
Regolazione dell’operone del lattosio (lac)
Nei batteri i geni sono organizzati in operoni, che
sono unità di espressione e di regolazione dei geni
batterici, che comprendono geni strutturali ed
elementi di controllo nel DNA che vengono
riconosciuti dai prodotti dei geni regolatori.
I geni di un operone sono sotto il controllo di un solo
promotore e si produce un singolo trascritto di
mRNA o “mRNA policistronico”.
Le caratteristiche dell’operone lac illustrano i
principi fondamentali universali della regolazione
dei geni.
Regolazione
dell’operone lac
da parte del
glucosio e del
lattosio
Uso in biologia molecolare del promotore lac
e del gene lac Z
Gene lac Z -galattosidasi X-gal prodotto
colorato (blu): strategie di screening di colonie
batteriche trasformate con DNA ricombinante
(clonaggio); usato come gene reporter per lo studio
della regolazione genica in animali transgenici o
cellule in coltura.
Il meccanismo trascrizionale del promotore lac è
usato in indurre l’espressione di proteine
etererologhe in E. coli (uso di IPTG per indurre
l’espressione della proteina ricombinate).
Meccanismi d’azione dei regolatori
trascrizionali
Legame cooperativo delle proteine al DNA: importante
sia nei procarioti che negli eucarioti; es. della proteina CAP che
ha un dominio di legame al DNA ed un dominio detto “regione di
attivazione” che entra in contatto con la RNA polimerasi che si
lega al promotore.
Modificazioni allosteriche e legame al DNA: ci sono due
esempi, 1) il legame del cAMP alla proteine CAP che induce un
cambiamento allosterico in quest’ultima per cui si lega più
saldamente al DNA; 2) il legame dell’allolattosio o dell’IPTG alla
proteina repressore dell’operone lac che induce un cambiamento
allosterico nel repressore che fa diminuire la sua capacità di
legarsi al DNA.
Formazione di anse da parte del DNA: la formazione di
anse di DNA permette a proteine, anche distanti, legate sul DNA
di interagire con la DNA polimerasi legata al promotore.
Complesso
CAP-DNA
L’operone del Triptofano (trp)
La regolazione dell’operone del trp nei batteri è un
classico esempio di attenuazione trascrizionale.
Due meccanismi di attenuazione:
Controllo post-trascrizionale mediante ripiegamento
differenziale dell’mRNA.
Controllo dell’inizio della trascrizione dell’operone trp
mediante il legame di un repressore, attivato dal
legame con il triptofano, a siti operatori, con
conseguente blocco dell’accesso della RNA polimerasi
al promotore trp.
Attenuazione
trascrizionale
dell’operone
trp di E. coli
Regolazione della trascrizione negli
eucarioti
Concetto di elementi cis-agenti and trans-agenti del
DNA e fattori trans-agenti.
Fattori che intervengono nella regolazione della
trascrizione negli eucarioti:
Interazioni DNA-proteina;
Interazioni proteina-proteina;
Struttura della cromatina;
Architettura del nucleo;
Compartimentazione cellulare.
Il controllo dell’espressione genica negli eucarioti
può essere
esercitato a molti livelli oltre che a livello
trascrizionale.
Regolazione della trascrizione negli
eucarioti
La RNA polimerasi II è responsabile della trascrizione
della grande maggioranza dei geni eucariotici:
Geni codificanti per mRNA;
Geni codificanti per snoRNA;
Geni codificanti per alcuni snRNA;
Geni codificanti per microRNA.
Negli eucarioti esistono altre due RNA polimerasi: RNA
polimerasi I localizzata nel nucleolo responsabile della
sintesi dell’rRNA grande; RNA polimerasi III si trova nel
nucleoplasma ed è responsabile della sintesi dell’tRNA,
dell’rRNA 5S e di alcuni snRNA.
Elementi regolatori dei geni codificanti
proteine
Fattori di trascrizione (attivatori o repressori)
CREB, CTCF, FOG-1, GATA-1, NF-E2, NF-B, Sp1.
Macchinario generale della trascrizione RNA
pol II, TFIIB, TFIID (TBP + TAF), TFIIE, TFIIF, TFIIH,
Mediatore.
Coattivatori e corepressori complessi di
modificazione della cromatina (HAT, HDAC, CBP, HMT,
LSD1), complessi di rimodellamento della cromatina
(SWI/SNF, ISWI, SWR1).
Fattori di allungamento FACT, allungatore, TFIIS.
Le unità trascrizionali (RNA pol II)
degli eucarioti
10 kb
2 – 3 kb
Le unità trascrizionali (RNA pol II)
degli eucarioti
Elementi regolatori cis-agenti degli eucarioti
multicellulari:
Elementi promotori (vicini al punto di inizio della
trascrizione) nucleo del promotore + elementi
prossimali del promotore (elementi a monte del
promotore od elementi regolatori a monte);
Elementi regolatori a lungo raggio.
Il nucleo del promotore
Sequenza di 60 bp di DNA sovrapposta al sito +1;
E’ il sito di riconoscimento per la RNA pol II e per i
fattori generali di trascrizione (TFII).
Motivi del nucleo del promotore della RNA pol II
Promotore
Posizione
Fattore di trascrizione
Sequenza consenso
Elementi a monte del nucleo del promotore
BRE
TATA Box
Iniziatore
-37 a -32
-31 a -26
-2 a +4
TFIIB
TBP
TAF1 e TAF2
(G/C)(G/C)(G/A)CGCC
TATA(A/T)AA(G/A)
PyPyA+1N(T/A)PyPy
Elementi a valle del nucleo del promotore
MTE
+18 a + 27
TFIID
DPE
+28 a + 32
TAF9 e TAF6
C(G/A)A(A/G)C(G/C)
(C/A/G)AACG(G/C)
(A/G)G(A/T)(C/T)(G/A/
C)
Elementi prossimali del promotore
CAAT box
GC box
-200 a -70
-200 a -70
CBF, NF1, C/EBP
Sp1
CCAAT
GGGCGG
CBF: fattore che lega CAAT;
C/EBP: proteina che lega CAAT/enhancer
Elementi regolatori a lungo raggio
Questi elementi degli eucarioti multicellulari comprendo:
Enhancer;
Silenziatori;
Isolatori;
Regioni di controllo del locus (LCR);
Regioni di attacco alla matrice (MAR).
Alcuni enhancer che sono negli introni contribuiscono
all’espressione tessuto-specifica enhancer nel secondo
introne del gene dell’apolipoproteina B (apoB delle LDL).
Enhancer e silenziatori
Questi elementi sono di solito a 700 – 1000 o più bp
di distanza dal sito di inizio della trascrizione.
Gli enhancer si possono trovare sia a monte che a
valle del gene, o dentro un introne, e possono
funzionare in entrambi gli orientamenti rispetto al
promotore.
Un enhancer è lungo 500 bp e contiene 10 siti di
legame per fattori di trascrizione diversi.
I silenziatori sono elementi simili agli enhancer, ma
reprimono l’espressione del gene.
Enhancer
e
Silenziatori
Isolatori
Un isolatore è una sequenza di DNA, di solito lunga
300 – 2000 bp, che ha la funzione di:
Marcatore di un confine di cromatina, tra regioni di
eucromatina ed eterocromatina;
Attività di blocco di un enhancer o di un silenziatore.
Contiene un gruppo di siti di legame per proteine
che legano sequenze specifiche di DNA.
Isolatori
Gli elementi isolatori sono riconosciuti da almeno 3 proteine diverse che
legano il DNA: fattore che lega CCCTC (CTCF) ed i fattori stimolatori a monte
(USF) 1 e 2. CTCF media l’attività di blocco dell’enhancer
dell’
mentre USFs
reclutano enzimi che modificano la cromatina.
Regioni di controllo del locus (LCR)
Le LCR sono sequenze di DNA che organizzano e
mantengono un dominio funzionale di cromatina attiva e
fanno aumentare la trascrizione dei geni a valle.
Siti ipersensibili (HS) alla DNasi a monte del gruppo dei
geni della -globina.
Le LCR sono presenti in altri loci genici fra cui: i gruppi di
geni che codificano le -globine, i pigmenti visivi, le
proteine del complesso maggiore di istocompatibilità, gli
ormoni della crescita umani, le serpine, le citochine delle
cellule T helper di tipo 2.
Regioni di controllo del locus (LCR)
L’intero locus dei geni della -globina occupa 200
kb.
La LCR è necessaria per la trascrizione ad alto livello
di tutti i geni della famiglia della -globina.
La regolazione della formazione di “anse” di
cromatina è il meccanismo che controlla
l’espressione durante lo sviluppo dei geni della globina.
La LCR interagisce con un solo promotore genico
alla volta, così da garantire l’espressione differenziale
dei diversi geni durante lo sviluppo.
Regioni di attacco alla matrice (MAR)
L’organizzazione tridimensionale della cromatina ha anche
un ruolo centrale nel controllo della trascrizione negli
eucarioti.
Organizzazione in anse indipendenti della cromatina. La
formazione di queste anse dipende da elementi specifici di
sequenza del DNA che sono sparsi in tutto il genoma ad
intervalli di 5 – 200 kb.
Queste sequenze di DNA sono chiamate regioni di attacco
alla matrice (MAR).
Le MAR organizzano il genoma in 60000 anse di
cromatina con una dimensione media di 70 kb.
Regioni di attacco alla matrice (MAR)
I geni attivi tendono a far parte di domini ad ansa piccoli,
mentre quelli inattivi sono associati a domini più grandi.
Più del 70% delle MAR sono ricche di sequenze AT
Le MAR si trovano vicino agli enhancers. Conferiscono
specificità tissutale e controllo durante lo sviluppo
dell’espressione genica reclutando fattori di trascrizione e
richiamando gli enzimi di rimodellamento della cromatina.
MAR strutturali servono come ancore; MAR funzionali
sono dinamiche e aiutano a portare i geni sulla matrice
nucleare.
Modello della regolazione trascrizionale da parte delle MAR
Macchinario generale (basale) della trascrizione
Componenti:
RNA polimerasi II (12 subunità) sintetizza
RNA in 5’ 3’ ed esegue correzione delle bozze del
trascritto nascente;
Fattori generali di trascrizione 5 fattori,
TFIIB, TFIID, TFIIE, TFIIF e TFIIH, sono
responsabili del riconoscimento del promotore e
dell’apertura della doppia elica del DNA;
Mediatore complesso di 20 subunità; trasduce
informazioni regolatrici da attivatori e repressori alla
RNA pol II.
I fattori di trascrizione
La regolazione dell’attività trascrizionale dei geni
avviene mediante cambiamenti della quantità o
dell’attività dei fattori di trascrizione.
Questi fattori influenzano la velocità di trascrizione
dei geni “target” positivamente o negativamente
tramite interazioni specifiche con gli elementi
regolatori del DNA e tramite interazioni con altre
proteine.
Meccanismi di regolazione della quantità e dell’attività dei
fattori di trascrizione
Meccanismi con cui agiscono fattori di trascrizione
attivatori per aumentare l’attività trascrizionale
1) Stimolazione del reclutamento e del legame dei
fattori generali di trascrizione e della RNA pol II sul
nucleo del promotore per formare un complesso di
preinizio.
2) Induzione di un cambiamento conformazionale od
di una modificazione post-traduzionale che stimola
l’attività enzimatica del macchinario generale di
trascrizione.
3)Interazione con i complessi di rimodellamento e di
modificazione della cromatina per permettere una
migliore accessibilità dei fattori generali di trascrizione
o di attivatori specifici al DNA.
Domini modulari dei
fattori di trascrizione
Fattori di trascrizione: motivi del dominio
che lega il DNA
Motivo elica-giro-elica (HTH) (es. repressore del trp,
proteina CAP, repressore lac, ecc.) l’omeodominio (60
amminoacidi) è una variante del motivo HTH classico che è
presente in molti fattori di trascrizione che regolano lo
sviluppo (anche nei mammiferi).
Fattori di trascrizione: motivi del dominio che
lega il DNA
L’omeodominio: un -elica contatta il solco maggiore del DNA
riconoscendo una sequenza di 6 pb (come nel HTH), mentre un braccio
flessibile interagisce specificamente con il solco minore del DNA.
Fattori di trascrizione: motivi del
dominio che lega il DNA
Motivo a dita di zinco: deriva il nome dallo schema della
sua struttura un atomo di Zn coordina dei residui di
cisteine e di istidine, formando un ansa che ricorda la
forma di un dito. Struttura diffusa in molte famiglie di
attivatori trascrizionali (es. fattore Sp1, recettori degli
ormoni steroidei).
Dominio a dito di Zn del recettore
dei glucocorticoidi (GR)
Motivo a dita di Zn
Fattori di trascrizione: motivi del
dominio che lega il DNA
Domini a cerniera lampo (leucine zipper): due lunghe strutture ad
-elica che contengono, contemporaneamente, il dominio di
dimerizzazione ed il dominio di legame al DNA. Queste strutture
possono formare sia omodimeri che eterodimeri.
Domini a cerniera di leucina
Questo motivo è stato descritto per la prima volta nella
proteina C/EBP che lega CAAT box presente in molti
enhancer.
Appartiene a questo gruppo di attivatori la proteina AP-1,
un eterodimero formato dall’unione di due protooncogeni
c-Jun e c-Fos, che regola l’espressione genica in risposta a
vari stimoli, quali, stress, infezioni virali e batteriche, e
l’attivazione da parte delle citochine.
Domini elica-ansa-elica basico (BHLH)
E’ una struttura caratterizzata da due -eliche connesse da un ansa. I
fattori con questo motivo sono in genere eterodimeri, formati da
un’elica più lunga che lega il DNA attraverso amminoacidi basici.
Appartengono a questa famiglia molti attivatori che hanno un ruolo
nello sviluppo, come Myo-D che svolge una funzione fondamentale nel
differenziamento delle cellule muscolari. Un altro esempio è la
proteina c-Myc, che è coinvolta nella trasformazione tumorale (linfoma
di Burkitt).
Controllo combinatoriale della trascrizione
Gli attivatori e gli inibitori sono coinvolti nella regolazione
di numerosi geni.
E’ la combinazione tra diversi attivatori ed inibitori che
rende molto specifico il meccanismo di regolazione.
Sequenze di importazione ed esportazione
nucleare
Il traffico fra nucleo e citoplasma avviene attraverso i complessi
dei posi nucleari (NPC).
I fattori di trascrizione, sintetizzati nel citoplasma, devono
attraversare questi pori per giungere nel nucleo.
Le proteine sono indirizzate al nucleo da una sequenza
amminoacidica specifica chiamata sequenza di localizzazione
nucleare (NLS); ne esistono diverse, ma le più studiate sono le
sequenze ricche di a.a basici (Arg e Lys).
Per l’uscita dal nucleo, le proteine devono anche avere una
sequenza di esportazione nucleare (NES); le meglio
caratterizzate sono le piccole sequenze idrofobiche ricche di Leu.
Per il passaggio attraverso i pori le proteine con sequenze di
localizzazione e/o esportazione, si avvalgono di una famiglia di
recettori solubili, importine od esportine (carioferine).
Importazione
nucleare regolata
I recettori degli ormoni
steroidei
Importazione nucleare regolata di NF-B
Risposta all’cAMP e fattore CREB
P-CREB si lega a siti sul DNA chiamati
CRE (cAMP Response Element).
CREB è strettamente correlato, sia per
struttura che per funzione, a CREM
(cAMP Response Element Modulator)
ed a ATF-1 (Activating Transcription
Factor-1).