INDICE
PRIMA PARTE: ASPETTI GENERALI
1 INTRODUZIONE………………………………………………….3
2
1.1 HIV: IDENTIFICAZIONE
3
1.2 HIV:CLASSIFICAZIONE
4
MORFOLOGIA E STRUTTURA DEL VIRIONE………………7
2.1
STRUTTURA DEL VIRIONE
7
2.2
STRUTTURA ED ORGANIZZAZIONE DEL GENOMA VIRALE
9
2.2.1 Long terminal repeat
2.2.2 Geni strutturali
2.2.3 Geni regolatori
2.2.4 Geni accessori
2.3
11
13
14
15
ENV
17
2.3.1 La gp120 o dominio SU
2.3.2 La gp41 o dominio TM
2.3.3 Variabilità del gene env e del dominio V3
3
4
CICLO REPLICATIVO…………………………………………..28
3.1 ADSORBIMENTO E PENETRAZIONE
28
3.2
35
SINTESI ED INTEGRAZIONE DEL DNA VIRALE
3.3 ESPRESSIONE E REGOLAZIONE DEI GENI VIRALI
37
3.4
38
ASSEMBLAGGIO E LIBERAZIONE DEL VIRUS
PATOGENESI E STORIA NATURALE DELL’INFEZIONE...39
4.1 SINTESI DEL DECORSO DELL’ INFEZIONE DA HIV
4.1.1
4.1.2
4.1.3
4.1.4
5
20
22
23
Infezione primaria
Fase di latenza clinica
Progressione della malattia: AIDS conclamato
Soggetti lungo sopravviventi
41
41
42
43
45
TERAPIA E CONTROLLO……………………………………..48
5.1 FARMACI ANTIVIRALI
48
5.2 RESISTENZE AI FARMACI
50
5.3 INIBITORI DELL’ENTRY
53
1
SECONDA PARTE: SPERIMENTAZIONE ESEGUITA E RISULTATI
DELLA RICERCA
6 RAZIONALE DELLO STUDIO…………………………………..54
7 MATERIALI E METODI…………………………………………57
7.1 COSTRUZIONE DEL VETTORE pNLmodΔV3GFP
57
7.2 ORIGINE DELLE REGIONI V3
59
7.3 SEQUENZIAMENTO DIRETTO DELLE REGIONI V3
60
7.4 CLONAGGIO MOLECOLARE DELLE REGIONI IN pNLmodΔV3GFP 63
7.5 SEQUENZIAMENTO DELLE CHIMERE VIRALI
65
7.6 COLTURE CELLULARI
65
7.7 TRASFEZIONI
66
7.8 PRIMERS OLIGONUCLEOTIDICI
69
8 RISULTATI…………………………………………………………71
8.1 CARATTERISTICHE DEI PAZIENTI
71
8.2 VALIDAZIONE DEL SAGGIO UTILIZZATO PER IL CLONAGGIO DI
SEQUENZE V3 IN pNLmodΔV3GFP
72
8.3 CONFRONTO TRA SAGGIO FENOTIPICO HOME BREW ED ESTA
73
8.4 VALUTAZIONE DEL TEST GENOTIPICO GENO2PHENO
74
8.5 CAPACITA’ REPLICATIVA DELLE VARIANTI VIRALI CLONATE
77
8.6 ANALISI FILOGENETICA DELLE SEQUENZE VIRALI
79
9 DISCUSSIONE E CONCLUSIONI………………………….80
10 BIBLIOGRAFIA……………………………………………...90
2
1 INTRODUZIONE
1.1 HIV: Identificazione
L’HIV (Human Immunodeficiency Virus) (1, 2) è un retrovirus T-linfotropico
citopatico appartenente al genere dei Lentivirus. E’ stato isolato per la prima
volta nel 1983 dai linfociti del sangue periferico di un paziente affetto da
linfoadenomegalia,
ed
è
il
virus
responsabile
della
sindrome
da
immunodeficienza acquisita (AIDS), malattia riconosciuta come entità clinica
già nel 1981 (3).
Attualmente risultano infettati milioni di soggetti in tutto il mondo; una volta
acquisita l’infezione, l’individuo rimane siero positivo per tutta la vita e se non
sottoposti ad adeguati trattamenti, quasi tutti i soggetti nel giro di un decennio
vanno incontro ad infezioni opportunistiche letali, a causa delle carenze
indotte dal virus a carico del sistema immunitario. L’AIDS rappresenta,
all’inizio del 3° millennio, uno dei più importanti problemi di salute pubblica
in tutto il pianeta.
L’epidemia globale da HIV è il risultato di un infezione cross-specie avvenuta
in alcuni individui della popolazione dell’Africa centrale con un lentivirus
specifico per gli scimpanzé (SIV cpz). Il virus SIVcpz da un infezione
asintomatica negli scimpanzé e non causa alcuna patologia nell’ospite
naturale. La trasmissione sperimentale di questi virus in ospiti non naturali
3
suscettibili risulta, comunque, in una progressiva e profonda immudeficienza e
nella sindrome da immunodeficienza acquisita (AIDS).
Ad oggi, si conoscono due sierotipi di HIV: HIV-1, che si riferisce a virus
geneticamente correlati, diffusi ormai in tutto il mondo e l’HIV-2 che è un
virus distinto, meno trasmissibile e geograficamente limitato all’Africa
occidentale (4). Tutti gli individui infettati con HIV-2 mostrano un lungo
periodo di latenza clinica e una più bassa mortalità.
1.2
HIV: Classificazione
HIV-1 appartiene alla famiglia delle RETROVIRIDAE, virus a RNA che fino
agli anni ‘50 erano conosciuti e studiati perché in grado di indurre patologie a
carattere neoplastico in alcune specie animali. Tutti i retrovirus noti fino ad
oggi, sono simili per struttura, per organizzazione genomica e per la modalità
di replicazione che è influenzata in maniera univoca dalla presenza della
trascrittasi inversa. Sono stati proposti diversi tipi di classificazione tra cui la
più
recente
distingue
i
retrovirus
in
due
sottofamiglie:
ORTHORETROVIRINAE e SPUMAVIRINAE. La prima sottofamiglia
comprende sei generi: Alpharetrovirus, Betaretrovirus, Gammaretrovirus,
Deltaretrovirus, Epsilonretrovirus e Lentivirus; alla seconda appartiene il solo
genere degli Spumavirus.
4
Gli Spumavirus non sono stati associati in maniera definitiva ad alcuna
particolare patologia e sono definiti virus schiumosi per la capacità di indurre
lesioni vacuolari che conferiscono un aspetto schiumoso alle cellule infette
coltivate in vitro.
I Lentivirus comprendono retrovirus responsabili di una varietà di malattie
neurologiche e immunologiche, ma non sono direttamente implicati nello
sviluppo di neoplasie. Spesso sono in grado di infettare le cellule linfoidi
preposte alla risposta immune, con una particolare propensione a replicare nei
macrofagi. Inducono malattie croniche degenerative nei loro ospiti, precedute
da un lungo periodo d’incubazione (da cui il nome “virus lenti”) e da un
coinvolgimento variabile del sistema immunitario e del sistema nervoso
centrale. Un’altra caratteristica che li contraddistingue è la complessità del
genoma. Ci sono due tipi distinti di virus umani dell’AIDS, HIV-1 e HIV-2,
distinguibili sulla base della loro organizzazione genetica e delle relazioni
filogenetiche con altri lentivirus dei primati (5, 6).
Dati recenti indicano che HIV-1 comprende tre distinti gruppi di virus: il
gruppo M (Main o Major), il gruppo N (New) e il gruppo O (Outlier). I
gruppi N e O rimangono largamente confinati ad una parte dell’Africa centrooccidentale, sebbene ci siano sporadiche infezioni attraverso contatti con
persone di quelle regioni. Il gruppo M è responsabile della grande
maggioranza delle infezioni nel mondo e consiste di 11 sottotipi (clades)
designati con le lettere da A a K. All’interno di un dato sottotipo, gli anticorpi
specifici per gli isolati virali provenienti da un dato paziente non riconoscono
5
gli isolati di altri pazienti. Sequenze di aminoacidi della glicoproteina virale
dell’envelope mostrano fino a 25-35% di divergenza tra differenti sottotipi e
fino al 20% di divergenza all’interno di un dato sottotipo.
Nel corso delle epidemie, a causa di frequenti superinfezioni, ceppi di HIV-1
possono ricombinare e formare virus mosaico, alcuni dei quali, chiamati
forme ricombinanti circolanti (CRF), possono diventare predominanti dal
punto di vista epidemiologico a causa della loro maggiore capacità replicativa
e/o per una maggiore capacità di adattamento all’ospite.
Similmente ad HIV-1, i ceppi HIV-2 comprendono 6 distinte linee
filogenetiche, denominate con lettere da A ad F. HIV-1 e HIV-2 differiscono
fra loro per circa il 55-60% della sequenza genetica. Nonostante l’elevata
divergenza nucleotidica, l’organizzazione del genoma è quasi identica e
differisce soltanto in un gene ausiliare: HIV-1 contiene il gene vpu, mentre
HIV-2 contiene vpx. Entrambi i virus usano il recettore CD4 e i corecettori
CCR5 e CXCR4 per poter entrare nelle cellule. Confrontata con HIV-1,
l’infezione con HIV-2 è associata a un periodo di latenza più lunga, una più
lenta progressione della malattia, una bassa carica virale e una ridotta velocità
di trasmissione.
6
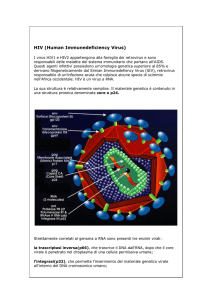
2 MORFOLOGIA E STRUTTURA DEL VIRIONE
2.1
Struttura del virione
Il virione ha un nucleocapside centrale troncoconico, quasi sferico con
diametro compreso tra i 100 e i 120 nm. Risulta costituito per il 60-70% da
proteine, per il 30-40% da lipidi derivati dalla cellula ospite, per il 2% da
carboidrati e infine per l’1% da RNA genomico.
Nella particella virale si può distinguere un pericapside esterno (envelope) e
una parte più interna, rappresentata dal nucleocapside (o core). Il pericapside è
costituito da un doppio strato fosfolipidico sul quale si ancorano proteine
virus-specifiche, come la proteina di superficie con funzione di antirecettore
gp120 (SU) e la proteina transmembrana con attività fusogena gp41 (TM). La
superficie dell’involucro presenta circa 72 protuberanze (7, 8) a simmetria
triangolare (peplomeri o spike); ogni peplomero è costituito da 4 eterodimeri
contenenti ciascuno una molecola SU che risulta ancorata con la porzione
COOH- all’estremo NH2 terminale della proteina TM.
Nell’envelope vengono inserite alcune proteine cellulari, come gli antigeni di
istocompatibilità (MHC), acquisite dai virioni nel processo di gemmazione
dalla membrana nucleare.
Associata alla faccia interna del doppio strato fosfolipidico si osserva una
struttura proteica di matrice costituita dalla proteina p17(MA). Studi
approfonditi mostrano una disposizione regolare della proteina p17 a formare
7
un guscio di 7 nm, che rappresenta un ispessimento interno del doppio strato
fosfolipidico. In ciascuna molecola p17 viene attuata la miristilazione, una
modifica post-traduzionale che consiste nel legame di molecole dell’acido
miristico a residui di glicina posti nella porzione N-terminale di tali proteine
(9). La miristilazione sembra assumere un ruolo fondamentale nel legame
della p17 alla faccia interna dell’involucro e nel garantire un regolare
montaggio delle glicoproteine dell’envelope durante le fasi tardive della
replicazione. All’interno della particella virale si trova il nucleocapside o
“core” centrale con una struttura a tronco di cono, la cui base maggiore ha un
diametro di circa 60 nm, mentre quella minore, legata alla proteina di matrice,
ha un diametro di 20 nm. La regione tra il nucleocapside e l’envelope è detta
regione paranucleoide (10), la cui composizione deve essere ancora
determinata.
Il nucleocapside è costituito da due molecole di RNA virale a singolo
filamento incapsulato da proteine che derivano dal polipeptide precursore
sintetizzato dal gene gag. Tale precursore è tagliato dalla proteasi virale in
quattro prodotti proteici: la p24, principale proteina capsidica, la cui funzione
è di impacchettare il genoma virale nei virioni; la p17 o proteina di matrice,
che si localizza tra il nucleocapside e l’envelope virionico, la p9 proteina del
nucleocapside che si lega strettamente al genoma e infine la p7 la cui
localizzazione non è ancora chiara (11). Tali proteine sono assemblate
secondo una simmetria cubica di tipo icosaedrico.
8
In ogni particella virale matura, il core contiene i componenti necessari alla
replicazione del virus: due copie identiche di RNA a singola catena a polarità
positiva, molecole di RNA transfer (tRNA), che vengono utilizzate con la
funzione di innesco (primer) per la trascrittasi inversa, e gli enzimi trascrittasi
inversa (RT), integrasi (IN), proteasi (PR), le cui funzioni si svolgono
rispettivamente nella replicazione del genoma virale, nell’integrazione nel
DNA della cellula ospite e nella maturazione dei virioni.
2.2 Struttura e organizzazione del genoma virale
Come tutti i retrovirus, HIV-1 è dotato di un genoma diploide, presente in due
forme: due filamenti identici di RNA mantenuti insieme mediante legami
idrogeno tra sequenze complementari poste vicino all’estremità 5’ di ciascuna
molecola e un DNA a doppia elica all’interno della cellula. Negli stadi iniziali
dell’infezione, l’RNA virionico è convertito nella forma di DNA a doppio
filamento mediante la RT del virus ed è, in seguito, integrato nel genoma della
cellula ospite.
Le due copie identiche di RNA a singola catena a polarità positiva sono di
circa 9,8 Kb per un peso molecolare di 30 Kd. Le due molecole sono
poliadenilate alle estremità 3’, una modificazione post-trascrizionale, tipica
della maggior parte degli mRNA eucariotici che avviene ad opera dei sistemi
presenti nella cellula ospite. Occasionalmente i residui di adenina possono
9
essere metilati. Alle estremità 5’, invece, durante la sintesi viene applicato un
gruppo cap (m7G5’ppp5’Gmp) dal sistema di trascrizione cellulare. Vicino
all’estremità 5’ di ogni filamento è posizionata una molecola di tRNAlys che
serve da primer per la sintesi del filamento negativo di DNA ad opera della
RT.
Il genoma provirale di HIV-1 (Figura 1) presenta due sequenze ripetute dirette
alle estremità, denominate long terminal repeat (LTR), che fiancheggiano le
tre principali open reading frame (ORF) del virus che sono gag, pol, env, e
altre sei piccole ORF, tat e rev essenziali per la replicazione virale, vif, vpr,
vpu, e nef, che sono anche detti geni accessori o ausiliari. La presenza di
queste diverse ORF conferisce al virus uno straordinario livello di
complessità.
Figura 1: Organizzazione del genoma provirale di HIV-1.
10
2.2.1 Long Terminal Repeat
Tutte le molecole di RNA presentano alle due estremità sequenze identiche
denominate R (Repeat) che hanno un ruolo importante durante la
retrotrascrizione. A fianco di ciascun segmento R, nella parte interna, si
trovano
sequenze
caratteristiche
U
denominate
U5
e
U3,
situate
rispettivamente alle estremità 5’ e 3’.
Durante la sintesi del DNA provirale, le sequenze U5 e U3 sono retrotrascritte
ad ambedue gli estremi del DNA nascente, dando origine a molecole più
lunghe rispetto all’RNA genomico. Le sequenze identiche U3-R-U5 presenti
ad ogni estremo del DNA provirale formano le cosiddette LTR (Long
Terminal Repeat), che rappresentano sequenze non codificanti e altamente
conservate, contenenti informazioni essenziali per l’inserzione del DNA virale
in quello della cellula ospite e per la trascrizione del genoma. La regione U3 si
estende dall’estremità 5’ del genoma fino al sito di inizio della trascrizione
(+1) e contiene il promotore virale, costituito da tre domini trascrizionali: il
dominio di modulazione, l’enhancer e il dominio di base o core. La prima
porzione della U3 è la regione di modulazione che contiene elementi cisacting regolatori della trascrizione ai quali si legano molti fattori cellulari
(come AP-1 e N-FAT-1). A valle della regione di modulazione si trova
l’enhancer, una regione che in HIV-1 si estende per due siti di 10 bp. Il fattore
cellulare NF-kB si lega a questi due siti aumentando l’attività trascrizionale
del virus in seguito alla sua stessa attivazione da parte di alcune proteine
11
regolatorie cellulari o di altri virus. All’estremità 3’ della regione U3 si trova il
core che contiene due brevi segnali, chiamati CAT box e TATA box. La
sequenza TATA box rappresenta il promotore per la trascrizione, mentre le
CAT box sono sequenze coinvolte nella regolazione della trascrizione.
Immediatamente dopo la TATA box sono posizionati siti per il legame del
fattore trascrizionale cellulare SP1 (12).
La regione R codifica per una sequenza di RNA che genera una struttura a
forcina (stem-loop) all’estremità 5’ del trascritto denominata tat-responsive
region (TAR). A questa struttura si lega la proteina tat di HIV-1, potente
attivatore della trascrizione virale. La regione R/U5 della 3’ LTR si trova nella
sequenza leader di tutti i trascritti virali e definisce l’estremità 3’. Tra le
sequenze adiacenti alle LTR all’estremità 5’ si trova la sequenza PBS (Primer
Binding Site), che è perfettamente complementare ai nucleotidi terminali
dell’estremità 3’del tRNAlys che funziona da primer.
Sebbene le sequenze nucleotidiche delle due LTR siano identiche, i retrovirus
hanno meccanismi che consentono loro di utilizzare la 5’ LTR come
promotore della trascrizione e la 3’ LTR come segnale di aggiunta della coda
di poli-A. Infatti, segnali nella regione U3 e R sono riconosciuti da fattori
cellulari che aggiungono code di poli-A all’estremità 3’ dei trascritti virali
(13).
12
2.2.2 Geni strutturali
Come tutti i retrovirus, il genoma di HIV-1 possiede tre geni strutturali
essenziali per la replicazione, denominati gag (group-specific antigen), pol
(polymerase) ed env (envelope) e organizzati nell’ordine 5’-gag-pol-env-3’. I
geni gag-pol sono tradotti inizialmente in un’unica poliproteina p160, che
viene successivamente scissa nei precursori delle proteine codificate da gag e
negli enzimi codificati da pol.
Il gene gag rappresenta la prima ORF del genoma di HIV-1 che codifica per
un peptide di 55 Kd tradotto da RNA messaggeri non sottoposti a processi di
splicing. Il precursore, polipeptide p55, a sua volta viene scisso attraverso
meccanismi di proteolisi in tre proteine strutturali: la proteina p24 che
rappresenta il principale costituente del capside, la proteina di matrice p17 e la
p15 da cui derivano p7 e p6 che possono stabilire legami con l’RNA.
Il gene pol si sovrappone per circa 241 bp al gene gag e viene espresso come
un proteina di fusione gag/pol da un trascritto unspliced lungo quanto il
genoma. Dalla segmentazione della proteina si originano alcuni enzimi virali:
la proteasi p10, con un taglio autocatalitico, l’integrasi p32 e la trascrittasi
inversa, un eterodimero composto di due polipeptidi, p51 e p66. Un dominio
indipendente di quest’ultima svolge la funzione di ribonucleasi.
Il gene env codifica per un precursore altamente glicosilato, la gp160, tradotta
da un mRNA policistronico derivato da un singolo processo di splicing
(singly-spliced). Questa proteina integrale di membrana è formata da 850
13
aminoacidi e viene scissa da un’endopeptidasi dell’ospite in due glicoproteine
a più basso peso molecolare: la N-terminale gp120, che si posiziona
sull’envelope virale, e la C-terminale gp41, proteina transmembranaria.
Queste due proteine costituiscono, attraverso interazioni non covalenti,
l’antigene di superficie di HIV-1, che media l’ingresso del virus. Studi relativi
alla comparazione di sequenze rivelano un modello secondo cui la regione di
env che codifica per la gp 120 presenta 5 regioni variabili (V1-V5) intersperse
tra regioni conservate (C1-C5), mentre la regione codificante per la gp41 è ben
conservata (14). Uno dei 5 domini variabili, la regione V3, risulta in realtà
ipervariabile ed è il principale bersaglio dell’attività degli anticorpi
neutralizzanti.
2.2.3 Geni regolatori
I geni regolatori principali tat e rev controllano l’espressione dei geni virali a
livello trascrizionale e post-trascrizionale e sono il risultato della traduzione di
mRNA originati da doppi eventi di splicing.
Il primo esone codificante di tat è localizzato nella regione centrale del
genoma virale tra il gene vpr e il gene env, mentre il secondo esone si
sovrappone alla cornice di lettura (open reading frame) di rev e della gp41 di
env (15). Il gene tat (Trans Activator of Transcription) codifica per una
proteina di 14Kd, costituita da 86 aminoacidi. Una volta sintetizzata, Tat entra
14
nel nucleo cellulare e agisce principalmente come un potente attivatore della
trascrizione del genoma provirale aumentando l’efficienza dell’Rna polimerasi
II cellulare (16). Tat si lega ad una specifica sequenza TAR (Tat Responsive
Region) presente all’estremità 5’ di ogni messaggero e interagisce con un
complesso poliproteico che comprende l’RNA polimerasi II, la ciclina T e la
Cdk9.
Il gene rev (Regulator of Virion Expression) codifica per la proteina rev di
19Kd, costituita da 116 aminoacidi e coinvolta nel trasporto nucleocitoplasmatico degli RNA virali che subiscono un unico evento di splicing e di
quelli che non subiscono splicing. Agisce in trans legandosi ad regione ben
definita dell’RNA trascritto detta RRE (Rev Responsive Element) e rimane
localizzata prevalentemente nel nucleo della cellula infetta (17). Una volta
prodotta, la proteina rientra nel nucleo per esercitare la sua funzione di
trasporto degli RNA. RRE è una struttura costituita da 351 nucleotidi ed è
localizzata in corrispondenza del gene env, verso l’estremità 3’ di tutti gli
mRNA. Rev forma multimeri ed è stato messo in evidenza che la
multimerizzazione è necessaria per espletare la sua funzione.
2.2.4 Geni accessori
I geni accessori di HIV-1 sono vif, vpr, vpu e nef. Le proteine di questi geni
sono tradotte per lo più da trascritti singly-spliced.
15
Vpr, viral protein R (p15), è una proteina regolatoria di 15KD che si trova
nelle particelle virali mature e sembra intervenire in maniera predominante
nella replicazione virale e nella patogenesi durante i primi stadi dell’infezione.
Uno dei ruoli principali di Vpr è di trasportare nel nucleo, dopo la
retrotrascrizione, il DNA virale associato con le proteine virali (il cosiddetto
PIC, complesso di preintegrazione). Inoltre, promuove l’arresto del ciclo
cellulare in G2 forse attraverso una inibizione indiretta dell’attività fosfatasica
di Cdc25 e favorisce l’infezione dei macrofagi e monociti, cellule in cui la
replicazione di HIV-1 dipende dalla possibilità di poter importare il PIC virale
nel nucleo. Oltre all’importo nucleare, agisce in modo da permettere il suo
rientro nel citoplasma per essere incorporato nei nuovi virioni e permettere la
distribuzione del virus maturo.
Vif, viral infectivity factor (p23), è una proteina basica di 23 KD espressa
tardivamente durante il ciclo replicativo e largamente localizzata nel
citoplasma. Diversi studi hanno evidenziato che linfociti CD4+ e macrofagi,
che sono le principali cellule nelle infezioni da HIV-1, in mancanza di Vif
diventano non permissive alla replicazione del virus e che la sua presenza è
indispensabile anche per il mantenimento dell’infettività virale. Inoltre,
contrasta l’effetto di APOBEC3G (citidina deaminasi cellulare) che produce
ipermutazione del genoma provirale e infezione abortiva.
Vpu ,viral protein U, è una proteina di 16 KD che forma canali ionici nel
doppio strato lipidico e permette l’aumento della permeabilità della membrana
plasmatica nei confronti di numerose molecole. Nel ciclo replicativo virale
16
svolge due funzioni, quella di indurre la degradazione dei recettori CD4 e il
conseguente aumento del rilascio dei virioni dalle cellule infettate. Vpu è
assente in HIV-2, dove invece è presente la proteina Vpx, la cui funzione è
ancora poco chiara, sebbene sembri svolgere un ruolo nell’importo nucleare.
Il gene nef (Negative Expression Regulatory Factor) si estende dall’estremità
3’ di env fino all’interno del dominio U3 della 3’ LTR e codifica per una
fosfoproteina
miristilata
di
205
aminoacidi,
questa
modificazione
postraslazionale gli permette di associarsi alla membrana plasmatica della
cellula infettata. La proteina Nef è una proteina multifunzionale non essenziale
per la replicazione in alcune culture cellulari, ma svolge un ruolo cruciale
nella replicazione virale in vivo e nella patogenesi. Infatti, virus Nef-difettivi
non inducono alti livelli di viremia negli animali infettati e la progressione
della malattia è più lenta. Nef agisce riducendo la presenza di CD4 sulla
superficie cellulare mediante endocitosi (18) e down-regolando l’espressione
di molecole MHC-I per proteggere la cellula infettata dal riconoscimento da
parte dei linfociti T citotossici. Inoltre, blocca l’apoptosi, eleva l’infettività e
altera lo stato di attivazione cellulare.
2.3 Env
Il gene env codifica per un precursore proteico altamente glicosilato, la gp
160. Questa glicoproteina, che risulta composta da 850 aminoacidi, matura nel
citoplasma all’interno del reticolo endoplasmatico ad opera di enzimi cellulari.
17
Un’endopeptidasi scinde il precursore proteico in due glicoproteine a più
basso peso molecolare: la proteina extracellulare gp120 (detta subunità SU) e
la proteina di trasmembrana gp41 (detta subunità TM), identificate sulla base
della mobilità elettroforetica su gel di poliacrilamide in condizioni denaturanti.
Nella forma matura la glicoproteina di env è un eterodimero costituito dalle
subunità gp120 e gp41 mantenute insieme da legami idrogeno. Le
glicoproteine del gene env si legano al recettore CD4 presente sulla membrana
plasmatica dei linfociti T CD4+, monociti, macrofagi e cellule dendritiche e
sono quindi responsabili delle prime fasi del ciclo replicativo di HIV-1,
permettendo l’attacco dei virioni alle cellule e facilitandone l’entrata. In
particolare, la subunità SU ha un ruolo prioritario nel riconoscimento
dell’antigene cellulare CD4 e dei recettori per le chemiochine (CCR-1, CCR2b, CCR-3, CCR-5, CXCR-4) presenti sulla superficie delle cellule bersaglio,
tramite domini che li riconoscono e li legano (19). La subunità TM consente in
seguito l’ingresso del virus nella cellula ospite mediante la fusione della
membrana cellulare con quella virale. Inoltre, tali proteine sono il bersaglio
principale della risposta immunitaria antivirale dell’ospite infetto in vivo e
sono anche responsabili della formazione di sincizi (cellule multinucleate)
nelle culture cellulari in vitro.
Il precursore poliproteico per la glicoproteina dell’envelope di HIV-1, deriva
da un trascritto virale, codificante anche per la proteina vpu, che subisce un
solo processo di splicing. Tale trascritto è codificato solo nelle fasi finali del
ciclo replicativo del virus e la sua espressione è strettamente dipendente dalla
18
funzione post-trascrizionale del gene virale rev. Durante la sua sintesi tale
precursore viene estensivamente glicosilato dando origine alla gp160, che è la
forma più abbondante del prodotto del gene env ritrovata nelle cellule infette.
Un taglio intracellulare della gp160 dà origine alla subunità N-terminale, la
gp120 di circa 550 aminoacidi, e alla subunità C-terminale, la gp41 lunga circa
350 aminoacidi. La subunità SU è una proteina idrofila altamente glicosilata
posizionata sia sulla superficie esterna della membrana virale, sia sulla
membrana citoplasmatica della cellula infettata. La subunità TM è, invece,
classificata come proteina integrale di membrana di tipo I, data la sua parziale
idrofobicità che le consente di attraversare il doppio strato lipidico delle
membrane virali e cellulari. Il legame tra le due subunità SU e TM è di tipo
non covalente e coinvolge residui aminoacidici al terminale amminico e a
quello carbossilico della gp120 che sono fondamentali per mantenere
l’associazione con la gp41. È stato ipotizzato che la formazione di un loop
disolfuro nella gp41, costituisca una protuberanza che si adatti perfettamente
all’interno di una tasca nella subunità della gp120 ripiegata. In ogni caso
l’assenza di legami covalenti non preclude la possibilità di una interazione
stabile tra gli eterodimeri della glicoproteina dell’envelope che risultano
organizzati in complessi oligomerici.
19
2.3.1 La gp120 o dominio SU
La gp120 presenta una estesa glicosilazione che, sulla base di algoritmi al
computer, lascia prevedere come pochissime regioni dello scheletro peptidico
possano sporgere dalla massa di carboidrati (20). I carboidrati possono, in
alcuni casi, alterare il riconoscimento della gp120 da parte del sistema
immunitario, nascondendo epitopi peptidici e fornendo così al virus un
meccanismo per sfuggire alla risposta immunitaria antivirale. Sono stati
individuati 24 siti di glicosilazione nel dominio SU in HIV-1, di cui 13
risultano conservati in differenti isolati virali. Il processamento intracellulare e
le proprietà funzionali ed immunologiche della glicoproteina matura sono
fortemente influenzate dalla glicosilazione del precursore gp160. Tramite
tecniche di mutagenesi sito-diretta nel gene env, è stato possibile dimostrare
che 5 dei 24 potenziali siti di glicosilazione sono essenziali per la replicazione
virale (21). In particolare, questi siti critici (Asn88, Asn141, Asn197, Asn262,
Asn276), che si trovano a metà del terminale amminico della gp120, se mutati,
rendono le particelle virali non infettive pur mantenendo la capacità di legarsi
al recettore CD4. Nella sequenza peptidica della gp120 sono presenti 18
residui di cisteina altamente conservati nelle glicoproteine di differenti ceppi
di HIV-1 e HIV-2. Ciò fa supporre che legami disolfuro giochino un ruolo
centrale nella struttura e nella funzione di questa proteina virale. Un modello
per la subunità gp120, basato su analisi biochimiche, mostra nove legami
disolfuro intramolecolari delineando, così, alcune regioni funzionali che
20
includono diversi domini conformazione-dipendente (Figura 2). Tali domini
sono responsabili del riconoscimento del recettore CD4 e dei corecettori
specifici presenti sulla superficie della cellula bersaglio. In particolare, sembra
che un limitato gruppo di aminoacidi conservati in differenti regioni della
gp120 sia richiesto per un efficiente legame al CD4, mentre una regione
conservata alla base del loop V1/V2 interagisce con il corecettore specifico
(22). Anche l’interazione di regioni non contigue sono importanti per la
produzione di una conformazione funzionale della gp120. Concludendo, sia la
sequenza primaria (aminoacidi conservati in regioni non contigue) che le
caratteristiche conformazionali (epitopi discontinui) del dominio SU,
producono una configurazione che riconosce il recettore CD4 ed i corecettori
specifici in modo selettivo e con alta affinità.
Figura 2: Modello della struttura ripiegata della gp120.
21
2.3.2 La gp41 o dominio TM
La sequenza peptidica della gp41 presenta 4 siti di glicosilazione e 3 residui di
cisteina. Anche in questo caso mutazioni a 2 dei 4 possibili siti di
glicosilazione, riducono l’infettività di HIV-1 e la formazione di sincizi in
colture cellulari in vitro dimostrando che la glicosilazione di tali siti è
importante per le diverse fasi della replicazione virale. Circa venti
amminoacidi al terminale amminico della gp41 (fusion peptide), sono
idrofobici e consentono al peptide di svolgere la funzione di fusione della
membrana virionica con la membrana citoplasmatica della cellula (23). Un
secondo dominio idrofobico (amminoacidi dal 684 al 705) attraversa le
membrane virioniche e cellulari, consentendo alla gp41 di fungere da ancora
per l’eterodimero della glicoproteina dell’envelope. Tra questi due domini
idrofobici si trova una regione esterna alla membrana (ectodominio) altamente
conservata; si è visto che tale sequenza è molto simile al motivo cerniera di
leucine (leucine-zipper) implicato nelle interazioni interproteiche di un gran
numero di proteine virali e cellulari. Durante l’ingresso del virus nella cellula,
una porzione dell’ectodominio assume una conformazione a spirale che
facilita l’inserzione della gp41 nella membrana della cellula bersaglio,
consentendo la fusione. Mutazioni a carico del motivo leucine-zipper
dell’ectodominio bloccano l’infettività virale e la fusione cellulare.
Inoltre, la gp41, presenta nel dominio citoplasmatico, due regioni anfipatiche
ad - elica, una tra la tirosina 768 e l’arginina 788 e l’altra tra l’arginina 826 e
22
la leucina 854 al terminale carbossilico. Queste regioni sono assimilabili a
quelle che codificano per i peptidi litici dei lentivirus (LLP-1 e LLP-2) perché
inducono la formazione di pori nella membrana citoplasmatica provocando la
lisi della cellula.
2.3.3 Variabilità del gene env e del dominio V3
Una caratteristica di HIV è l’estrema variabilità genetica. Isolati indipendenti
di HIV-1 e HIV-2, presentano un’ampia eterogeneità di sequenza a livello del
gene env; la subunità SU presenta ben 5 regioni altamente variabili (da V1 a
V5) interspaziate da regioni conservate (C1-C5), mentre la subunità TM è
molto più conservata (24). Queste variazioni di sequenza consistono in
sostituzioni nucleotidiche che determinano variazioni amminoacidiche, come
pure in delezioni o inserzioni di grandezza variabile. (25). Più del 25% degli
amminoacidi codificati dal gene env risulta variabile in ceppi di HIV isolati da
distretti geograficamente separati. Come descritto precedentemente, l’esame
epidemiologico molecolare, basato sulla sequenza del gene env di HIV-1,
rivela distinti sottotipi o clade (14). La variabilità genetica di HIV-1 dipende
da mutazioni e da processi di ricombinazione ed è correlata al tasso di
replicazione virale. La frequenza mutazionale è di circa 3x10 -5 per nucleotide
per ciclo replicativo ed è dovuta alla mancanza di attività di “proof-reading”
esonucleasica in direzione 3’-5’della trascrittasi inversa di HIV-1 (26). Questa
23
elevata velocità di mutazione conferisce al virus la capacità di rispondere alla
pressione selettiva a cui è sottoposto all’interno dell’ospite. La maggior parte
delle mutazioni conferisce al virus uno svantaggio biologico, tuttavia molte
varianti vengono selezionate dalla pressione immunitaria, da terapie o da altre
forze selettive; di conseguenza tali varianti conferiscono al virus la capacità di
sfuggire alla risposta immunitaria umorale e cellulo-mediata dell’ospite e sono
indicate come escape mutants. L’attività degli anticorpi neutralizzanti è diretta
contro la glicoproteina dell’envelope, quindi varianti virali con mutazioni a
livello del gene env possono risultare resistenti alla neutralizzazione e si
espandono nell’ospite soppiantando la popolazione virale originaria. A sua
volta, il sistema immunitario evolverà una risposta tesa ad eliminare i nuovi
virus esercitando, in tal modo, una pressione selettiva verso l’affermazione di
altre varianti antigeniche virali. Queste variazioni intra-ospite, portano alla
produzione di una popolazione di genomi virali strettamente correlati ma
distinti, denominati quasispecie.
È ormai chiaro che le mutazioni a carico dei geni codificanti per le proteine
dell’involucro influenzano profondamente la cinetica di replicazione del virus,
la capacità sinciziogena e soprattutto il tropismo virale nonché la
neutralizzazione da parte degli anticorpi. Nell’ambito della gp120 le regioni
ipervariabili V1, V2 e V3 sono determinanti del tropismo virale e del fenotipo
SI (syncitia-inducing) per i virus linfocitotropici o NSI (non syncitia-inducing)
per i virus macrofagotropici. Tali regioni pur non essendo connesse con il
riconoscimento del CD4, sono implicate nel riconoscimento del corecettore
24
presente sulle cellule bersaglio e possiedono epitopi per anticorpi
neutralizzanti che agiscono sia prima che dopo l’assorbimento del virus alla
cellula; da ciò consegue che una maggiore efficienza ad infettare tipi e
sottotipi cellulari, determinata dall’adattamento all’ospite, potrebbe guidare la
selezione di mutanti a livello di queste regioni. Quindi si può affermare che la
variabilità genetica di HIV-1 consente al virus sia la fuga da anticorpi
neutralizzanti che la capacità di infettare tipi cellulari diversi.
La terza regione ipervariabile della gp120 detta V3, è quella che presenta la
più significativa eterogeneità di sequenza ed è il principale bersaglio degli
anticorpi neutralizzanti nonché un importante determinante di patogenesi, in
quanto la struttura terziaria del V3 loop sembra influenzare il coreceptor
usage del virus (27,28); da essa dipende la capacità di formare sincizi e il tasso
di replicazione (29). Il dominio V3 è formato da 35 amminoacidi arrangiati in
un loop disolfuro che coinvolge la Cys301 e la Cys336 (30, 31). Il tripeptide
Gly317-Pro318-Gly319, localizzato all’apice del loop, è altamente conservato
mentre alla base del loop le sequenze vicino alla Cys301 e alla Cys336
mostrano una bassa variabilità. I principali cambiamenti si verificano in 10 dei
12 aminoacidi in ciascun lato dell’apice conservato del loop. Mutazioni che
alterano singoli residui del tripeptide Gly317-Pro318-Gly319, rendono i virus
non infettanti sebbene la gp160 di questi mutanti mostra normali modelli di
sintesi e di processamento mantenendo la capacità di legare il recettore CD4
(32, 33). Alcuni di questi mutanti perdono anche la capacità sinciziogena.
Studi genetici hanno dimostrato che gli amminoacidi conservati nel loop V3
25
influenzano alcune proprietà di HIV-1 che sono controllate dal gene env. Ad
esempio, la mutazione della Cys336 preclude il taglio della gp160 ed il legame
al recettore CD4, presumibilmente, alterando la struttura terziaria della
glicoproteina dell’envelope e modificando la conformazione del sito di legame
per il recettore CD4. E’ stato inoltre dimostrato che i due aminoacidi in
posizione 306 e 320 sono responsabili del differente fenotipo virale, inclusa la
capacita fusogena e il monocitropismo (34).
Mutazioni puntiformi della regione V3, hanno dimostrato che amminoacidi
basici in una o più delle posizioni 11, 13, 25, iniziando dalla Cys301,
conferiscono un fenotipo inducente sincizi, mentre amminoacidi idrofobici
neutri o carichi negativamente in queste posizioni sono correlati ad un
fenotipo non inducente sincizi. Sia la posizione del residuo amminocidico
carico, sia la carica totale influiscono sul fenotipo. Generalmente i loop V3
delle varianti SI hanno una carica totale, a pH neutro, maggiore o uguale a 5
mentre gli NSI hanno una minore carica positiva di circa 3 (35,36). I virus Ttropici hanno un aminoacido non acido o un’alanina in posizione 328
(posizione 32 del loop), mentre virus macrofagotropici presentano o un
aminoacido acido o un’alanina in questa posizione (34). È stato proposto che il
dominio V3 di differenti isolati di HIV-1 può assumere due diverse
conformazioni che determinano un tropismo preferenziale per i linfociti o per i
macrofagi (37). Comunque, l’analisi di provirus chimerici ha rilevato che il
dominio V3 è coinvolto, ma non è sufficiente per determinare un particolare
fenotipo.
26
Anche le sequenze della gp120 esterne al loop V3 possono influenzare i
fenotipi assegnati a questo dominio. L’analisi di virus chimerici, costruiti da
cloni di HIV-1 con distinti fenotipi, indica che l’interazione funzionale tra il
V3 e la seconda regione conservata è importante per l’infettività come pure
per la formazione di sincizi e per il tropismo cellulare. È stata individuata
anche un’interazione intramolecolare tra il loop V3 e gli amminoacidi nella
quarta regione conservata della gp120 (38).
In ultimo va ricordato che, subito dopo l’infezione, la popolazione virale
mostra una eterogeneità di sequenza relativamente bassa e consiste per lo più
di virus macrofagotropici NSI; virus T-tropici compaiono tardivamente
nell’infezione, in individui che hanno progredito verso l’AIDS (39). Ciò fa
supporre che i ceppi macrofagotropici NSI siano più facilmente trasmessi tra
individui o che siano fortemente selezionati nel nuovo ospite, subito dopo
l’infezione, dalle cellule che per prime vengono infettate. È possibile che i
macrofagi o le cellule che processano l’antigene, presenti al di sotto delle
barriere costituite dalle mucose ed esprimenti il corecettore CCR5, selezionino
i virus permettendo a quelli capaci di penetrare in queste cellule di replicare
con una maggiore efficienza (40). Inoltre, durante l’infezione primaria, il
sistema immunitario, non compromesso, sopprime varianti SI che replicano
più velocemente delle varianti NSI che, per altro, possono essere meno
immunogeniche forse grazie al loro monocitrotopismo (34). Le varianti virali
SI, linfocitotropiche si presume derivino dalle prime quasispecie e sono
associate con una immunodeficienza fatale nell’ospite.
27
3 CICLO REPLICATIVO
Il ciclo replicativo di HIV-1 è complesso, con una durata e una produzione
virale che dipendono dal tipo di target cellulare e dall’attivazione cellulare e
può essere suddiviso in una fase precoce ed una fase tardiva. La fase precoce
inizia con il legame del virione al recettore cellulare CD4 e allo specifico
corecettore, la penetrazione nella cellula ospite ed il denudamento del
nucleocapside. La trascrittasi inversa del virus, associata al nucleocapside,
converte l’RNA in DNA che è mantenuto in un complesso nucleoproteico e
trasportato all’interno del nucleo dove la doppia elica di DNA si integra nel
genoma cellulare. La fase tardiva della replicazione inizia con la trascrizione e
il processamento dell’RNA virale derivato dalla forma provirale e termina con
il rilascio di nuovi virioni dalla cellula.
3.1 Adsorbimento e penetrazione
Il ciclo di replicazione inizia con il riconoscimento delle cellule target da parte
del virione maturo. L’attacco di HIV-1 alle cellule è mediato dall’iniziale
interazione tra il dominio extracellulare dell’antigene di superficie del virus
(gp120) e il recettore CD4 localizzato sulla membrana cellulare dei linfociti Thelper, delle cellule dendritiche e cellule della linea monocito-macrofagica,
incluse le cellule della microglia nel sistema nervoso (41). Il recettore CD4 è
un membro della superfamiglia delle immunoglobuline ed è costituito da
28
quattro domini extracellulari (D1-D4) che hanno una struttura simile a quelli
ritrovati nelle altre famiglie di immunoglobuline (42). L’alta affinità di legame
per il sito di legame del CD4 nella gp120 è stato localizzato in un piccolo
segmento del dominio extracellulare N-terminale, analogo al loop CDR-2 nel
dominio variabile della catena leggera delle immunoglobuline. Le interazioni
tra il recettore CD4 e l’antirecettore virale sono sufficienti per l’attacco del
virus alla cellula ma non per la penetrazione, che è possibile ottenere solo
dopo il legame con il corecettore. A tutt’oggi, fermo restante il tropismo
preferenziale per i linfociti T CD4+, è stata dimostrata una permissività
all’HIV-1, da parte di monociti, macrofagi, cellule dendritiche, linfociti B,
linfociti T CD8+, cellule gliali, cellule endoteliali, fibroblasti, cellule
dell’epitelio intestinale e precursori ematopoietici del midollo osseo. In alcune
di queste cellule non è mai stata dimostrata direttamente la presenza di CD4
sulla membrana. È possibile ipotizzare, quindi, che in queste cellule il
recettore CD4 sia espresso in bassissime quantità, tali da non renderlo
rilevabile, o che esistano recettori alternativi. Tra quelli proposti vi sono: il
galattocerebroside, recettore usato da HIV sulle cellule epiteliali intestinali e
particolarmente abbondante nelle cellule nervose; la molecola CD26, un
antigene dotato di attività enzimatica (dipeptidil-peptidasi) espresso in grandi
quantità sui linfociti T, monociti e piastrine e le molecole HLA-DR (43). A
conferma di ciò numerosi studi hanno chiarito che l’HIV utilizza, oltre al CD4
(recettore primario), anche un secondo recettore, definito corecettore,
appartenente alla superfamiglia di recettori accoppiati alla proteina G (44). I
29
ligandi naturali di questi corecettori, sono rappresentati dalle e chemiochine, una serie di citochine che, tra le svariate funzioni (45), sono
responsabili del reclutamento dei leucociti nella sede dell’infiammazione (43).
A conferma di quanto detto, numerosi studi hanno dimostrato come anticorpi
diretti contro tali corecettori o contro le chemiochine, ad opportuni dosaggi,
siano in grado di inibire in maniera significativa la replicazione di HIV-1 (46).
Fino ad oggi sono stati individuati numerosi recettori delle chemiochine che
possono essere utilizzati dall’ HIV-1 per l’ingresso, tra questi il CCR5 che
lega le -chemiochine RANTES, MIP-1, MIP-1 (47); il CXCR4
precedentemente
chiamato
LESTER
(Leukocyteche-Expressed
Seven
Transmenbrane-domain-Receptor) o fusina che riconosce la proteina SDF-1; il
CCR3 che è il corecettore per l’eotaxina e per il MCP-4, fattore chemiotattico
per le cellule dendritiche; il CCR2b legante MCP-1, MCP-3, MCP-4 (48).
I principali corecettori per HIV sono CCR5, specifico per i ceppi
monocitotropici, e CXCR4, specifico per i ceppi linfocitotropici (49,50).
Diversi studi hanno dimostrato che le chemiochine MIP-1, MIP-1 e
RANTES possono bloccare l’infettività virale dei ceppi macrofago-tropici (Mtropici) in vitro, ma non quella dei ceppi T-linfocitotropici (T-tropici) (46,48).
I ceppi M-tropici sono responsabili della trasmissione virale e sono gli isolati
prevalenti negli individui asintomatici. Successivamente compaiono i ceppi
virali T-tropici correlati ad un’accelerata progressione della malattia.
Un modello basato sulla struttura cristallina della gp120 di HIV-1 TCLA
(virus adattati a linee cellulari T) complessata con due domini di CD4,
30
suggerisce che il sito di legame sulla gp120 per il CD4 è mascherato dal loop
V1/V2 e in parte dal loop V3. La gp120 può oscillare tra due conformazioni
transitorie: “aperta” in cui il sito di legame per il CD4 è esposto; “chiusa” in
cui alcuni loop variabili bloccano l’accesso a tale regione (51). È ragionevole
presumere che la gp120 di virus TCLA, è più frequentemente nello stato
“aperto” dove esplica una elevata affinità per il CD4, mentre negli isolati
primari la gp120 è più ostacolata al legame, mostrandosi nella configurazione
“chiusa”.
Sebbene il CD4 sia il principale responsabile dell’attacco del virus alle cellule
T, interagendo con la gp120, gli studi effettuati in proposito hanno delineato
un meccanismo ben più complesso e multi-step, utilizzato da HIV-1 per
entrare nelle cellule bersaglio. Tale meccanismo individua una prima
interazione tra la gp120 e il CD4 cellulare, in seguito alla quale la gp120
subisce un cambiamento conformazionale che permette l’esposizione di una
regione altamente conservata (bridging sheet), presente alla base della
struttura V1/V2 e vicino al loop V3. Il bridging sheet andrà così ad interagire
con il corecettore chemiochinico costituito dal CCR5 o dal CXCR4, presente
rispettivamente sulla superficie dei macrofagi o dei linfociti. Si pensa che sia
la carica che la conformazione del loop V3 determinino il diverso tipo di
recettore chemiochinico che interagisce con il bridging sheet (19,31,52).
Comunque, il legame di forme solubili di env con il corecettore è dipendente
dal CD4, suggerendo che proprio il legame con il CD4 è un pre-requisito
essenziale per la formazione del complesso trimolecolare di env, CD4 e
31
corecettore (48). A questo primo evento segue l’esposizione e l’attivazione
della gp41, che, una volta esposta, interagisce con la membrana cellulare
inducendo il processo di fusione del pericapside virale con il doppio strato
fosfolipidico della membrana citoplasmatica. È stato inoltre proposto che a
questo evento partecipi anche il CD26 (enzima cellulare che si può trovare
sulla membrana plasmatica o in endosomi), il cui ruolo sarebbe quello di
interagire con il loop V3, riconoscendo l’Arg320 come sito di taglio e
facilitando in questo modo l’assorbimento e la successiva penetrazione di
HIV-1 nella cellula (43). A seguito del taglio, infatti, si verificano
cambiamenti conformazionali tali per cui le sequenze leucine zipper della
gp41, presenti in ciascun complesso oligomerico e inizialmente separate tra
loro, interagiscono costituendo un dominio di fusione che facilita il processo
di fusione delle membrane. Quindi, si presume che la presenza di
un’appropriata proteasi possa essere un determinante per l’infettività virale in
alcuni tipi cellulari.
CCR5 e CXCR4, come gli altri recettori chemiochinici sono proteine di
transmembrana, contenenti 4 domini extracellulari: un dominio aminoterminale e 3 loops extracellulari (48). Attraverso studi con recettori chimerici
e mutazioni sito dirette, è stato dimostrato che l’interazione env-CCR5 è
confomazionalmente complessa e coinvolge residui in tutti e quattro i domini
extracellulari di CCR5. In particolare, il dominio N-terminale è estremamente
importante essendo in grado di conferire la funzione di corecettori ad una
varietà di altri recettori chemiochinici. Comunque, un numero di ceppi virali
32
M-tropici può tollerare significative alterazioni del dominio N-terminale di
CCR5, includenti anche una delezione di 16 residui, a testimonianza del fatto
che i loops extracellulari, particolarmente il secondo, possono supportare
l’infezione virale. Il corecettore CCR5 permette la fusione della membrana
virale di ceppi M-tropici di HIV-1 con la membrana cellulare, l’utilizzo di
questo recettore è associato al fenotipo NSI. La fusina o CXCR4, è implicata
specificatamente nell’infezione dei ceppi linfocitotropici ed è associata al
fenotipo SI; nel suo legame con env, è principalmente il secondo loop ad avere
un ruolo chiave (49).
Nei pazienti con infezione recente l’utilizzo del CCR5 è dominante, ma in
circa il 50% dei casi la variante CXCR4 compare negli ultimi stadi della
malattia caratterizzata dalla progressione verso l’AIDS. Le cause di questo
coreceptor switch non sono state ancora comprese completamente (53), ma
dall’analisi della struttura e delle funzioni dei corecettori, emerge una
possibile strategia con cui i ceppi T-tropici evolvono da virus M-tropici
acquisendo la capacità di utilizzare CXCR4 (54). Virus che utilizzano il CCR5
acquisiscono mutazioni nella proteina env, specialmente a carico del loop V3,
che consente loro di interagire con il secondo loop di CXCR4 pur mantenendo
la capacità di interagire produttivamente con il dominio N-terminale di CCR5.
Con il tempo questa capacità viene persa e il virus risulta in un ceppo
strettamente T-tropico. Generalmente il loop V3 dei T-tropici è più basico
rispetto allo stesso negli M-tropici, mentre il primo e il secondo loops
extracellulare di CXCR4 sono considerevolmente più acidi di quelli presenti in
33
CCR5. Così l’acquisizione di residui basici nel loop V3 può aumentare la
capacità di env ad interagire con i loops extracellulari acidi di CXCR4 (48).
Nei virus che utilizzano il CXCR4 o nei dual-tropici, V3 ha una notevole
carica positiva che le permette di interagire fortemente con molecole anioniche
come l’HSPG (heparan sulphate proteoglycans) e la porzione acida
extracellulare del CXCR4. Virus utilizzanti il CCR5 contano, invece, su altre
interazioni non elettrostatiche per un attacco efficiente. L’attacco dei ceppi R5
è molto meno influenzato da HPSG, rispetto ai ceppi X4; tale indipendenza è
correlata alla debole basicità del loop della gp120 che varia generalmente tra
+2,+5 (contro +7, +9 della V3 dei ceppi X4) (51).
Altre molecole possono giocare un ruolo primario nella regolazione dei
passaggi critici successivi al binding del virus e nella trasmissione da cellula a
cellula, come la fusione delle membrane dei linfociti T CD4+ e monociti
infettati da HIV o la conseguente formazione di sincizi. A tale riguardo, studi
recenti hanno dimostrato che la molecola CD4 è necessaria ma non sufficiente
a mediare la formazione di sincizi e che questo processo è regolato
dall’interazione tra la molecola di adesione LFA-1 e i suoi recettori ICAM-1,
ICAM-2 e ICAM-3 presenti sui linfociti (55).
Un fattore importante è il differente tropismo cellulare che distingue diversi
isolati di HIV-1 della medesima persona infetta. Isolati di HIV-1 del sistema
nervoso centrale crescono preferenzialmente su colture macrofagiche (ceppi
macrofagotropici) e sfruttano il CCR3 come corecettore per l’ingresso nella
cellula, mentre quelli ottenuti da cellule mononucleate di sangue periferico,
34
stimolate con PHA e IL2, si propagano meglio nei linfociti T (ceppi
linfocitotropici). Anche le cellule dendritiche del sangue e le cellule di
Langherans della pelle e delle mucose permettono la replica di molti ceppi di
HIV-1 con diverso tropismo. Al contrario, le cellule dendritiche follicolari dei
linfonodi, presentanti l’antigene ai linfociti T nei follicoli linfatici, possono
solamente legare l’HIV-1 senza essere infettate.
3.2 Sintesi ed integrazione del DNA virale
L’RNA genomico viene retrotrascritto nel citosol in una doppia catena di
DNA ad opera dell’enzima trascrittasi inversa (RT, p66/p51) che modula
diverse attività: DNA polimerasi-RNA dipendente, DNA polimerasi -DNA
dipendente e ribonucleasi (RNasi H) (56). Essa viene attivata nel citoplasma
da un segnale non ancora ben identificato. La sintesi del primo filamento di
DNA, cioè quello a polarità negativa, inizia con l’annealing del tRNAlys al sito
PBS (primer binding site) dell’RNA stampo che funge da primer per la RT. Il
prodotto della retrotrascrizione è una molecola bicatenaria ibrida costituita
dalla catena di RNA originale e dalla catena complementare di DNA. A questo
punto la seconda azione enzimatica della RT, detta RNAsi H, permette la
degradazione della molecola dell’RNA presente nell’ibrido. Segue la
trascrizione della seconda catena di DNA, ad opera dell’attività DNA
polimerasi-DNA dipendente, che utilizza come stampo la catena di DNA già
esistente. Dopo che la sintesi del filamento lineare di DNA si è completata nel
35
citoplasma, il DNA virale, sotto forma di complesso nucleoproteico, migra
verso il nucleo per essere integrato nel genoma della cellula ospite. La
struttura e la precisa composizione di questo complesso di preintegrazione
(PIC) sono sconosciute, ma è certo che fanno parte di questo complesso il
DNA virale, l’integrasi, la proteina di matrice e la vpr (57). Il trasporto del
complesso nel nucleo è un processo attivo che richiede ATP, ma è
indipendente dalla divisione cellulare (58) e permette ai lentivirus di replicare
anche in cellule differenziate non proliferative in contrasto con gli altri
retrovirus (59).
L’integrazione del DNA virale retrotrascritto nel genoma della cellula ospite è
assolutamente richiesta per un’infezione stabile e produttiva. L’integrasi è
l’enzima virale che interviene in questa reazione e risulta altamente conservato
tra gli isolati clinici di HIV-1. Una volta integrato, il DNA virale resta in
maniera permanente come provirus fino a che la cellula non muore. L’analisi
di sequenze cellulari che fiancheggiano i provirus rivela che HIV-1 si inserisce
preferenzialmente all’interno o vicino due classi di elementi ripetuti di DNA
nel genoma umano: L1 e Alu (60). Questi elementi sono trasposoni e mostrano
proprietà comuni con i retrovirus. Così, l’integrazione preferenziale di HIV-1
all’interno di questi elementi può riflettere la struttura cromatinica locale che è
più suscettibile all’acquisizione di un trasposone. Analisi in vitro del
meccanismo di integrazione provirale mostrano che l’intermedio per il
provirus covalentemente legato nel genoma cellulare è DNA virale lineare e
non circolare. Tuttavia la forma circolare, che non può replicare, può essere
36
ritrovata dopo l’infezione e può rimanere stabile per molti giorni al contrario
della forma lineare che è rapidamente degradata.
3.3 Espressione e regolazione dei geni virali
Il controllo della sintesi del genoma è complesso e coinvolge l’azione
combinata di elementi virali agenti in cis, transattivatori virali e diverse
proteine cellulari. Nel nucleo, il DNA provirale integrato è trascritto dalla
RNA polimerasi II cellulare per produrre RNA precursori della stessa
lunghezza del genoma. L’espressione del provirus porta alla formazione di tre
classi di trascritti: RNA genomico per la progenie virale, RNA messaggero per
la traduzione nel citoplasma di poliproteine Gag e Gag-Pol e precursori per
oltre 30 RNA messaggeri alternativamente spliced che sono tradotti nel
citoplasma per produrre glicoproteine di env e proteine accessorie.
Inizialmente in assenza del transattivatore tat, il livello di trascrizione
specifica virale è basso. Gli RNA precursori vengono trasportati attraverso un
meccanismo di trasporto rev-indipendente, ma prima del trasporto gli
spliceosomi eliminano gli introni dai trascritti multiply-spliced che vengono
tradotti per produrre le proteine regolatorie tat, rev e nef che ritornano al
nucleo. Con il procedere della trascrizione i livelli di tat aumentano e di
conseguenza aumenta l’attività trascrizionale stessa. Man mano che anche rev
si accumula, gli RNA precursori vengono trasportati al citoplasma mediante
37
un meccanismo rev-dipendente che non prevede lo splicing prima del
trasporto. In questa fase, vengono tradotti nel citoplasma mRNA virali
unspliced e singly spliced per le proteine gag, pol, env, vpr, vpu e vif. Rev,
quindi, agisce come uno chaperone molecolare interagendo con la sequenza
RRE presente in tutti i trascritti in corrispondenza del gene env.
3.4 Assemblaggio e liberazione del virus
Il primo evento nell’assemblaggio dei virioni è l’interazione tra il polipeptide
precursore del gene gag (Pr55), il polipeptide precursore Gag-Pol (Pr160) ed il
genoma virale per produrre un complesso nucleoproteico. Questo processo
può avvenire nel citoplasma o sulla membrana. La maturazione delle
poliproteine gag durante l’assemblaggio è mediato dal dominio della proteasi
nella Pr160 per produrre un nucleocapside maturo. Parallelamente, la gp120 e
la gp41 vengono inserite nella membrana plasmatica e in seguito
all’interazione della proteina di matrice con la coda citoplasmatica della gp41
il complesso nucleoproteico virale estrude o gemma attraverso la membrana
per produrre un virione maturo. Il meccanismo preciso di gemmazione è
sconosciuto.
38
4 PATOGENESI E STORIA NATURALE
DELL’INFEZIONE
Il virus dell’immunodeficienza umana riconosce come cellule bersaglio i
linfociti T, i macrofagi e le cellule dendritiche e pertanto attacca in modo
diretto, il sistema immunitario dell’ospite, per di più replica con maggiore
efficacia in queste cellule quando sono attivate. Così, una serie di eventi che
stimolano il sistema immunitario a difendersi da HIV-1, ironicamente lo
espongono a ripetuti cicli di attivazione e di conseguenza all’infezione e
distruzione di un maggior numero di cellule del sistema immunitario stesso.
Approfonditi studi sul meccanismo d’ingresso e di replicazione di HIV-1 nelle
cellule target e sullo sviluppo di una risposta immunitaria specifica messa in
atto dall’ospite, hanno permesso di organizzare una serie di informazioni
scientifiche relative alla patogenesi dell'infezione, ovvero alla trasmissione
virale ed alla progressione della malattia (24).
La principale via di trasmissione dell’infezione è rappresentata dai rapporti
omosessuali ed eterosessuali e la probabilità di infezione dipende dal numero
dei partners sessuali e dalle differenti pratiche sessuali (61). Il virus viene
trasmesso attraverso le secrezioni genitali, femminili e maschili, e il sangue
(cellule, plasma e fattori della coagulazione) (62). Una forte incidenza di
trasmissione si registra tra i tossicodipendenti a causa dell’uso di aghi
contaminati. Il virus può essere trasmesso dalla madre infetta al bambino sia
per via transplacentare sia attraverso il parto o in seguito all'allattamento al
39
seno. Contatti personali non sessuali, esposizione a saliva (63) contatti con
urine (64) ed esposizioni ad insetti (61) non sono mai stati direttamente
implicati nella trasmissione di HIV-1.
Le cellule bersaglio iniziali sono molto probabilmente quelle della linea
dendritica (monociti-macrofagi), le cellule di Langherans, e i linfociti nei tratti
genitali e rettali. L’infezione di nuove cellule nel sangue e la disseminazione
nell'organismo avviene attraverso il virus libero o per interazione cellulacellula. I maggiori bersagli di HIV-1 sono il sistema linforeticolare, il sistema
ematopoietico ed il sistema nervoso. Cellule bersaglio critiche per
l’immunopatogenesi sono le cellule dendritiche, i linfociti T CD4 + ed i
macrofagi-monociti.
Nonostante i devastanti effetti di HIV-1 sull’immunità dell’ospite, gli
individui infettati sviluppano una risposta umorale e cellulare contro gli
antigeni associati ad HIV-1. Anticorpi neutralizzanti sono prodotti dalla
maggior parte degli individui (62) e alcuni ricercatori hanno notato una
correlazione tra la progressione della malattia e i bassi titoli di anticorpi
neutralizzanti (65). Le glicoproteine dell’envelope sono i maggiori bersagli per
la neutralizzazione degli anticorpi e i diversi epitopi sulla gp120 hanno
mostrato l’abilità di evocare anticorpi neutralizzanti in animali da esperimento
(66, 67). Alcuni di questi sono in regioni ipervariabili (68) mentre altri sono in
aree conservate. La reattività ai prodotti del gene gag, ricercata attraverso
saggi immunoenzimatici (ELISA) sembra rispecchiare la progressione
dell'infezione. Infatti, la diminuzione di anticorpi circolanti anti-p24 e la
40
comparsa di antigeni p24 circolanti, corrisponde in genere alla diminuzione
dei linfociti T CD4 associata alla progressione della disfunzione immunitaria e
allo sviluppo dell'AIDS.
Anche la risposta cellulare è diretta contro antigeni associati ad HIV-1, infatti,
linfociti T citotossici reagiscono con i prodotti del gene env, pol, gag e geni
regolatori.
4.1 Sintesi del decorso dell'infezione da HIV
Il decorso clinico tipico dell’infezione da HIV-1 è stato chiaramente definito
in seguito all’isolamento di HIV-1 da pazienti con AIDS o con segni clinici
che precedono l’AIDS (69,70,71). Essa è caratterizzata essenzialmente da tre
fasi distinte e include una fase di infezione primaria, con una sindrome acuta
di variabile gravità, un periodo prolungato di latenza clinica e uno stadio finale
di malattia caratterizzato da un aumento della suscettibilità alle infezioni
opportunistiche e alle malattie neoplastiche (72).
4.1.1 Infezione primaria
La fase precoce dell'infezione nell’uomo è caratterizzata da un periodo di
incubazione di tre-sei settimane, può essere asintomatica, ma generalmente
41
culmina in una sintomatologia acuta a evoluzione rapida che si esaurisce
spontaneamente, in media in meno di 14 giorni. Dopo di che HIV-1 colonizza
l’intero tessuto linfoide e appare in circolo prima della sieroconversione. Da
questo momento in poi il virus può essere isolato dal plasma libero (73,74) e
di solito è di fenotipo non inducente sincizi (NSI). Nell’infezione primaria è
frequente la linfoadenopatia. Sono evidenti anche marcati cambi nella conta
dei linfociti con un rapporto CD4:CD8 rovesciato (<1), dovuto sia a un calo
dei CD4+, sia a un aumento delle cellule CD8+. Si riscontrano in questa fase
alti livelli di virus nel plasma, con un picco di viremia tra 106 e 107 copie/ml
(75,76) permettendo così la disseminazione del virus e lo sviluppo di
un’intensa risposta immunitaria, inclusa la sieroconversione, con produzione
di anticorpi specifici anti-HIV. In poche settimane una forte risposta
immunitaria, soprattutto cellulo-mediata, riduce il livello degli indici virali
mentre, parallelamente si osserva un recupero parziale e qualche volta totale
delle cellule T CD4+. Comunque la completa eliminazione di HIV-1 dal
plasma non accade nel decorso naturale dell’infezione acuta (75).
4.1.2 Fase di latenza clinica
L’inizio di questa fase corrisponde a una notevolissima riduzione della viremia
come conseguenza della risposta immunitaria. Ciò non corrisponde ad una
latenza virologica: infatti il virus è sequestrato negli organi linfoidi, soprattutto
42
all’interno delle cellule dendritiche follicolari, dove rimane attivo e continua a
replicare, permettendo la liberazione di particelle virali e di prodotti specifici
del virus. La fase di latenza clinica è caratterizzata in genere da un basso
livello di tutti gli indici di attività virale. A questo stadio l’isolamento virale
dal plasma è difficile a causa della bassa viremia e talvolta a causa di una
buona attività neutralizzante del siero, mentre è generalmente possibile isolare
il virus attraverso la cocoltivazione di cellule mononucleari linfonodali
stimolate con fitoemoagglutinina.
La durata della fase asintomatica può essere variabile (4) ed è legata alla
gravità dell’infezione primaria, alle caratteristiche dei virus infettanti, allo
stato di immunità dell’ospite (77) e all’uso di terapie antiretrovirali. In questa
fase i pazienti dovrebbero essere monitorati per il numero di linfociti T CD4 e
per la carica virale nel sangue, perché questi due valori insieme rappresentano
il più accurato metodo per la determinazione della prognosi del paziente.
4.1.3 Progressione della malattia: AIDS conclamato
La
progressiva
immunodeficienza
nelle
infezioni da
HIV
conduce
all’insorgenza di infezioni opportunistiche e neoplasie che caratterizzano
l’AIDS; tali patologie sono spesso multiple e contribuiscono ad un rapido
deterioramento clinico. Una volta accertata la diagnosi di AIDS conclamato, la
43
sopravvivenza è spesso meno di due anni, sebbene possa esistere una
considerevole variabilità, che dipende da fattori come l’età e la terapia.
L’entrata di HIV nel sistema nervoso centrale, che avviene precocemente nel
corso dell’infezione, è associata all’insorgenza di segni clinici e patologici in
questo comparto, nonché alla comparsa di diversi disordini neurologici. Le
alterazioni neurologiche si riscontrano nel 40-90% dei pazienti. In particolare,
bisogna evidenziare l’encefalite subacuta associata ad HIV-1, che è stata
chiamata AIDS dementia complex. La sindrome è caratterizzata da un
progressivo deficit cognitivo accompagnato da anormalità motorie e
comportamentali.
Come conseguenza della profonda soppressione dell’immunità cellulomediata, l’AIDS è caratterizzata dallo sviluppo di neoplasie che hanno una
prevalente eziologia virale.
Alcuni ricercatori tendono ad identificare una soglia negli indici molecolari di
attività virale come marker generale per la progressione irreversibile della
malattia. Due sono gli aspetti cruciali che possono essere analizzati per
stabilire le relazioni tra carica virale e progressione della malattia: i) la
variabilità da paziente a paziente nei parametri relativi all’attività di HIV-1 è
tale che può succedere che portatori totalmente asintomatici presentino indici
di replicazione più alti dei pazienti con AIDS conclamato e in questa
situazione solo il monitoraggio individuale potrebbe rivelare l’andamento
dell’infezione; ii) l’incremento dell’attività virale nella maggior parte dei casi
non è così netto come avviene in un improvviso evento patogenetico ma
44
sembra piuttosto essere graduale. Diversi studi (74,78) hanno dimostrato che
l’infezione da HIV-1 è lontana dall’essere una tipica infezione lentivirale,
infatti il turnover di cellule infettate e di virioni circolanti è assai rapido
durante l’infezione. In definitiva l’evoluzione dell’infezione può essere vista
come una continua corsa tra la distruzione cellulare e il rimpiazzamento delle
cellule stesse.
2.1.1 Soggetti lungo sopravviventi
Una piccola frazione dei soggetti infettati con HIV-1 (inferiore al 5%) mostra
una fase asintomatica più lunga e mantiene livelli superiori e più stabili di
CD4+, durante tale fase. Questi soggetti vengono definiti long-term non
progressors o slow progressors (79,80) e sono di solito persone infettate con
HIV che sopravvivono per più di sette anni con consistenti bassi livelli di
RNA virale nel plasma e poca o nessuna perdita dei linfociti T CD4+. Lo
studio di questi soggetti è particolarmente interessante per comprendere quali
fattori siano responsabili della loro abilità a sfuggire agli effetti dell’infezione
da HIV-1. L’interesse si concentra sui meccanismi di difesa della risposta
immunitaria, sulla variabilità virale e in particolare sullo studio dell’ospite
ponendo attenzione allo stato dei corecettori del virus, allo scopo di
individuare peculiarità nei progressori lenti che li rendono refrattari alla
spiccata patogenicità di HIV-1.
45
Numerosi studi hanno individuato mutazioni nel genoma umano, fornendo
evidenza che la resistenza verso HIV-1 può essere geneticamente determinata.
In particolare, variazioni nei geni che codificano per le chemiochine e i
recettori per le chemiochine sono risultati importanti sia per la suscettibilità
all’infezione da HIV-1 sia per la velocità di progressione della malattia dopo
avvenuta infezione. Esistono individui che, nonostante la ripetuta esposizione
al virus dell’immunodeficienza umana dovuta a frequenti intercorsi sessuali
con partners sieropositivi, sono rimasti indenni all’infezione da HIV.
Approfondite analisi hanno dimostrato che alcuni di questi individui, erano
omozigoti per una delezione di 32 bp a livello del gene codificante per il
CCR5 (mutazione definita CCR532). La mutazione CCR532, risulta in un
codone di stop prematuro e i soggetti omozigoti per questa mutazione
mostrano una completa mancanza di espressione di CCR5. Sebbene tale
mutazione conferisca una notevole resistenza all’infezione da HIV, essa non è
infallibile; sono stati pubblicati dati relativi ad almeno 8 soggetti omozigoti
per questa delezione, infettati con HIV-1 (81). Lo studio approfondito delle
caratteristiche genetiche del corecettore nell'ospite ha prodotto una serie di
lavori in cui la rallentata progressione e una migliore prognosi vengono
associati con lo stato e la funzionalità del/i corecettore/i; ad esempio, è stata
descritta una mutazione a livello del gene codificante per il CCR2b
(CCR2b64I) che, tuttavia, non è in grado di ridurre il rischio d’infezione.
Soggetti con tale mutazione presentano, 9-12 mesi dopo la sieroconversione,
46
una carica virale molto più bassa: in media 18.000 copie genomiche/ml contro
le 85.000 dei soggetti privi della mutazione (24).
Infine, va ricordato che anche alterazioni dei ligandi naturali per i corettori
possono influire sulla patogenesi, ad esempio soggetti omozigoti per la
mutazione a livello della regione 3’ non trascritta del gene codificante per il
fattore 1 di derivazione stromale (SDF-1) (ligando naturale del CXCR4),
presentano un ridotto e ritardato switch dai ceppi R5 agli X4, come avverrebbe
normalmente nelle fasi avanzate della malattia, probabilmente per un
meccanismo di competizione della proteina mutata e over-espressa per il
recettore CXCR4. Evidentemente il beneficio di tale mutazione, compare
tardivamente nel corso della malattia.
47
5 TERAPIA E CONTROLLO
5.1 Farmaci antivirali
A differenza di batteri e protozoi, che per la loro replicazione non dipendono
dalla cellula ospite, i virus sono dei parassiti intracellulari obbligati; per questo
motivo i farmaci antivirali devono essere selettivi per inibire le funzioni del
virus, senza danneggiare la cellula ospite.
Negli ultimi anni, gli studi di virologia molecolare, hanno consentito di
identificare funzioni virali specifiche che possono servire da bersagli per la
terapia antivirale. Per quanto riguarda il trattamento delle infezioni da HIV
viene approvato un numero sempre maggiore di farmaci. Per il trattamento
delle infezioni da HIV-1, attualmente sono disponibili quattro classi di farmaci
approvati dalla Food and Drug Administration Americana (FDA) e
comprendono inibitori della trascrittasi inversa, nucleosidici (NRTI) e non
nucleosidici (NNRTI), inibitori della proteasi virale (PI) e inibitori della
fusione. Gli inibitori della RT inibiscono la replicazione virale dopo l’entrata
del virus nella cellula ospite ma prima dell’integrazione, mentre i PI
prevengono la maturazione dei virioni permettendo il rilascio di particelle
virali non infettanti.
Inizialmente il trattamento degli individui infetti veniva praticato mediante la
cosiddetta monoterapia, che prevedeva l’uso di una singola classe di inibitori.
48
Questo approccio ha avuto un modesto impatto sui livelli di RNA virale
plasmatici e ha portato alla comparsa di ceppi virali farmaco-resistenti.
Nel 1996 si iniziò ad utilizzare un trattamento terapeutico combinato che prese
il nome di terapia antiretrovirale altamente attiva (HAART, highly active
antiretroviral therapy) e consiste nell’uso di tre o più farmaci appartenenti a
due o più differenti classi di inibitori. Grazie a questo utilizzo di combinazione
di farmaci, oggi la mortalità è drammaticamente diminuita, trasformando una
malattia inesorabilmente mortale in una patologia cronica a lunga decorrenza.
La tripla terapia tipica comprende un inibitore della proteasi o un inibitore
della trascrittasi inversa non nucleosidico, in combinazione con due inibitori
nucleosidici della trascrittasi inversa (82). Tale terapia riesce a diminuire la
replicazione virale e quindi la carica virale nei tessuti linfatici, a permettere il
recupero della risposta immunitaria verso patogeni opportunisti e a prolungare
la vita del paziente. Inoltre, mentre la monoterapia di solito produce
rapidamente l’insorgenza di forme mutanti di HIV resistenti al farmaco,
l’associazione terapeutica, che agisce a diversi livelli della replicazione virale,
in genere rallenta la selezione dei mutanti. Tuttavia, l’HAART non riesce ad
eradicare il virus guarendo le infezioni da HIV, infatti, quest'ultimo continua
ad essere presente anche dopo anni nelle cellule infettate, come le cellule
memoria CD4 e i macrofagi (83). La terapia HAART inoltre, non è ben
tollerata dai pazienti in cui può indurre un ampio range di effetti tossici di
differente severità (84), richiede esperienza da parte dei medici ed induce, se
pur più lentamente, l'insorgenza di resistenze multiple ai farmaci (85).
49
5.2 Resistenze ai farmaci
I problemi legati al fallimento terapeutico sono dovuti prevalentemente
all’insorgenza della resistenza ai farmaci e all’inadeguata concentrazione dei
farmaci causata a sua volta da fattori come la mancata aderenza del paziente,
la farmacocinetica e la tossicità. La farmaco-resistenza è una conseguenza di
una non completa soppressione della replicazione virale. L’alta velocità di
replicazione di HIV causa mutazioni che si accumulano nel genoma virale,
portando alla diminuzione dell’efficacia antivirale del farmaco e rendendo
gradualmente inattivo ciascun componente del regime terapeutico. La
comparsa di mutazioni è un fenomeno naturale dovuto agli elevati tassi di
replicazione di HIV-1 e alla incapacità della RNA-polimerasi di correggere
errori di trascrizione. Il trattamento terapeutico induce una pressione selettiva
sul virus favorendo l’insorgenza di ceppi virali resistenti in grado di
sopravvivere e replicare. Le varianti farmaco-resistenti possono essere
generate de novo durante la terapia, oppure possono originare da un pool di
provirus integrati prima dell’inizio della terapia, considerando che in un
singolo individuo HIV-1 esiste un miscuglio di varianti geneticamente
differenti, descritte come quasispecie. Anche in assenza di pressione selettiva
farmacologica viene quotidianamente prodotta una quantità di particelle virali
contenenti almeno una sostituzione correlata a resistenza ai farmaci e la
50
comparsa di mutazioni nel genoma virale che si traduce spesso in una
resistenza fenotipica a uno o più antiretrovirali.
La selezione di varianti resistenti nel corso della terapia dell’infezione da
HIV-1 è un fenomeno frequente (86) e dovuto al fatto che, in presenza di
farmaci, la quasispecie resistente diventa predominante poiché in grado di
replicarsi più efficacemente rispetto al virus wild-type. Spesso, inoltre, queste
varianti sono resistenti a più farmaci appartenenti alla stessa classe (crossresistenza) (87). Non è infrequente, a causa dell’enorme diffusione di HIV,
che alcuni soggetti vengano infettati da virus con resistenza primaria, ovvero
quella presente nel genoma virale già al momento dell’infezione e non insorta
successivamente a seguito della terapia antiretrovirale. Una ridotta carica
virale e l' aver acquisito l’infezione da HIV prima del 2000 sono fattori
predittivi di resistenza primaria ai farmaci. In questo caso la rapidità
dell’analisi è cruciale. Nel corso dell’infezione, infatti, la popolazione virale si
evolve; se il paziente non viene trattato con antiretrovirali, come spesso accade
nei primi tempi dopo il contagio, i ceppi resistenti eventualmente presenti
all’inizio possono modificarsi e perdere le loro caratteristiche, sicché
individuarli diventa più difficile; è consigliato, quindi, eseguire il test di
resistenza in tutti i pazienti naive prima di iniziare la terapia antiretrovirale. I
risultati di alcuni studi suggeriscono che, mediante il test di resistenza
genotipico, è possibile ottenere nei pazienti con resistenza primaria
un’efficacia di trattamento sovrapponibile a quella dei pazienti senza tale
resistenza. Aumenti transitori della viremia in corso di HAART, chiamati blip,
51
si associano all’emergenza di nuove varianti di HIV resistenti ai farmaci ma
non predicono un fallimento virologico.
Dato che HIV può sviluppare resistenza ad ogni farmaco introdotto nella
terapia, esiste la necessità di sviluppare nuovi composti e nuove strategie
terapeutiche. Sarà di fondamentale importanza in proposito individuare
molecole che inibiscono anche altre fasi del ciclo vitale del virus. Al momento
numerosi farmaci antiretrovirali sono candidati all’approvazione e si trovano
in vari stadi degli studi clinici e preclinici (trial clinici). Alcuni sono nuovi
composti che fanno parte delle classi di farmaci già esistenti ma che vengono
sviluppati per ottenere una migliore farmacocinetica, per diminuire lo sviluppo
di resistenze e per aumentare la potenza antivirale. Altri agenti in studio
appartengono a nuove classi di farmaci e sono diretti verso nuovi bersagli e
caratterizzati da meccanismi da azione unici.
Tra questi sono da menzionare gli inibitori dell’integrasi, gli inibitori
dell’entrata e quelli della maturazione. Tra gli inibitori dell’entrata, quelli che
sono in uno stadio più avanzato nella sperimentazione clinica sono i cosiddetti
antagonisti del corecettore CCR5.
52
5.3 Inibitori dell’entry
La terapia di combinazione (Highly Active Antiretrovaral Treatment,
HAART) con almeno tre agenti antiretrovirali efficaci sopprime
la
replicazione virale a così bassi livelli che la comparsa di varianti di HIV-1
farmaco-resistenti, se non prevenuta, viene almeno ritardata. Tuttavia il virus
si è dimostrato capace di sviluppare resistenze a qualsiasi molecola introdotta
in terapia e di frequente un mutante resistente ad un farmaco risulta resistente
anche agli altri inibitori della stessa classe. Pertanto, la continua ricerca di
nuovi farmaci che possano interferire con diverse funzioni virali ha portato
alla recente introduzione nella pratica clinica di una nuova generazione di
agenti antiretrovirali : gli inibitory dell’entry, l’Enfuvirtide (T20) il primo ad
essere approvato nella pratica clinica è un peptide sintetico che interferisce
con l’attività fusogena di gp41.
L’evidenza che i recettori per le chemochine sono utilizzati da HIV per entrare
ha fornito le basi per lo sviluppo di una nuova classe di inibitori dell’entry: tra
questi l’unico attualmente approvato nella pratica clinica è il Maraviroc
(Celsentri, UK – 427.857;Pfizer, NewYork, NY, USA) antagonista del
corecettore virale CCR5 (88,89).
Il Maraviroc è un antagonista funzionale per i ligandi naturali (chemochine
MIP-1α, MIP-1β e RANTES) di CCR5 espresso sulla membrana delle cellule,
ha una attività antivirale, in quanto inibisce in modo competitivo il legame di
gp120 a CCR5, è efficace a basse concentrazioni nanomolari ed ha una
53
permanenza fisica e funzionale nel comparto extracellulare tale da giustificare
un rialzo tardivo della viremia nei pazienti che assumono la terapia in modo
discontinuo, (90,91) inoltre il Maraviroc non induce l’internalizzazione del
recettore CCR5 rendendo il blocco funzionale permanente (92).
6
RAZIONALE DELLO STUDIO
Gli antagonisti del CCR5 di HIV stanno diventando sempre più importanti
come agenti terapeutici ed è pertanto fondamentale identificare quei pazienti
che possono giovarsi del trattamento con Maraviroc; il che implica la necessità
di conoscere quale virus è presente nel paziente e questo è possibile
utilizzando il saggio per la determinazione del tropismo, che può essere un test
fenotipico in vitro o un test genotipico (fenotipo virtuale).
Il test Trofile distribuito dalla Monogram Bioscences è stato fino a pochi mesi
fa il saggio fenotipico più utilizzato su scala mondiale, esso prevede
l’inserimento delle varianti virali del gene env amplificate dal paziente
all’interno di un plasmide replicativo env-deleto e l’utilizzo di un plasmide
contenente un marker bioluminescente a base di luciferasi, in seguito a
cotrasfezione si ottengono delle particelle virali replicative che sono utilizzate
per infettare due linee cellulari bersaglio una che utilizza esclusivamente il
corecettore CCR5 per penetrare nella cellula l’altra che utilizza solo il
corecettore CXCR4, in sostanza la quasispecie virale presente nel paziente è in
questo modo riprodotta in vitro.
54
Le cellule infettate con successo emetteranno luce durante la replicazione
virale. Questo test presenta tuttavia notevoli svantaggi quali: i) il tempo di
attesa per la risposta, ii) una resa non soddisfacente per quei campioni con
viremia <500 copie/ml e, iii) costi elevati.
A causa di tali limitazioni, la comunità scientifica ha puntato l’attenzione sulla
messa a punto di test genotipici per la predizione del tropismo. I vantaggi che
il test genotipico offre sono: i) la rapidità d’esecuzione, ii) la possibilità di
ottenere un risultato su campioni di pazienti con viremia <500 copie/ml, iii)
costi contenuti.
È stato ampiamente dimostrato attraverso studi geno-fenotipici che la regione
V3 è la principale responsabile del tropismo virale, pertanto la sequenza V3
accoppiata all’utilizzo di un algoritmo interpretativo (che studia la funzione
del gene) per la definizione del fenotipo, così detto fenotipo virtuale, è il modo
più semplice per identificare il tropismo del virus, tuttavia a causa dell’elevata
variabilità del gene env i saggi genotipici possono essere inaffidabili,
specialmente a causa della possibile presenza di sequenze minoritarie che non
vengono evidenziate con i comuni metodi di sequenziamento. Al contrario, i
saggi biologici, specialmente quelli basati su metodi clonali, sono sempre in
grado di valutare in modo completo il fenotipo effettivo (la quasispecie virale).
A tal fine abbiamo messo a punto un vettore di espressione fluorescente che si
caratterizza per consentire il clonaggio delle sequenze V3 amplificate dal
paziente di interesse. Questo plasmide è dotato di un gene per la proteina
fluorescente GFP che viene espressa quando il virus replica nelle cellule, è
55
pertanto possibile conoscere sia il fenotipo conferito da quella specifica
sequenza V3 che la capacità replicativa della variante V3 attraverso l’analisi
fluorimetrica.
Obiettivo di questo lavoro è confrontare il saggio fenotipico home-brew con il
saggio commerciale Trofile nella sua forma più sensibile l’Enhanced
sensitivity Trofile assay (Esta), in breve la regione V3 del gene Env di HIV
amplificata dal plasma di 38 pazienti HIV-1 positivi, il cui coreceptor usage
era stato precedentemente definito con il saggio Esta, è stata sequenziata e
clonata nel nostro plasmide di espressione; è stato quindi valutato il fenotipo
virtuale e il tropismo in vitro (saggio fenotipico) dei 117 cloni replicativi.
Il fine ultimo del nostro lavoro è conseguire una valutazione comparativa tra i
diversi saggi fenotipici e tra i saggi fenotipici e saggi virtuali nella definizione
del coreceptor usage.
56
7 MATERIALI E METODI
7.1 Costruzione del vettore pNLmodV3GFP
Al fine di determinare se il saggio genotipico accoppiato all’utilizzo di un
algoritmo predittivo della funzione del gene possa dare informazioni
equivalenti al saggio biologico commerciale (test Trofile), abbiamo modificato
un saggio fenotipico home-brew, precedentemente messo a punto in questo
laboratorio e validato per l’inserimento della regione V3 amplificata da
pazienti HIV-1, in modo da renderlo capace di esprimere la GFP o Green
Fluorescent Protein. Questo vettore permette di studiare l’entry del virus a
poche ore dalla trasfezione/infezione analizzando la fluorescenza delle cellule
infettate dalle chimere virali.
In sintesi la GFP è stata amplificata dal vettore pEGFP-C1 utilizzando i
primers GFPS e GFPAS, questi primers sono stati progettati in modo tale da
contenere al loro interno il sito di restrizione dell’enzima BplI; il prodotto
ottenuto mediante PCR è poi stato digerito alle estremità con l’enzima BplI
con una reazione a 37°C per 2h, con il medesimo enzima il vettore
pNLmodV3 è stato linearizzato a livello del gene NEF e la GFP è stata
inserita in modo da mantenere il frame di lettura. Poiché l’enzima BplI non si
disattiva al calore, per interromperne l’attività enzimatica vettore ed
57
amplificato GFP sono stati purificati con kit gel extraction Quiagen. I prodotti
di purificazione sono stati poi verificati su gel d’agarosio al 1% TBE1X.
Successivamente
0.5µl di vettore e 4µl di amplificato GFP (digeriti e
purificati) sono stati ligati o/n a temperatura ambiente in un volume finale di
10µl contenente tampone di ligazione 10X e T4-ligasi HC (Promega, Madison
WI U.S.A.).
Infine, 2 l del prodotto di ligazione sono stati utilizzati per la trasformazione
mediante shock termico di una sospensione di cellule batteriche competenti
(JM109 Competent Cells, Promega, Madison WI U.S.A.) seguendo il
protocollo consigliato dalla ditta produttrice. La sospensione batterica è stata
seminata su LB (Luria Bertani: 10g/l bactotriptone, 5g/l estratto di lievito,
10g/l NaCl pH7), con Agar e contenente ampicillina (100g/ml) come
marcatore di selezione dei batteri resistenti. Le colonie ottenute sono state
controllate per la presenza dell’inserto GFP mediante amplificazione con i
primers NEF3 e GFPINTAS. Alcune colonie risultate positive allo screening,
sono state fatte crescere in SB liquido con ampicillina (100g/ml).
È stata quindi eseguita una estrazione plasmidica dai batteri cresciuti in
terreno SB liquido (35g/l bactotriptone, 20g/l estratto di lievito, 5 g/l NaCl
pH7), per ottenere il nuovo plasmide pNLmodV3GFP (Wizard Plus SV
Minipreps DNA Purification System, Promega, Madison WI U.S.A.).
Infine, per controllare che l’inserzione della GFP sia avvenuta correttamente e
che il frame di lettura sia rimasto invariato sono state effettuate due reazioni di
sequenza con GFPINTS e GFPINTAS. Inoltre l' esattezza dell'intero vettore è
58
stata verificata mediante digestione con l’enzima HIND III (Promega,
Madison WI U.S.A.)
7.2 Origine delle regioni V3
Le regioni V3 sono state amplificate a partire dall’RNA estratto con il kit
QIAamp Viral Rna MiniKit Quiagen dal plasma di 38 pazienti HIV-1 positivi
precedentemente sottoposti al test Trofile.
Per la retrotrascrizione dell’RNA di HIV-1 è stato utilizzato il primer
antisenso V52 (50pmoli) e 200U di trascrittasi inversa M-MLV Invitrogen a
37°C per 30 minuti in un volume finale di 20µl in presenza buffer di reazione
1X(Euroclone), 10mM MgCl2 (Euroclone), 1mM di ciascun dNTP
(Invitrogen), e 10U di inibitore della RNAsi (Ribonuclease inhibitor Porcine
Liver, Takara).
Il cDNA ottenuto dopo la retrotrascrizione è stato utilizzato per la reazione di
PCR in tampone 1X contenente 0.2mM di ciascun dNTP, 50pmoli del primer
senso V31 e 2,5U di Taq polimerasi (Euroclone) in un volume finale di 50µl.
Il profilo di amplificazione dopo una denaturazione iniziale a 94°C per 2
minuti, una denaturazione a 94°C per 15secondi, annealing a 50°C per 15
secondi, estensione per 50 secondi a 72°C è stato ripetuto per 40 cicli seguiti
da un’estensione finale a 72°C per 10 minuti. 5µl del prodotto di PCR sono
stati quindi utilizzati per una reazione di amplificazione nested utilizzando la
coppia di primer V3s e V3as utilizzando una DNA polimerasi altamente
processiva e con attività esonucleasica 3'-5', anche nota come attività proofreading (Pwo DNA polymerase Roche Diagnostics, Milano, Italia), in un
volume finale di 50l contenente: tampone di reazione 1X [10 mM Tris-HCl
pH 8.85, 25 mM KCl, 50 mM di (NH4)2SO4], 2mM MgSO4, 25 pmoli di
59
ciascun primer, 100M di ciascun dNTP, 1U di DNA polimerasi. L’
amplificazione prevedeva una fase di denaturazione (94C per 15 secondi),
una di annealing (53°C per 15 secondi) e una di estensione (72C per 45
secondi), ripetute per 30 cicli totali ed una estensione finale di 10 minuti a
72°C. Tutte le regioni V3 contengono all'inizio e alla fine una sequenza
identica, rispettivamente di 25 e 21 bp, che corrisponde alla sequenza dei due
primer utilizzati per l'amplificazione (tabella 2). I due primer sono stati
disegnati in modo tale che la sequenza inserita rimanga in frame per la
traduzione della proteina di env. La presenza e la qualità di un prodotto di
amplificazione della lunghezza appropriata è stata controllata su gel di
poliacrilamide al 10%.
7.3 Sequenziamento diretto delle regioni V3
Prima di essere clonata la regione V3 è stata sequenziata, in particolare sono
stati sequenziali gli amplificati ottenuti dalla prima pcr, V31-V52.
Tutte le sequenze sono state ottenute utilizzando la tecnica di Sanger e coll.
(1977), come modificata da Chen e Seeburg (1985). Il metodo si basa sul
blocco dell’estensione della catena di DNA crescente quando dideossiribonucleosiditrifosfati (ddNTP) vengono incorporati dalla DNA
polimerasi. DNA stampo da sequenziare, DNA polimerasi termostabile, e
primer di sequenza sono aggiunti ad una miscela di dNTP/ddNTP per una
reazione di sequenziamento ciclico. I prodotti della reazione rappresentano
una popolazione di molecole di DNA neosintetizzate che terminano in
60
ciascuna delle posizioni nucleotidiche del frammento stampo, perciò tutti
questi frammenti hanno in comune l’estremità 5’ ma differiscono in
lunghezza. Nel nostro laboratorio il sequenziamento del DNA è stato ottenuto
utilizzando un Kit in commercio (BigDye Terminator V1.1 Cycle Sequencing
Kit, Applied Biosystems CA U.S.A.) in cui ciascun ddNTP è marcato da un
diverso fluorocromo, che dà una specifica emissione di fluorescenza quando
eccitato da un raggio laser; pertanto la reazione di sequenza può avvenire in
un’unica fiala ed i prodotti della reazione vengono separati durante la corsa
elettroforetica in un gel denaturante ad alto potere risolutivo, e
successivamente vengono letti dal sequenziatore automatico a raggio laser
(3130 Genetic Analyzer, Applied Biosystems) in grado di riconosce la diversa
emissione dei quattro fluorocromi.
Le reazioni di sequenza sono state fatte in un volume finale di 10 l contenenti
il DNA stampo (amplificato), 2l di miscela di reazione pronta all'uso
(Applied Biosystems), 2µl di buffer di diluizione (Applied Biosystems)e 250
pmoli di primer. Questa miscela è stata sottoposta a 25 cicli di denaturazione a
96°C per 10 secondi, annealing a 50°C per 5 secondi ed estensione a 60°C per
2 minuti. Dopo precipitazione con etanolo e sospensione del precipitato in
formamide, le reazioni venivano trasferite su piastre a 96 pozzetti e caricate
nel sequenziatore . Per ogni amplificato sono state effettuate 4 reazione al fine
di ottenere una sequenza dettagliata della regione V3 utilizzando i primers
V3S6, V3S5, V3AS1 E V3AS3BIS.
61
Al fine di ottenere la predizione del fenotipo è importante determinare il
livello di significatività, cioè di selezionare quanto conservativa è la
determinazione del virus X4, questo livello di significatività è determinato dal
tasso di falsa positività per la classificazione di un virus X4. Questo tasso detto
FPR (False Positive Rate) è un cut off, una soglia al di sopra della quale i virus
sono definiti R5 e al di sotto X4. Attualmente l’FPR utilizzato dalla comunità
scientifica è del 10%; questo valore sembra raggiungere un buon
compromesso tra sensibilità e specificità.
Il fenotipo virtuale viene definito mediante l’utilizzo del programma
implementato nel pacchetto geno2pheno per l’analisi del corecettore, poiché
nessuna classificazione è così perfetta da distinguere tutti gli R5-tropici da
tutti gli X4-tropici bisogna trovare un compromesso attraverso la definizione
di un valore cut-off, che identifica un livello di significatività. La soglia
stabilita permette di decidere quanto conservativa debba essere la predizione
per un virus X4 e riflette la probabilità di definire falsamente un virus R5
come X4, in tal senso si valuta per ogni sequenza un tasso di falsa positività
(FPR) che esprime la probabilità per un virus non X4 di essere classificato
erroneamente X4. In sostanza le sequenze che mostrano un valore inferiore
alla soglia stabilita utilizzano con alta probabilità il CXCR4 quelle che hanno
un valore di FPR superiore alla soglia al contrario utilizzano CCR5; tanto più
è alto il valore cut-off arbitrario tanto maggiore è la predizione di un virus X4,
al contrario tanto più basso quel valore tanto più virus X4 saranno classificati
come R5 con il rischio di somministrare il farmaco ad un paziente in cui si
62
potrebbe più facilmente selezionare, sulla base della sequenza, una variante
X4-tropica.
7.4 Clonaggio molecolare delle regioni V3in pNLmodV3GFP
Definito il fenotipo virtuale di ogni paziente si è proceduto al clonaggio della
sola regione V3.
Gli
amplificati
V3s-V3as
ottenuti
sono
stati
clonati
nel
vettore
pNLmodV3GFP appositamente preparato (Figura 3)
Brevemente, 30 ng di vettore pNLmodV3C5, precedentemente linearizzato
con NRU I, sono stati utilizzati per una reazione di ligazione in presenza
tampone di reazione 10X, 10U di T4 DNA ligasi HC, (Promega, Madison WI
U.S.A.), un'aliquota del prodotto di amplificazione in modo di avere un
corretto rapporto molare inserto/vettore, in un volume finale di 10 l. La
reazione è stata mantenuta a temperatura ambiente per quattro ore. La
trasformazione delle cellule competenti è stata eseguita per shock termico,
come già descritto, utilizzando 2 l del prodotto di ligazione.
Le piastre sono state incubate a 37°C per tutta la notte. Al fine di ottenere un
migliore isolamento dei batteri trasformati, colonie isolate sono state seminate
su una nuova piastra d’agar, e lasciate crescere a 37C per 6 ore circa. Poiché
si tratta di una ligazione di frammenti blunt-end, l' amplificato può inserirsi in
due orientamenti alternativi, di cui uno soltanto è corretto per il frame di
63
lettura. Per questo motivo l’orientamento dell’inserto è stato valutato mediante
PCR utilizzando il primer V3.1, che si ibrida sul vettore a monte dell'inserto,
ed il V3.2, presente sull'inserto; soltanto i cloni aventi l’inserto nel giusto
orientamento danno il prodotto atteso. Le colonie singole isolate attraverso il
passaggio su una seconda piastra sono trasferite nelle provette di reazione
contenente 50 l della miscela di amplificazione (tampone di PCR 1X,
25pmoli di ciascun primer V3.1 e V3.2, 0.2 mM di ciascun dNTP, 2.5 U di
DNA polimerasi). Al fine di ottenere una completa lisi dei batteri, si esegue
una denaturazione iniziale a 94C per 5 minuti, seguita da 25 cicli con il
seguente profilo di amplificazione: denaturazione a 94°C per 20 secondi,
annealing a 55°C per 30 secondi, estensione a 72°C per 45 secondi. I prodotti
di reazione vengono controllati su gel di poliacrilamide al 10% per identificare
i cloni contenenti l’inserto amplificato della lunghezza attesa.
Le colonie positive allo screening vengono seminate in terreno SB liquido
contenente ampicillina (100mg/ml), la coltura viene mantenuta a 37°C per una
notte sotto agitazione continua, la torbidità del brodo di coltura testimonierà
l’avvenuta crescita batterica. Si procede, quindi, all’estrazione plasmidica
mediante il metodo della lisi alcalina dei batteri seguendo il protocollo della
ditta produttrice del kit di estrazione (Wizard Plus SV Minipreps DNA
purification System, Promega, Madison WI U.S.A.). La concentrazione del
plasmide purificato viene calcolata tramite lettura spettrofotometrica, mentre
la qualità del prodotto viene valutata dopo digestione con l’enzima Hind III
(Promega, Madison WI U.S.A.) che taglia cinque volte il plasmide
64
ricombinante. Il materiale digerito viene caricato su gel di acrilamide al 10% e
le cinque bande della lunghezza attesa, vengono confrontate con quelle di
pNLmodGFP, digerito nella stessa seduta.
7.5 Sequenziamento delle chimere virali
La tecnica del sequenziamento è stata utilizzata anche per controllare l' intero
pNLmodGFP e per verificare l’esattezza delle sequenze dei plasmidi
ricombinanti estratti dalle colture batteriche prima di utilizzarli per le
trasfezioni della linea cellulare U87 CD4+CCR5+/CXCR4.
La regione V3 dei plasmidi ricombinanti è stata sequenziata utilizzando il
primer V3.1. La regione sequenziata comprende tutta la regione V3 inserita
nel vettore.
7.6
Colture cellulari
Per studiare il fenotipo dei cloni molecolari ricombinanti di HIV-1, venivano
utilizzate le linee cellulari di glioma umano U87/CCR5 e U87/CXCR4,
ingegnerizzate in modo da esprimere il CD4 e i corecettori per HIV-1.
Queste cellule venivano mantenute in coltura aderente in terreno DMEM
(Dulbecco’s Modified Eagle Medium), 10% FCS (Siero Bovino Fetale),
aminoacidi non essenziali (NEAA), 0,5X antibiotici ed antimicotici e Hepes
Buffer 12,5mM.
65
G418 (300µg/ml) e puromicina (1µg/ml) venivano aggiunti al terreno di
crescita per mantenere sotto selezione l’espressione del CD4 e dei corecettori.
Le cellule venivano incubate a 37°C in atmosfera al 5% di CO 2 ed al 95% di
umidità in fiasche da 25cm2 (T25).
Raggiunta la confluenza, le colture venivano passate una o due volte la
settimana e trattate con tripsina ed EDTA per permettere il distacco dal
substrato; un’aliquota della sospensione cellulare veniva, quindi, introdotta in
nuove fiasche in presenza di terreno completo.
7.7
Trasfezioni
Le cellule U87/CCR5 e U87/CXCR4 venivano trasfettate con i plasmidi
ricombinanti mediante uso del reagente Lipofectina (Invitrogen).
Si tratta di una sospensione di liposomi con membrana lipidica carica
positivamente. Grazie a questa carica possono legare sia le cellule, per affinità
con la membrana plasmatica, che il DNA plasmidico, favorendone l’ingresso
fino al nucleo delle cellule. Successivamente venivano seminate su una piastra
da 96 pozzetti Il giorno precedente la trasfezione, le cellule confluenti
venivano staccate con tripsina, risospese e contate nella camera di Burker.
Successivamente venivano seminate su piastra nera da 96 pozzetti con fondo
piatto e trasparente (Corning, NY USA), in modo da depositarne circa 6500
per pozzetto in 150µl di terreno completo con 10% di FCS. Il giorno dopo le
cellule ricoprivano circa l’80% della superficie del fondo ed erano pronte per
66
la trasfezione. Per la trasfezione venivano preparate due soluzioni separate:
l’una contenente il plasmide ricombinante diluito nel terreno senza antibiotici
e FCS, l’altra contenente la lipofectina diluita sempre nello stesso terremo
(0,4µl di lipofectina in 29,6µl di terreno per pozzetto).
Dopo circa 45 minuti, le due miscele venivano unite e incubate a 37°C per 15
minuti.
Il terreno delle cellule seminate il giorno precedente veniva aspirato e al suo
posto veniva aggiunta la miscela di trasfezione. Dopo un’incubazione di 5-6
ore, la miscela veniva, a sua volta, rimossa per aspirazione e sostituita con
150µl di terreno completo.
Il plasmide entrato nelle cellule ha la possibilità di esprimersi in modo
transitorio e la proteina di fusione GFP-NEF si fonde con la membrana
plasmatica della cellula infettata rendendola fluorescente. Dopo 6 giorni
veniva effettuata la lettura della fluorescenza grazie all’utilizzo dello
strumento Fluorskan Ascent, un fluorimetro per micropiastre; la validazione
dello stesso è stata confermata da numerosi esperimenti effettuati in replica i
quali hanno dimostrato una correlazione positiva fra questa lettura e i valori
ottenuti mediante il dosaggio della p24 (p24 Ag) di Hiv presente nel
sovranatante delle cellule infettate.
Il
dosaggio di antigene p24, mediante saggio immunoenzimatico ELISA
(Abbott AXSYM™ system) è stato comunque effettuato al fine di quantificare
gli
stock
virali
prodotti
dalle
trasfezioni,
67
rendendo
possibile
la
standardizzazione delle infezioni che sono state realizzate con i vari virus
ricombinanti
Figura 3: Strategia utilizzata per il clonaggio delle sequenze V3 in
NlmodΔV3GFP.
68
7.8 Primers oligonucleotidici
La coppia di primers GFPS/GFPAS è stata utilizzata per la costruzione del
vettore di clonaggio pNLmodV3GFP.
I primers V3S6, V3S5, V3AS1 E V3AS3BIS sono stati utilizzati per il
sequenziamento diretto degli amplificati V31-V52.
Tutte le regioni V3 inserite nel vettore, sono state amplificate utilizzando la
coppia di primers V3s-V3as la cui sequenza rappresenta il consensus derivato
dall'allineamento di tutte le sequenze depositate in banca dati. Il primer V3.1 è
stato utilizzato per controllare, mediante sequenziamento, l'esatta inserzione
della sequenza V3 nei plasmidi ricombinanti.
Il nome e la sequenza dei primers impiegati sono riportati in Tabella 1.
Primer
Lunghezza
Senso
Sequenza nucleotidica dal 5' al 3'
V3.1
28 nt
+
TCAGCACAGTACAATGTACAGATGGAAT
V5.2
23 nt
-
TTCTTGGGAGCAGCAGGAAGCAC
V3.2
28 nt
-
TTTAATTGTGGAGGGGAATTTTTCTACT
V3s
25 nt
+
TACAGCTGAATGAATCTGTAGAAAT
V3-as
21 nt
-
TATTCCATTTTGCTCTACTAA
GFPS
22nt
+
gctgagccaATGGTGAGCAAGG
GFPAS
21nt
-
gctcaggTCGAGATCTGAGTC
NEF3
24 nt
+
ACATACCTAGAAGAATAAGACAGG
GFPINTAS
21 nt
-
AAGTCGTGCTGCTTCATGTGG
69
V3S6
20 nt
+
CTGTTAAATGGCAGTCTAGC
V3S5
20 nt
+
GTTAAATGGCAGTCTAGCAG
V3AS1
21 nt
-
GAAAAATTCCCCTCCACAATT
V3AS3BIS
18 nt
-
CAATTTCTGGGTCCCCTC
Tabella 1: Elenco dei primers utilizzati in questo lavoro.
70
8
RISULTATI
8.1
Caratteristiche dei pazienti
Questo studio comprende 38 pazienti HIV-1 positivi con una viremia>100
cp/ml e naïve al Maraviroc. Le loro caratteristiche cliniche, virali e
immunologiche sono riassunte nella tabella 2.
Numero di pazienti
38
Sesso M/F
22/16
Età
26-84 (mediana=43)
Genotipo:
B
C
F
G/B
CRF02
27
4
2
1
4
Viremia cp/ml
CD4 cell/mmc
Terapia
Fenotipo Esta
109-867.086
(mediana=83.580)
2-786 (mediana=303)
si=24
no=10
non aderenti=4
R5=25
X4/DM=9
NR=4
Tabella 2: Caratteristiche cliniche dei pazienti analizzati
71
8.2
Validazione del saggio utilizzato per il clonaggio di sequenze
V3 in pNLmodV3GFP
Prima di effettuare i saggi di fenotipo ricombinante, la strategia sperimentale
basata sul clone molecolare pNLmodΔV3GFP è stata validata clonandovi sia
la regione V3 di NL(CXCR4tropico) che di AD8(CCR5 tropico), in modo da
ricostruire la sequenza completa del virus parentale. La replicazione del virus
chimerico, valutata dopo infezione su cellule U87/CCR5 e U87/CXCR4, ha
dimostrato che il virus ricombinante contenente la V3 wild-type di NL-AD8
replica come il virus originario. Questi risultati mostrano che la strategia usata
è affidabile ed è in grado di fornire informazioni utili per la valutazione del
fenotipo ricombinante e quindi del coreceptor usage della V3 amplificata dai
pazienti analizzati. Come descritto nei materiali e metodi l’RNA virale è stato
estratto dal plasma di 38 pazienti e sottoposto a RT-PCR, l’amplificato V3 è
stato poi clonato nel vettore Home-Brew .
Per ogni chimera virale ottenuta è stata effettuata una trasfezione in doppio, e
l’intero esperimento ripetuto in tre repliche, perciò il valore di fluorescenza
ottenuto per ognuno dei 117 cloni corrisponde alla media di sei valori ottenuti.
72
8.3 Confronto tra saggio fenotipico Home Brew ed ESTA
Sono state ottenute 117 chimere virali dal plasma di 38 pazienti HIV-1 positivi
di cui era noto il coreceptor usage determinato dal saggio Trofile.
I pazienti HB (home brew) sono stati valutati nel fenotipo considerando ogni
clone virale ottenuto dal campione, se tutti i cloni del paziente risultavano
essere R5, il paziente veniva valutato come R5, se emergeva anche un solo
clone X4/DM allora il paziente veniva valutato come X4/DM.
Il saggio ricombinante home brew ha identificato 90 cloni con fenotipo R5
corrispondenti a 28 pazienti, 20 cloni con fenotipo X4/DM corrispondenti a
9 pazienti e tutte le chimere virali ottenute da 1 paziente sono risultate essere
non replicative ( il paziente è risultato essere un X4/DM con Esta).
73
Il saggio Esta ha identificato 25 pazienti con fenotipo R5, 9 pazienti con
fenotipo D/M e 4 NR .
Come si può notare c’è una quasi perfetta corrispondenza tra il saggio
ricombinante home brew e il test commerciale; i quattro campioni che il
saggio Esta
ha valutato come non replicativi, clonati nel nostro vettore
ricombinante sono risultati essere rispettivamente due con fenotipo R5 e
altrettanti con fenotipo X4/DM.
Il nostro test si mostra pertanto più sensibile rispetto al test commerciale Esta
perché ha una maggior probabilità di definire il coreceptor usage.
8.4 Valutazione del test genotipico Geno2Pheno
L’algoritmo geno2pheno è attualmente riconosciuto come lo strumento
interpretativo più affidabile per la determinazione del tropismo, data la
sequenza. L’algoritmo geno2pheno ci fornisce un valore quantitativo, l’FPR o
False Positive Rate, che è una misura della probabilità che il virus ha di
utilizzare il corecettore CCR5 per entrare nella cellula. Il cut-off per
discriminare i virus CCR5 tropici dai virus CXCR4 tropici attualmente
consigliato dal National Reference Centre for Retrovirus in Germania è del
10%, noi abbiamo, tuttavia, nella nostra valutazione preso in considerazione
anche il cut off del 5% e del 20%.
74
La concordanza tra geno2pheno e saggio ricombinante home brew è del
90,9%, 92,7% e 80,9% quando la soglia di FPR è rispettivamente del 10%,
del 5% e del 20%.
La specificità, che misura la concordanza tra geno2pheno e saggio home brew
nell’identificazione degli R5 tropici (falsi negativi) raggiunge il 96% quando
la soglia stabilita è del 5%, ma otteniamo una sensibilità, che misura la
concordanza nel rilevare i virus CXCR4 tropici (veri positivi), massima
quando la soglia stabilita è del 10% e del 20%, tuttavia se la soglia fosse
fissata al 20% significherebbe che molti virus sarebbero identificati come
probabili CXCR4 quando sono in realtà CCR5 tropici, ovvero che molti che
potrebbero beneficiare della terapia con il Maraviroc nella pratica non lo
faranno.
Come mostra la tabella 4, il nostro lavoro sostiene la raccomandazione di
fissare come soglia il 10% in quanto a questo valore si ottiene una sensibilità
massima nell’identificazione dei CXCR4 e una buona specificità nella
G2P
FPR
valutazione dei CCR5 tropici.
<10
>10
G2P
FPR
TOTALI
<5
>5
G2P
FPR
TOTALI
<20
>20
TOTALI
X4/DM
R5
TOTALI
20
0
20
15
5
20
20
0
20
10
80
90
3
87
90
21
69
90
30
80
110
18
92
110
41
69
110
CONCORDANZA
%
SENSIBILITA’
(95%CI)
90,9
1,00
92,7
80,9
SPECIFICITA’
(95%CI)
ACCURATEZZA
(95%CI)
0,88
0,94
0,81 - 0,95
0,89 - 0,98
0,75
0,96
0,85
0,56 – 0,94
0,92 – 1,00
0,78 – 0,92
0,76
0,88
0,82 – 0,94
1,00
0,67 – 0,85
Tabella 4: Valutazione dei risultati ottenuti con il test Geno2Pheno utilizzando
come riferimento il saggio fenotipico Home Brew (7 cloni sono risultati NR)
75
La concordanza tra geno2pheno e saggio commerciale Trofile è dell’ 88,2%,
85,2% e 76,4% quando la soglia di FPR è rispettivamente del 10%, del 5% e
del 20%.
La tabella 5 mostra che quando il test geno2pheno viene paragonato al test
Trofile i valori di specificità e sensibilità peggiorano sensibilmente, e si
conferma come miglior scelta la soglia di FPR il 10%.
Le tabelle mettono inoltre in evidenza come il nostro test ricombinante home
brew sia più sensibile e più accurato, quando paragonato al test genotipico,
G2P
FPR
del test commerciale Trofile.
<10
>10
G2P
FPR
TOTALI
<5
>5
G2P
FPR
TOTALI
<20
>20
TOTALI
X4/DM
R5
TOTALI
7
2
9
5
4
9
8
1
9
2
23
25
1
24
25
7
18
25
9
25
34
6
28
34
15
19
34
CONCORDANZA
%
88,2
85,2
76,4
SENSIBILITA’
(95%CI)
SPECIFICITA’
(95%CI)
ACCURATEZZA
(95%CI)
0,77
0,92
0,84
0,50-1,04
0,82 – 1,02
0,72 - 0,96
0,55
0,96
0,75
0,23 - 0,87
0,88 – 1,03
0,60 – 0,90
0,88
0,72
0,67-1,09
0,54 – 0,90
0,80
0,67 – 0,93
Tabella 5: Valutazione dei risultati ottenuti con il test Geno2Pheno utilizzando
come riferimento il saggio commerciale Trofile (4 campioni sono risultati NR)
76
8.5 Capacita’ replicativa delle varianti virali clonate
La capacità replicativa delle chimere virali è stata valutata mediante
trasfezione con la lipofectina delle cellule U87/CCR5 e U87/CXCR4 come
descritto nei materiali e metodi.
I valori riportati nei grafici di seguito sono stati ottenuti calcolando la media
sui risultati di tre esperimenti effettuti in doppio. La capacità replicativa di
ogni variante virale è stata rapportata a quella del virus wild-type a cui è stato
attribuito un valore pari a 1. Le barre in alto ad ogni colonna rappresentano le
deviazioni standard.
Come è possibile notare alcune varianti virali mostrano una ridotta capacità
replicativa rispetto al wild-type; altre invece hanno una fitness molto più alta,
non abbiamo notato alcuna correlazione tra questo andamento e il valore di
FPR attribuito dall’algoritmo geno2pheno alla sequenza del gene V3 di ogni
chimera virale valutata.
77
L’elevata complessità del pattern aminoacidico della regione V3 non ha reso
possibile correlare l’andamento della fitness di ogni variante virale con la
rispettiva sequenza genetica.
78
8.6 Analisi filogenetica delle sequenze virali
La figura 4 mostra l’albero filogenetico ottenuto confrontando tutte le
sequenze dei 117 cloni corrispondenti a 38 pazienti.
La ricostruzione filogenetica è stata ottenuta mediante allineamenti multipli
utilizzando l’algoritmo relativo al metodo di Clustal che raggruppa le
sequenze in cluster esaminando le distanze tra tutti gli appaiamenti, le
sequenze sono state analizzate insieme al ceppo HIV-1 MN del clade-B
utilizzato come sequenza di riferimento.
Figura 4
79
L’albero mostra che le varianti ottenute dai diversi campioni formano cluster
separati in modo statisticamente significativo mediante analisi di bootstrap,
questo significa che tutti i cloni derivano dai pazienti e non sono frutto di
contaminazioni di laboratorio o riarrangiamenti genetici.
9 DISCUSSIONE E CONCLUSIONI
È stato ampiamente dimostrato attraverso studi geno-fenotipici che la regione
V3 è la principale responsabile del tropismo virale, pertanto la sequenza V3
accoppiata all’utilizzo di un algoritmo interpretativo per la definizione del
fenotipo, così detto fenotipo virtuale, è il modo più semplice per identificare il
tropismo del virus, tuttavia a causa dell’elevata variabilità del gene env i saggi
genotipici possono essere inaffidabili, specialmente a causa della possibile
presenza di sequenze minoritarie che non vengono evidenziate con i comuni
metodi di sequenziamento. Al contrario, i saggi biologici, specialmente quelli
basati su metodi clonali, sono sempre in grado di valutare in modo completo il
fenotipo della quasi specie virale purchè si possa lavorare su un numero di
cloni adeguato ed elevato.
Questo lavoro si è posto come obiettivo la valutazione del saggio fenotipico
home-brew che si basa sull’utilizzo del clone molecolare replicativo
80
pNLmodΔV3GF e del test genotipico geno2pneno utilizzando come
riferimento il test commerciale Trofile.
Partendo dall’amplificato per la regione V3 possiamo ottenere una library di
plasmidi ricombinanti che riflette la quasispecie presente nel campione, dopo
lo screening e la purificazione, ogni singolo plasmide è utilizzato in un saggio
di trasfezione - infezione che consente di conoscere il fenotipo conferito da
quella particolare V3 e la capacità replicativa della variante.
Poiché i vettori sono anche modificati per l'espressione di una proteina
fluorescente (GFP) sotto il controllo del promotore virale la capacità
replicativa può essere valutata mediante una semplice analisi fluorimetrica.
I nostri risultati hanno evidenziato una quasi perfetta corrispondenza tra i
saggi biologici presi in esame, il saggio ricombinante home-brew e il test
commerciale Esta, in particolare il nostro test ricombinante home brew ha
mostrato una maggior capacità nell’identificazione del coreceptor usage (34
pazienti identificati da Esta, 37 dal saggio home brew). Un risultato altrettanto
incoraggiante è stato osservato nel paragone tra test genotipico e test home
brew, dove si evidenzia una elevata concordanza nella rilevazione dei virus
R5-tropici e una sensibilità pari al 100% per la rilevazione dei virus CXCR4
tropici quando la soglia di FPR utilizzata è del 10% o del 20%.
E’ interessante notare che il 51% delle sequenze V3 clonate identificate come
X4 dall’algoritmo geno2pheno quando inserite nel backbone di NL 4-3
conferiscono alla chimera virale un fenotipo R5, quando l’FPR è del 20%;
questa percentuale scende al 33% se l’FPR stabilito è del 10%, e ancora fino al
81
16% se la soglia stabilita è del 5%; in termini clinici tutto questo significa che
più aumentiamo il valore di soglia FPR e maggiore è il rischio di non
assegnare la terapia con il Maraviroc, un antagonista del CCR5 e pertanto un
inibitore dell’entry per virus con fenotipo R5, a pazienti che potrebbero
giovare del farmaco perché in realtà albergano ceppi virali R5; al contrario
tenere bassa la soglia di FPR significa avere la certezza di identificare un virus
come CXCR4 ma anche rischiare di classificare come CCR5 tropico un virus
che in realtà è CXCR4 tropico, e in questo caso la somministrazione del
Maraviroc favorirebbe la selezione delle possibili varianti X4 presenti.
Attualmente le raccomandazioni europee prevedono un FPR del 10%, ed i
nostri dati concordano con il fatto che questa soglia mantiene un ottimo
compromesso tra specificità e sensibilità.
Nel confronto tra test geno2pheno e test Trofile i risultati sono in linea con
quelli pubblicati (93), inoltre si osserva una netta inferiorità nella specificità e
sensibilità di test Trofile rispetto al nostro saggio ricombinante, risultato in
parte atteso e determinato dalla differente strategia utilizzata nei due saggi
fenotipici, nel nostro saggio viene infatti analizzata ogni singola sequenza per
la sua intrinseca capacità re plicativa e la valutazione del fenotipo virale deriva
dall’analisi globale di più varianti ottenute per ricombinazione, nel saggio
commerciale viene globalmente riprodotta in vitro la quasi specie virale
presente in vivo e talvolta in alcuni casi questo potrebbe comportare una
perdita in sensibilità.
82
Per quanto riguarda la capacità dell’algoritmo interpretativo utilizzato con il
test genotipico per la definizione del coreceptor usage, sia confrontando con
Trofile che con il saggio ricombinante home-brew si conferma che la scelta
migliore rispetto alla soglia da fissare è quella di un FPR al 10%.
Per concludere, la metodica descritta nel nostro studio per l’analisi fenotipica
delle sequenze V3 amplificate dai pazienti presi in esami, si è rivelata versatile
ed affidabile, e dopo l’ottimizzazione del processo, di esecuzione
relativamente semplice.
83
BIBLIOGRAFIA
1)
Barre-Sinousi F, J.C. Chermann, F. Rey, et al. 1983. Isolation of a Tlymphotropic retrovirus from a patient at risk for acquired immunodeficiency
sindrome (AIDS). Science 220: 868-871.
2)
Gallo R.C, S.Z. Salahuddin, M. Popovic, et al. 1984. Frequent detection
and isolation of cytophatic retroviruses (HTLV III) from patients with AIDS.
Science 224: 500-503.
3)
Blattner, W.A. 1991. HIV epidemiology: past, present, and future.
FASEB J. 5: 2340-2348.
4)
Quinn T.C. 1994. Population migration and the spread of types 1 and 2
human immunodeficiency viruses. Natl. Acad. Sci. USA. 91: 2407-2414.
5)
Myers G, and G.N. Pavlakis. 1992. Evolutionary potential of complex
retroviruses. In: Levy J.A., ed. The retroviridae. New York: Plenum. Press.:
51-105.
6)
Myers G. 1994. HIV: between past and future. AIDS Res. Hum.
Retroviruses. 10: 1317-1324.
7)
Gelderblom H.R.1991.Assembly and morphology of HIV: potential effect
of structure on viral function. AIDS. 5: 617-638.
8)
Ozel M, G. Pauli, H.R. Gelderblom. 1988.The organization of the
envelope projections on the surface of HIV. Arch. Virol. 100: 255-266.
84
9)
Henderson LE, Krutzsch HC, Oroszlan S. 1983. Myristyl aminoterminal
acylation of murine retrovirus proteins: an unusual post-traslational protein
modification. Proc. Natl. Acad. Sci USA. 80: 339-343.
10)
Yu O.C, Z. Matsuda, X. Yu, S. Ito, M. Essex, and T.H. Lee. 1994. An
electrol-lucent region within the virion distinguishes HIV-1 from HIV-2 and
simian immunodeficiency viruses. AIDS Res. Hum. Retroviruses. 10: 757761.
11)
Marquet R, F. Baudin, C. Gabus, J.L. Darlix, M. Mougel, C.
Ehresman,
and
B.
Ehresman.
1991.
Dimerization
of
human
immunodeficiency virus (type 1) RNA: stimulation by cations and possible
mechanism. Nucleic. Acids. Res. 19: 2349-2357.
12)
Jones K.A. and B.M. Peterlin. 1994. Control of RNA iniziation and
elongation at the HIV-1 promoter. Annu. Rev. Biochem. 63: 717-743.
13)
Cherrington J. and D. Gamen. 1992. Regulation of polyadenylation in
human immunodeficiency virus (HIV): contributions of promoter proximity
and upstream sequences. EMBO J. 11: 1513-1524.
14)
Myers G, B. Korber, S. Wain-Hohson, K.T. Jeang, L.E. Henderson,
and G.N. Pavlakis. 1994. Human retroviruses and AIDS. A compilation and
analysis of nucleic acid and amino acid sequences. Los Alamos, NM: Alamos
National Laboratory.
15)
Arya S.K, C. Guo, SF. Jsephs, F. Wong-Staal. 1985. Transactivator gene
of human T-lymphotropic virus type III (HTLV-III). Science. 229: 69-73.
85
16)
Hauber J, A. Perkins, E.P. Heimer, and B.R. Cullen. 1987. Transactivation of human immunodeficiency virus gene expression is mediated by
nuclear events. Proc. Natl. Acad. Sci. USA. 84: 6364-6368.
17)
Parslow T.G. 1993. Post-transcriptional regulation of human retroviral
gene expression. In: Cullen B.R., ed. Human retroviruses. Oxford: IRL Press:
101-136.
18)
Garcia JV, Miller AD. 1991. Serine phosphorylation-indipendent
downregolation of cell-surface CD4 by nef. Nature. 350: 508-511.
19)
Rizzuto, C.D, R. Wyatt, N. Hernandez-Ramos, Y. Sun, P.D. Kwong,
W.A. Hendrickson, and J. Sodroski. 1998. A conserved gp120 glycoprotein
structure involved chemokine receptor binding site. Science. 280: 1949-1953.
20)
Peter D. Kwong, Richard Wyatt, James Robinson, Raymond W.
Sweet, Joseph Sodroski, Wayne A. Hendrickson. 1998. Structure of an HIV
gp120
envelope glycoprotein in complex with the CD4 receptor and a
neutralizing human antibody. Nature. 393: 648-659.
21)
Lee W-R, W-J Syu, B. Du, M. Maatsuda, S. Tan, A. Wolf, M. Essex, TH Lee. 1993. Norandom distribution of gp 120 N-linked glycosylation sites
important for infectivity of human immunodeficiency virus type 1. Proc. Natl.
Acad. Sci. USA. 89: 2213-2217.
22)
Sophia Rits-Volloch, Gary Frey, Stephen C. Harrison and Bing Chen.
2006. Restraining the conformation of HIV-1 gp120 by removing a flexible
loop. The EMBO Journal. 25: 5026-5035.
86
23)
Freed EO, Myers DJ. 1990. Characterization of the fusion and processing
domain of the human immunodeficiency virus type 1 envelope glycoprotein
gp41. Proc. Natl. Acad. Sci. USA. 87: 4650-4654.
24)
Nelson L. Michael. 1999. Host genetic influences on Hiv-1 pathogenesis.
Current Opinion in Immunology. 11:466-474.
25)
Leigh-Brown
A.J.
1991.
Sequence
variability
of
human
immunodeficiency viruses: pattern and process in viral evolution. AIDS. 5S:
35-42.
26)
Bakhanashvili M. And A. Hizi. 1992. Fidelity of reverse transcriptase of
human immunodeficiency type 2. FEBS Lett. 306: 151-156.
27)
Jensen MA, van’t Wout AB. 2003. Predicting HIV-1 coreceptor usage
with sequence analysis. AIDS Rev. 5: 104-112.
28)
Teruaki Watabe, Hirohisa Kishino, Yoshiyasu Okuhara and Yasuhiro
Kitazoe. 2006. Fold recognition of the Human Immunodeficiency Virus Type
1 V3 Loop and Flexibility of its Crown Structure During the Course of
Adaptation to a Host. Genetics. 172: 1385-1396.
29)
Yamaguchi Y, and T. Gojobori. 1997. Evolutionary mechanisms and
population dynamics of the third variable envelope region of HIV within
single hosts. Proc. Natl. Acad. Sci. USA. 94: 1264-1269.
30)
Moore J.P. and P.L. Nara. 1991.The role of the V3 loop of gp 120 in
HIV infection. AIDS. 5 (suppl. 2): S21-S33.
31)
Moore J.P. and J. Binley. 1998. Envelope’s letters boxed into shape.
Nature 393: 630-631.
87
32)
Grimalia R.J, B.A. Fuller, P.D. Rennert, M.B. Nelson, M.L.
Hammarskjold,
B.
Potts,
M.
Murray,
S.D.
Putney,
G.
Gray.
1992.Mutations in the principal neutralization domain of the human
immunodeficiency virus type 1 affect syncytium formation, virus infectivity,
growth kinetics, and neutralization. J. Virol. 66: 1875-1883.
33)
Page K.A, S.M. Stearns, D.R. Littman. 1992.Analysis of mutations in
the V3 domain of gp 160 that affect fusion and infectivity. J. Virol.. 66: 524533.
34)
Fouchier R.A.M, M. Groenik, N.A. Koostra, M. Tersmette, H.G.
Huisman, F. Miedema, and H. Schuitemaker. 1992. Phenotype associated
sequence
variation
in
the
third
variable
domain
of
the
human
immunodeficiency virus type 1 gp120 molecule.J. Virol. 66: 3183-3187.
35)
Shioda T, J.A. Levy, Cheng C. Mayer. 1992. Small amino acid changes
in the V3 hypervariable region of gp 120 can affect the T-cell-line and
macrophage tropism of human immunodeficiency virus type 1. Proc. Natl.
Acad. Sci. USA. 89: 9434-9438.
36)
Westervelt P, D.B Trowbridge, L.G. Epstein, Y. Li, B.H. Hahn, G.M.
Shaw, R.W. Price, L. Ratner. 1992. Macrophage tropism determinants of
human immunodeficiency virus type 1 in vivo. J. Virol. 66: 2577-2582.
37)
Ebenbichler C, P. Westervelt, A. Carillo, T. Henkel, D. Johnson, L.
Ratner. 1993. Structure-function relationships of the HIV-1 envelope V3 loop
tropism determinant: evidence for two distinct conformations. AIDS. 7: 639646.
88
38)
Willey R.L, Martin M.A. 1993. Association of human immunodeficiency
virus type 1 evelope glycoprotein with particles depends on interactions
between the third variable and conserved regions of gp 120, J. Virol. 67: 36393643.
39)
McNearney T, Z. Hornickowa, R. Markham, A. Birdwell, M. Arens,
A. Saah, and L. Ratner. 1992. Relationship of human immunodeficiency
virus type 1 sequence heterogeneity to stage of disease. Proc. Natl. Acad. Sci.
USA. 89: 10247-10251.
40)
Zhu T, H. M.O, N. Wang, D.S. Nam, Y. Cao, R.A. Koup, and D.D. Ho.
1993. Genotypic and phenotypic characterization of HIV-1 in patients with
primary infection. Science. 261: 1179-1181.
41)
Weiss S, B. Konig, H-J. Muller, H. seidel, and R.S. Goody. 1992.
Synthetic human tRNAlys3 and natural bovine tRNAlys3 interact with HIV-1
reverse transcriptase and serve as specific primers for retroviral cDNA
Synthesis. Gene. 111: 183-197.
42)
Capon D.J. and R.H.R. Ward. 1991. The CD4-gp120 interaction and
AIDS pathogenesis. Annu. Rev. Immunol. 9: 649-678.
43)
Dianzani F, G. Antonelli, M.R. Capobianchi e A. Dolei. 1997.
Retrovirus umani. Manuale Di Virologia Medica. 139-154.
44)
Paolo Lusso. 2006. HIV and the chemokine system: 10 years later. The
EMBO Journal. 25: 447-456.
45)
Horuk Richard. 1998. Chemiokines beyond inflammation. Nature 393:
524-525.
89
46)
Smidmayerova Helena, Barbara Sherry and Michael Bukrinsky. 1996.
Chemokines and HIV replication. Nature 382: 767.
47)
Cocchi F, De Vico AL, Garzino-Demo A, Arya SK, Gallo RC, Lusso P.
1995. Identification of RANTES, MIP-1α, and MIP-1β as the major HIVsuppressive factors produced by CD8+ T cells. Science. 270: 1811-1815.
48)
Horuk Richard. 1999. Chemokine receptors and HIV-1: the fusion of two
major research fields. Immunology Today 20: 89-94. (7-8-9-11-12-15-16-17).
49)
Alkhatib Ghalib, Fang Liao, Edward A. Berger and Joshua M. Farber
1997. A new SIV co-receptor, STRL33. Nature. 388: 238.
50)
Dittmar T. Matthias, Aine Mcknight, Graham Simmons, Paul R.
Clapham and Robin A. Weiss. 1997.HIV-1 tropism and co-receptor use.
Nature 385: 495-496.
51)
Ugolini Sophie, Isabelle Mondor and Quentin J. Sattentau. 1999. HIV1 attachement: another look.Trends. Microbiology 7: 144-149.
52)
Wyatt R, P.D. Kwong, E. Desjardins, R.W. Sweet, J. Robinson, W.A.
Hendrickson and J.G. Sodroski .1998. The antigenic structure of the HIV gp
120 envelope glycoprotein. Nature. 393: 705-711.
53)
Oliver Sander, Tobias Sing, Ingolf Sommer, Andrew J. Low, Peter K.
Cheung, P. Richard Harrigan, Thomas Lengauer, Francisco S.
Domingues. 2007. Structural Descriptors of gp120 V3 Loop for the Prediction
of HIV-1 Coreceptor Usage. PLos Comput Biol. 3(3): e58.
54)
Cristina Pastore, Rebecca Nedellec, Alejandra Ramos, Oliver Hartley,
John L. Miamidian et al. 2007. Conserved Changes in Envelope Function
90
During Human Immunodeficiency Virus Tipe 1 Coreceptor Switching. J.
Virol. 8165-8179.
55)
Butini L, AR. De Fougerolles, M. Vaccarezza, Graziosi, D.I. Cohen, M.
Montroni, T.A. Springer, G. Pantaleo, A.S. Fauci. 1994.C Intercellular
adhesion molecules ICAM-1 ICAM-2 and ICAM-Function
receptors for lymphocyte
function-associated
as
counter-
molecule 1 in human
immunodeficiency virus-mediated syncytia formation. Eur. J. Immunol. 24:
2191-2195.
56)
Baltimore D. 1970. RNA-dependent DNA polymerase in virions of RNA
tumour viruses. Nature. 226: 1209-1211.
57)
Bukrinsky M.I, N. Sharova, T.L. McDonald, T. Pushkarskaya, WG.
Tarpley, and M. Stevenson. 1993. Association of integrase, matrix, and
reverse transcriptase antigens of human immunodeficiency virus type 1 with
viral nucleic acids following acute infection. Proc. Natl. Acad. Sci USA. 90:
6125-6129.
58)
Gallay P, S. Swingler, C. Aiken, and D. Trono. 1995. HIV-1 infection of
nondiving cells: C-terminal tyrosine phosphorylation of the viral matrix
protein is a key regulator. Cell. 80: 379-388.
59)
Lewis P.F, and M. Emerman. 1994. Passage through mitosis is required
for oncoretroviruses by not for the human immunodeficiency virus. J. Virol.
68:510-516.
91
60)
Stevens S.W, and J.D. Griffith. 1994. Human immunodeficiency virus
type 1 may preferentially integrate into chromatin occupied by L1Hs ripetitive
elements. Proc. Natl. Acad. Sci. USA. 91: 5557-5561.
61)
Anderson R.M. and R.M. May. 1998. Epidemiological parameters of
HIV transmission. Nature. 313: 514-51.
62)
Schreiber GB, Busch MP, et al. 1996. The risk of transfusion-transmitted
viral infections. The Retrovirus Epidemiology Donor Study. N. Engl. J. Med.
334(26): 1685-1690.
63)
Ho D.D, R.E. Byington, R.T. Schooley, K. Flynn, T.R. Rota, and M.S.
Hirsch. 1985. Infrequency of isolation of HTLV-III virus from saliva in
AIDS. N. Engl. J. Med. 313: 1606.
64)
Skolnik P, B. Kosloff, and L.J. Bechtel et al. 1989. Absence of infectious
HIV-1 in the urine of seropositive viremic subjects. J. Infect. Dis. 160: 10561060.
65)
Groopman J.E, P.M. Benz, R. Ferriani, K. Mayer, J.D. Allen and L.A.
Weymouth. 1987. Characterization of serum neutralization response to the
human immunodeficiency virus (HIV). AIDS Res. Hum. Retroviruses. 3: 7185.
66)
Allain J.P, Y. Laurian and D.A. Paul et al. 1987. Long term evaluation
of HIV antigen and antibodies to p24 and gp41 in patients with hemophiliapotential clinical importance. N. Engl. Med. 317: 1114-1121.
67)
De Wolf F, J.M.A. Lange and J.T.M. Houweling et al. 1998. Numbers
of CD4+ cells and the levels of core antigens of and antibodies to the human
92
immunodeficiency virus as predictors of AIDS among seropositive
homosexual men. J. Infect. Dis. 158: 615-622.
68)
Rusche J.R, K. Javahetian, and C. McDanal et al. 1988. Antibodies that
inhibit fusion of human immunodeficiency virus-infected cells bind a 24amino acid sequence of the viral envelope, gp120. Proc. Natl. Acad. Sci. USA.
85: 3198-3202.
69)
Albert J, H. Gaines, A. Sonnerborg, G. Nystrom, P. O. Pehrson, F.
Chiodi, M. von Sydow, L. Morberg, K. Lydman, B. Christensson, B. Asyo,
and E. M. Fenyo. 1987. Isolation of human immunodeficiency virus (HIV)
from plasma during primary HIV infection. J. Med. Virol. 23: 67-73.
70)
Fauci A.S. 1993. Multifactorial nature of human immunodeficiency virus
disease: implications for therapy. Science. 262: 1011-1018.
71)
Greene W.C. 1991. The molecular biology of human immunodeficiency
virus type 1 infection. N. Engl. J. Med. 324: 308-317.
72)
Fauci A.S. 1988. The human immunodeficiency virus: infectivity and
mechanism of pathogenesis. Science. 239: 617-622.
73)
Piatak M, M.S. Saag, L.C. Yang, S.J. Clark, J.C. Kappes, K.C. Luk,
B.H. Hahn, G.M. Shaw, and J.D. Lifson. 1993 High levels of HIV-1 in
plasma during all stages of infection determined by competitive PCR. Science.
259: 1749-1754.
74)
Wei X, S. K. Ghosh, M.E. Taylor, V.A. Johnson, E.A. Emini, P.
Deutsch, J.D. Lifson, S. Bonhoefer, M.A. Nowak, B.H. Hahn, M.S. Saag,
93
and G.M. Shaw. 1995. Viral dynamic in human immunodeficiency virus type
1 infection. Nature (London) 373: 117-122.
75)
Clementi M, P. Bagnarelli, S. Menzo, A. Valenza, A. Manzin, and P.E.
Varaldo. 1993. Clerance of HIV-1 viremia after seroconversion. Lancet. 341:
315-316.
76)
Bagnarelli P, A. Valenza, S. Menzo,
A. Manzin, G. Scalise, P.E.
Varaldo, and M. Clementi. 1994. Dynamics of molecular parameters of
human immunodeficiency virus type 1 activity in vivo. J. Virol. 68: 24952501.
77)
Fauci A.S, S.M. Schnittman, G. Poli, S. Koenig, and G. Pantaleo. 1991.
Immunopathogenic mechanism in human immunodeficiency virus (HIV)
infection. Ann. Intern. Med. 114: 678-693.
78)
Ho D.D, A.U. Neuman, A.S. Perelson, W. Chen, J.M. Leonard, and M.
Markowitz. 1995. Rapid turnover of plasma virions and CD4 lymphocytes in
HIV-1 infection. Nature (London). 373: 123-126.
79)
Buchbinder S. P, M. H. Katz, N. A. Hessol, P. M. O’Malley and S. D.
Holmberg. 1994. Long term HIV-1 infection without immunologic
progression. AIDS. 8: 1123-1128.
80)
Lifson A.R, S.P. Buchbinder, H.W. Sheppard, A.C. Mawle, J. Wilber,
M. Stanley, C.E. Hart, N.A. Hessol, and D. Holmberg. 1991. Long term
human immunodeficiency virus infection in asymptomatic homosexual men
with
normal CD4+
lymphoyte
counts: immunologic
characteristics. J. Infect. Dis. 163: 959-965.
94
and
virologic
81)
Hill C.Mark and Dan R Littman. 1996. Natural resistence to HIV?
Nature 382: 668-669.
82)
Richman D.D. 2001. HIV chemotherapy. Nature. 410: 995-1001.
83)
Ahmad R. Sedaghat, Robert F. Siliciano and Claus O. Wilke. 2008.
Low-Level HIV-1 Replication and the Dynamics of the Resting CD4+T cell
Reservoir for HIV-1 in the setting of HAART. BMC Infectious Diseases. 8: 2.
84)
Richmann DD, Morton SC et al. 1987. The toxicity of azidothymidine
(AZT) in the treatment of patients with AIDS and AIDS-related complex. A
double-blind, placebo-controlled trial. N Engl J Med. 317 (4):192-197.
85)
Cohen J. Therapies. 2002. Confronting the limits of success. Science.
296: 2320-2324.
86)
Hirsch M.S. et al. 2000. Antiretroviral drug resistance testing in adults
HIV-1 infection. Recommendations of an international AIDS Society-USA
Panel. JAMA. 283: 2417-2426.
87)
Hertogs K et al. 2000. Phenotypic and genotypic analysis of clinical HIV1 isolates reveals extensive protease inhibitors cross-resistance: a survey of
over 6000 samples. AIDS. 14: 1203-1210.
88)
Fatkenheuer G:, Pozniak AL, Johnson MA, et al. 2005. Efficacy of
short-term monotherapy with maraviroc, a new CCR5 antagonist, in patients
infected with HIV-1. Nat Med; 11:1170-72.
89)
Watson C, Jenkinson S, Kazmierski W, Kenakin T. 2005. The CCR5
receptor-based mechanism of action of 873140, a potent allosteric
noncompetitive HIV entry inhibitor. Mol Pharmacol; 67:1268-82
95
90)
Schols D et al. Hiv co-receptor inhibitors as novel class of anti-HIV drugs.
Antiviral res. 2006; 71: 216-26.
91)
Kilby JM, Eron JJ. Novel therapies based on mechanisms of HIV-1 cell
entry. N Engl J Med 2003; 2003 348:2228-38
92)
Dorr P, Westby M, Dobbs S, et al. Maraviroc (UK-427,857), a potent,
orally bioavailable, and selective small-molecule inhibitor of chemokine
receptor CCR5 with broad-spectrum anti-human immunodeficiency virus type
1 activity. Antimicrob. Agents Chemother. 2005; 49:4721-32.
93)
Svicher V. Et al. Performance of genotypic tropism testing in clinical
practice using the enhanced sensitivity version of Trofile as reference
assay:results from the OSCAR Study Group. 2010; 33:195-206
94)
Bagnarelli P., F.Mazzola, S.Menzo, M.Montoni, L.Butini and
M.Clementi. 1999. Host-Specific Modulation of the Selective Constraints
Driving Human Immunodeficiency Virus Type 1 env Gene Evolution. J.Virol.
73:3764-3777.
95)
Canducci F., M.C.Marinozzi, M.Sampaolo, S.Berrè, P.Bagnarelli,
M.Degano, G.Gallotta, B.Mazzi, R.Burioni and M.Clementi. High
selective pressure to the gp120 CD4 binding-site of human immunodeficiency
virus type 1 (HIV-1) characterizing the replicating virus in samples from long
term non-progressor subjects. (Articolo sottomesso per la pubblicazione).
96)
Carrington M., and S.J.O’Brien. 2003. The influence of HLA genotype
on AIDS. Annu. Rev. Med. 54:535-51.
96
97)
Bagnarelli P., L.Fiorelli, M.Vecchi, A.Monachetti, S.Menzo and
M.Clementi. 2003. Analisys of the functional relationship between V3 loop
and gp120 context with regard to human immunodeficiency virus coreceptor
usage using naturally selected sequences and different viral backbones.
Virology. 307:328-340.
98)
Yang Z. 1997. PAML: a program package for phylogenetic analysis by
maximum likelihood. Computer Applications in BioSciences. 13:555-556.
99)
Bagnarelli P., M.Vecchi, N.Burighel, D.Bellanova, S.Menzo, M.
Clementi and A.De Rossi. 2004. Genotypic and phenotypic correlates of the
HIV type 1 cnv gene evolution in infected children with discordant response to
antiretroviral therapy. AIDS research and Human Retoviruses. Vol.20
n°12:1306-1313.
100)
Nowak M.A. and C.R.Bangham. 1996. Population dynamics of immune
responses to persistent viruses. Science. 272:74-79.
101)
Regoes R.R., D.Wodarz and M.A.Nowak. 1998. Virus dynamics: the
effect of target cell limitation and immune responses on virus evolution. J.
Theor. Biol. 191:451-462.
102)
Quinones-Mateu M.E., S.C. Ball, A.J. Marozsan, V.S. Torre, J.L.
Albright, G. Vanham, G. van DER Groen, R.L. Colebunders and
E.J.Arts. 2000. A dual infection/competition assay shows a correlation
between ex vivo human immunodeficiency virus type 1 fitness and disease
progression. J.Virol. 74:9222-9233.
97
103)
Menzo S., R.Sampaolesi, E.Vincenzi, E.Santagostino, G.Liuzzi,
A.Chirianni, M.Piazza, O.J.Cohen, P.Bagnarelli and M.Clementi. 1998.
Rare mutations in a domain crucial for V3 loop structure prevail in replicating
HIV from long-term non progressors. AIDS. 12:985-997.
98