LA NATURA DELLE MUTAZIONI
La Bibbia (Genesi) indicava che la creazione era avvenuta in 6 giorni, circa 6000 anni prima. Presa
alla lettera, questa nozione aveva generato la teoria del Fissismo, contrapposto all’Evoluzionismo, che
proponeva un evento creativo indipendente per ogni specie.
Jean Baptiste Lamarck, nel 1809, nella Philosophie zoologique, introduce il concetto di evoluzione
della specie. Per Lamarck, “fattori” dell’evoluzione sono:
1. tendenza al perfezionamento, insita e innata;
2. ereditarietà dei caratteri acquisiti.
Charles Darwin, nel 1859, pubblica Origin of Species. Per Darwin, “fattori” dell’evoluzione sono:
1. mutazione, casuale (errori della DNA polimerasi, agenti mutageni);
2. selezione naturale;
3. deriva à «survival of the fittest» (Huxley).
CONCETTI BASE DI STATISTICA
Il valore medio
Nel caso di una variabile casuale discreta X (ad esempio, la statura), i cui valori possibili sono x1,
x2, …, xn (la statura dei singoli soggetti), il valore medio (μ) di X è definito, quando le probabilità sono
tutte uguali, da:
μ = (x1 + x2 + … + xn) / n
che viene definito la media aritmetica di X
La varianza
La varianza è una misura della dispersione dei valori della variabile casuale attorno alla media μ:
· valori concentrati vicino alla media
à
varianza piccola
· valori dispersi lontano dalla media
à
varianza grande
Nel caso in cui tutte le probabilità siano uguali:
Var(X) = σ2 = [(x1 – μ)2 + (x2 – μ)2 + … + (xn – μ)2] / n
La deviazione standard
La deviazione standard è la radice quadrata della varianza: s = Var ( X )
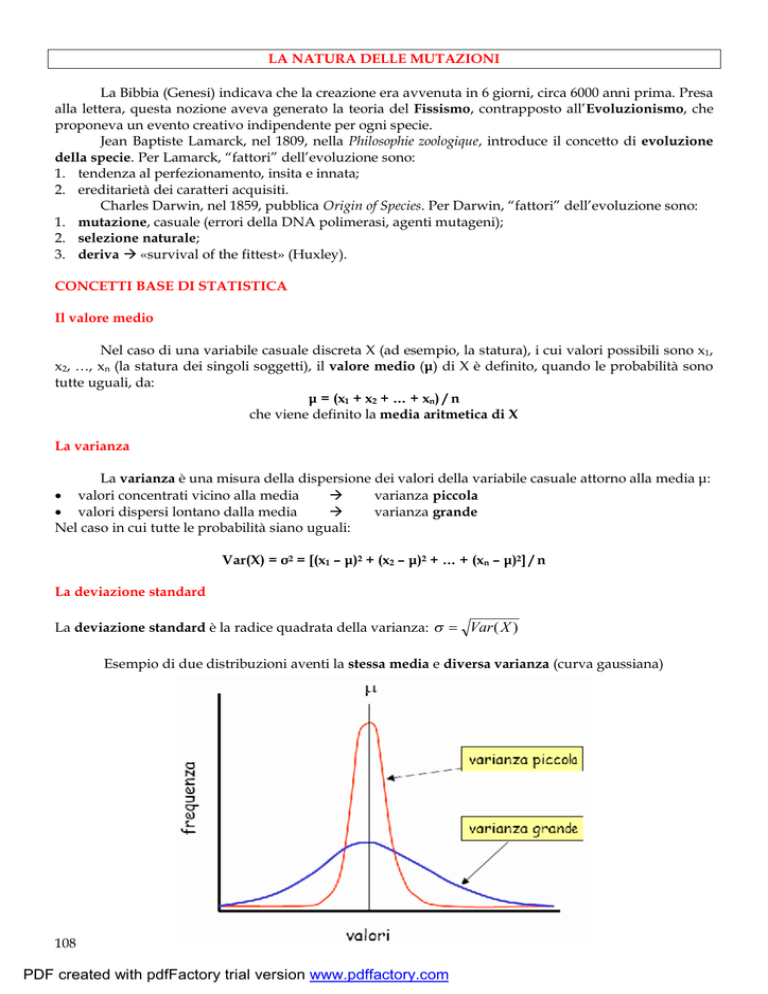
Esempio di due distribuzioni aventi la stessa media e diversa varianza (curva gaussiana)
108
PDF created with pdfFactory trial version www.pdffactory.com
In una distribuzione normale:
· il 68,27% dei dati rientra nella zona μ + σ
· il 95,45% dei dati rientra nella zona μ + 2σ
· il 99,73% dei dati rientra nella zona μ + 3σ
Il rapporto σ / μ è una misura della
fluttuazione attorno alla media.
Natura della mutazione genetica: teoria adattativa vs. teoria selettiva
Osservazione: seminando 108 batteri su piastre contenenti 1010 batteriofagi T1, si trovano circa 10 colonie
batteriche per piastra, che ovviamente derivano da batteri resistenti all’infezione (TonR).
Questa osservazione trova due possibili spiegazioni:
1. teoria adattativa o induttiva (lamarckiana): probabilità di uno su dieci milioni che la mutazione
venga “indotta” dal contatto con l’agente antibatterico;
2. teoria preadattativa o selettiva (darwiniana): preesistenza nella popolazione, prima del contatto con
l’agente antibatterico, di rari mutanti resistenti.
Si prevede che le due ipotesi diano risultati diversi per quanto riguarda la distribuzione
numerica dei fenotipi TonR nelle colture batteriche:
Ipotesi adattativa
a=n/N
Ipotesi preadattativa
n / N = g * a * 2g / 2g = g * a
a: probabilità di conversione da TonS a TonR
n: numero di batteri TonR osservati
N: numero di batteri piastrati
a: tasso di mutazione da TonS a TonR
g: numero di generazioni trascorse per passare da 1 a N
batteri
N: 2g raddoppi, di cui
n: g * a * 2g producono batteri mutanti TonR
¯
n deve aumentare con l’aumentare di g
¯
n deve essere una frazione costante di N
Esperimento di Luria (test di fluttuazione)
L’esperimento prevede di far crescere O/N cellule batteriche in
una coltura di grandi dimensioni ed in numerose subcolture
molto piccole, inoculando i batteri ad una concentrazione di 5x103
cellule/ml. Il giorno dopo, un’aliquota di tutte le colture viene
esposta al batteriofago T1 e si valuta la presenza di colonie
resistenti al fago.
Crescita overnight (O/N) a 37°C fino a 5 x 108 batteri/ml
109
PDF created with pdfFactory trial version www.pdffactory.com
Piastre provenienti dalle 10 colture indipendenti
Mutanti TonR: totale 210; frequenza 2,1 x 10-7; μ = 21; σ = 43,9
Piastre provenienti dalle 10 repliche della singola coltura
Mutanti TonR: totale 212; frequenza 2,1 x 10-7; μ = 21,2; σ = 6,2
La fluttuazione può essere calcolata come
F=σ/μ
dove σ è la deviazione standard e μ è la media
Colture indipendenti
Repliche
à
à
F = 2,09
F = 0,29
Secondo la teoria adattativa, F dovrebbe essere uguale in entrambi i casi, dal momento che il
numero di mutanti dipende solo dall’esposizione all’agente selettivo. Poiché il fago T1 viene aggiunto a
tutte le colture nello stesso momento, ci si aspetta Find = Frepliche e Fmutanti = Tmutazione.
Secondo la teoria preadattativa o selettiva, F dovrebbe essere maggiore nelle colture
indipendenti, dal momento che il numero di mutanti che si ottengono dipende dal momento in cui la
mutazione è comparsa e dal fatto che le dimensioni dei cloni resistenti saranno tanto maggiori quanto
più tempo è passato dalla comparsa della mutazione. Questo può verificarsi in maniera diversa per
ciascuna subcoltura. Ci si aspetta quindi che Find >> Frepliche e Fmutanti >> Tmutazione.
110
PDF created with pdfFactory trial version www.pdffactory.com
L’equazione di Poisson permette di risalire al tasso di mutazione (a) conoscendo il numero di
colture che non hanno mutanti.
P0 = e-aN
·
·
P0: numero di piastre senza mutanti diviso per il totale delle piastre
N: numero di batteri seminati in ciascuna piastra
Luria e Delbruck (1943), secondo la distribuzione di Poisson, ricavano che:
a = (-lnP0) / N
a = -ln0,4 / 108 = 0,916 / 108 = 9,16 x 10-9
Quindi, il tasso di mutazione è 9,16 x 10-9
Secondo la teoria adattativa, la frequenza di mutanti osservati (F), dovrebbe essere pari alla Tm. Invece:
F = 2,1 x 10-7 >> 9, 16 x 10-9
Da ogni mutazione, quindi, sono derivati in media 23 mutanti.
Pertanto, gli esperimenti di Luria e Delbruck dimostrano che la mutazione è preadattativa.
MUTAZIONE DI UN GENE
Non tutte le mutazioni causano un’alterazione delle proteine e non tutte le mutazioni avvengono
nella regione codificante del gene.
Rappresentazione di una popolazione in divisione di E. coli selvatico, sensibile al fago T1: alla quarta
generazione viene aggiunto il fago T1:
a. se la teoria dell’adattamento fosse corretta, le cellule muterebbero solo con l’aggiunta del fago T1,
quindi la frazione di cellule resistenti nelle colture duplicate sarebbe la stessa;
b. se fosse corretta la teoria della mutazione casuale, le cellule muterebbero indipendentemente dalla
presenza del fago T1 e quindi la frazioni di cellule resistenti sarebbe diversa nelle colture duplicate.
A sinistra: se una cellula muta diventando resistente al fago T1 nella terza generazione, 2 cellule su 16
della quarta generazione saranno resistenti al fago. A destra: se una cellula diventa resistente al fago
T1 nella prima generazione, 8 cellule su 16 della quarta generazione saranno resistenti a T1.
111
PDF created with pdfFactory trial version www.pdffactory.com
Tipi di mutazione per sostituzione di una coppia di basi
La trascrizione del segmento mostrato produce un mRNA
UCUCAAAAAUUUACG… 3’ che codifica per …- Ser – Gln – Lys – Phe – Thr -…
112
PDF created with pdfFactory trial version www.pdffactory.com
con
sequenza
5’
Mutazione nonsenso e sue conseguenze sulla traduzione
Meccanismo di azione di un soppressore intergenico di mutazione nonsenso, dovuto a mutazione in
un gene per un tRNA.
In questo esempio, un gene per un Tyr.tRNA, mutato in modo che l’anticodone del tRNA è
cambiato da 59-GUA-39 a 59-CUA-39, può leggere un codone nonsenso UAG, inserendo tirosina nella
posizione corrispondente del polipeptide.
113
PDF created with pdfFactory trial version www.pdffactory.com
Appaiamenti corretti e non corretti delle basi del DNA
Appaiamento normale secondo Watson e Crick
Appaiamento vacillante pirimidina-purina: appaiamenti non corretti tra T e G e C e A
Appaiamento vacillante purina-purina (A e G) e pirimidina-pirimidina (T e G)
Produzione di una mutazione causata da un appaiamento errato
114
PDF created with pdfFactory trial version www.pdffactory.com
Origine spontanea di mutazioni per inserzione o delezione in seguito a ripiegamento del DNA
durante la replicazione
Deaminazione della citosina ad uracile
Deaminazione della 5-metil-citosina (5mC) a timina
115
PDF created with pdfFactory trial version www.pdffactory.com
Effetti mutageni dell’analogo delle basi 5-bromouracile (5BU)
a. Nel suo stato normale, il 5BU si appaia con l’adenina.
b. Nel suo stato raro, il 5BU (indicato da 5BU evidenziato) si appaia con la guanina.
c. I due possibili meccanismi di mutazione. Il 5BU induce mutazioni per transizione quando viene
incorporato nel DNA in uno stato e poi passa alla forma alternativa durante il successivo ciclo di
replicazione del DNA.
116
PDF created with pdfFactory trial version www.pdffactory.com
Azione di tre agenti modificatori di base
a. L’acido nitroso (HNO2) modifica:
1. la guanina
à
nessuna mutazione;
2. la citosina
à
mutazione;
3. l’adenina
à
mutazione.
b. L’idrossilamina (NH2OH) reagisce solo con la citosina.
c. Il metilmetansolfuro (MMS), un agente alchilante, alchila la guanina.
117
PDF created with pdfFactory trial version www.pdffactory.com
Mutazioni per intercalazione
Mutazione frameshit per inserzione,
causata dall’inserimento dell’agente
intercalante nell’elica stampo.
Mutazione frameshit per delezione,
causata dall’inserimento dell’agente
intercalante nell’elica di neosintesi.
Test di Ames per verificare la potenziale mutagenicità di composti chimici
118
PDF created with pdfFactory trial version www.pdffactory.com
Riparazione per excisione dei nucleotidi (NER) dei dimeri di pirimidine e altri danni che causano
distorsioni nel DNA
119
PDF created with pdfFactory trial version www.pdffactory.com
TUMORI
Cause che possono trasformare una cellula normale in una cellula tumorale:
La scoperta degli oncogèni
à
120
PDF created with pdfFactory trial version www.pdffactory.com
à
Mutazioni in cellule tumorali
·
·
·
Due categorie:
1. oncogèni, tipicamente dominanti;
2. geni onco-soppressori mutati, tipicamente
recessivi.
Associate talvolta ad anomalie cromosomiche, ad
es. traslocazioni, che pongono un gene sotto il
controllo di un promotore forte di un altro gene.
Cellule che perdono la capacità di andare incontro
ad apoptosi hanno tempo di accumulare
mutazioni che promuovono la crescita tumorale
incontrollata.
Oncogeni
·
·
·
Proto-oncogeni generalmente codificano proteine che regolano la proliferazione normale delle cellule
o l’apoptosi.
Di norma codificano regolatori positivi o negativi.
Accumulano mutazioni e diventano oncogeni:
· mutazioni puntiformi alterano struttura/funzione;
· perdita di domini in seguito a delezioni;
· fusione di geni, spesso dovute a traslocazioni;
· a volte la mutazione determina espressione della proteina in sedi o in tempi sbagliati.
Apoptosi
·
·
·
·
Morte cellulare programmata (PCD).
Attivata da numerosi segnali.
Distruzione sequenziale della cellula:
· frammentazione dei cromosomi;
· disgregazione degli organelli;
· frammentazione della cellula.
Causata dall’attività delle caspasi:
· cysteine-containing aspartate-specific proteases;
· di norma sono inattive (nessun danno cellulare);
· lo zimogeno viene attivato mediante proteolisi;
· le caspasi attive determinano la distruzione di altre proteine.
121
PDF created with pdfFactory trial version www.pdffactory.com
Fattori di crescita e loro omologhi alterati
Proteine G e loro omologhi alterati
122
PDF created with pdfFactory trial version www.pdffactory.com
Regioni funzionalmente rilevanti di p21ras
Regioni coinvolte nel legame a GDP/GTP
Regioni attivabili da GAP
Regioni importanti per la risposta a
GNRP
Proteinchinasi e loro omologhi alterati
Proteina pp60 src
Meccanismo di spegnimento della pp60 src
123
PDF created with pdfFactory trial version www.pdffactory.com
Geni onco-soppressori
·
·
·
Possono codificare regolatori negativi del ciclo cellulare o regolatori positivi dell’apoptosi.
Retino blastoma causato da un gene RB mutato.
p53 (proteina di 53 kDa):
· ~50% dei tumori presentano una forma mutata;
· la p53 normale è un fattore trascrizionale che viene attivato in risposta a danni del DNA:
· blocca la progressione lungo il ciclo cellulare per permettere il riparo;
· determina la morte per apoptosi di cellule gravemente danneggiate;
· la p53 mutata elimina la risposta apoptotica, permette alle cellule danneggiate di sopravvivere,
accumulando mutazioni.
124
PDF created with pdfFactory trial version www.pdffactory.com
Anti-oncogeni: la p53
La p53 è l’anti-oncogene più importante. La proteina è espressa in tutte le cellule, localizzata nel
nucleo e dotata di diversi siti sensibili alla fosforilazione da parte di ciclina/p34. Ha la capacità di
formare omotetrameri che attivano la trascrizione. La formazione di tetrametri attivi è inibita dal
complesso p34/ciclina per fosforilazione di p53, oppure per interazione con le oncoproteine di SV40 ed
HPV.
125
PDF created with pdfFactory trial version www.pdffactory.com
126
PDF created with pdfFactory trial version www.pdffactory.com
127
PDF created with pdfFactory trial version www.pdffactory.com
128
PDF created with pdfFactory trial version www.pdffactory.com
129
PDF created with pdfFactory trial version www.pdffactory.com
Alcune malattie ereditarie umane e tumori associati a difetti di riparazione del DNA
130
PDF created with pdfFactory trial version www.pdffactory.com
RETROVIRUS ONCOGENI
Il “dogma centrale” prima di Peyton Rous:
Peyton Rous, nel 1929, isola il primo virus oncogeno. Prima i virus
erano definiti solo come “agenti filtrabili”.
* (non della sintesi di RNA RNA-diretta)
131
PDF created with pdfFactory trial version www.pdffactory.com
Dopo aver scoperto che è richiesta una neo-sintesi di DNA (sensibilità agli inibitori FUDR e
CAra) e che l’RNA del virus è prodotto da una sintesi di RNA DNA-diretta (sensibilità all’inibitore
Actinomicina D), Temin propose l’ipotesi del provirus a DNA.
vRNA à vDNA à vRNA
L’effetto degli inibitori della sintesi di DNA sulla trasformazione indicano che all’entrata del
virus nella cellula deve avvenire una sintesi di DNA indipendente dalla sintesi del DNA cellulare (che è
comunque bloccata in assenza di siero).
L’esposizione alla luce non danneggia le cellule, ma blocca la sintesi di virus: quindi il virus ha
un intermediario a DNA che viene sintetizzato all’entrata nella cellula e in totale indipendenza dalla
sintesi di DNA cellulare.
132
PDF created with pdfFactory trial version www.pdffactory.com
L’infezione è resistente agli inibitori della sintesi proteica, quindi l’enzima che sintetizza il DNA
virale non ha bisogno di essere sintetizzato ex novo. Temin (e Baltimore) ipotizzarono che fosse
contenuto nei virioni e associato all’RNA virale.
L’enzima, purificato da Baltimore da
virioni di MLV (murine leukemia virus), fu
chiamato trascrittasi inversa. La trascrittasi
inversa possiede tre attività enzimatiche:
1. DNA polimerasi RNA-diretta;
2. RNasi H (attiva sull’ibrido RNA-DNA);
3. DNA polimerasi DNA-diretta.
Trascrittasi inversa di HIV-1
modello space-filling da dati cristallografici
Dopo la scoperta della trascrittasi inversa,
il “dogma centrale” diventa:
Il gene che conferisce il potere oncogeno a RSV è il gene v-src, che codifica per una tirosina
chinasi. V-src deriva da c-src, un gene normale del genoma del pollo, e contiene mutazioni che lo
rendono non regolabile dai normali meccanismi di controllo.
133
PDF created with pdfFactory trial version www.pdffactory.com
Ibridazione tra v-src (nero) e c-src (rosso): v-src corrisponde
al cDNA di c-src.
Un mutante ts di v-src permette di dimostrare che la
trasformazione è reversibile e dovuta solo a v-src.
HIV
Il genoma dell’HIV è composto da circa 9300 nucleotidi.
134
PDF created with pdfFactory trial version www.pdffactory.com
Ciclo vitale di un retrovirus (HIV)
135
PDF created with pdfFactory trial version www.pdffactory.com
Se i recettori usati dal virus non sono presenti il soggetto è immune (10% della popolazione
europea). Riducendo il colesterolo della membrana si ostacola il processo di fusione.
136
PDF created with pdfFactory trial version www.pdffactory.com
La latenza del virus permette un’infezione cronica, e può essere causata da:
· integrazione del virus nell’eterocromatina;
· mancanza della proteina Tat (antiterminatrice), necessaria per una trascrizione ad alta efficienza.
137
PDF created with pdfFactory trial version www.pdffactory.com
Le 9300 basi dell’RNA di HIV codificano otto geni.
La trascrizione di HIV è regolata da fattori cellulari che
agiscono sull’enhancer e sul promoter del provirus.
138
PDF created with pdfFactory trial version www.pdffactory.com
All’inizio del ciclo vitale del virus, la trascrizione è poco efficiente e viene quasi sempre terminata
dopo circa 90 basi. Il trascritto viene tutto double-spliced e si traducono solo le proteine tat, rev e nef.
Due proteine virali intervengono nella regolazione della trascrizione:
1. la proteina Tat è un anti-terminatore, che legandosi all’RNA stimola il
proseguimento della trascrizione (x 2000);
2. la proteina Rev blocca lo splicing e fa esportare l’RNA nel citoplasma.
139
PDF created with pdfFactory trial version www.pdffactory.com
Dopo l’integrazione nei cromosomi dell’ospite,
HIV entra a far parte di una struttura cromatinica che
reprime la trascrizione.
Il promotore è relativamente libero, ma si
formano treni di nucleosomi sia a monte che a valle.
La
coda
CTD
della
RNApolII può essere forsforilata
controllando
il
tasso
di
trascrizione. Tat si associa a TAR
reclutando Cyclin T1 e la
corrispondente chinasi CDK9, che
fosforila CTD. Viene reclutata
anche p300 (acetyl-transferasi) che
acetila gli istoni e sblocca la
struttura
cromatinica
che
rallentava il progresso della
polimerasi.
Nuclear Speckles è un
comparto nucleare ricco di fattori
per lo splicing. Nuclear Body è un
altro comparto che coordina la
presenza di p300, Cyclin T1 e
PML.
La FRET (fluorescence resonance energy transfer) permette di rivelare quali proteine entrino
fisicamente in contatto, come Tat & Cyc T1 e Cyc T1 & PML.
140
PDF created with pdfFactory trial version www.pdffactory.com
Il trascritto primario di HIV: frame di lettura e segnali sull’RNA.
Oltre al trascritto primario, lo
splicing genera altri 5 mRNA:
· gli mRNA I, IV e VI sono bi-cistronici;
· il V e VI subiscono un doppio splice.
Le poliproteine gag e
gag-pol,
miristilate,
si
ancorano alla membrana. La
proteasi si auto-attiva (
),
e taglia le poliproteine nelle
proteine mature.
La p24 si auto-polimerizza con
simmetrie esagonali a formare un capside
tubulare. Alcune interazioni a simmetria
pentagonale richiudono il capside “a forma
di pera”.
Vista laterale Vista dal basso
141
PDF created with pdfFactory trial version www.pdffactory.com
Motivi per cui non si riesce a trovare una cura per l’AIDS
1. La trascrittasi inversa è un enzima molto infedele e introduce molti errori. In ogni particella, ad ogni
ciclo, vengono introdotti 2 o 3 errori, quindi in ogni paziente ci sono centinaia di varianti del virus.
Ad esempio, se si utilizza l’AZT (desossinucleotide che contiene un gruppo ammidico al posto
dell’ossidrile) per interrompere la sintesi di DNA, dopo poco tempo viene selezionato un virus
resistente e l’AZT non funziona più.
2. Nel caso dell’AIDS, al contrario di tutte le altre infezioni virali, la vaccinazione non è efficace. Le
proteine del virus somigliano ad alcune proteine umane, quindi la reazione dei linfociti killer è poco
efficace.
3. Mentre solitamente la presenza di anticorpi su una particella ne determina la distruzione tramite una
reazione di complemento, questa reazione non avviene con l’HIV perché la gp120 lega un inibitore
del sistema di complemento.
PCR (reazione a catena della polimerasi)
La PCR (Polimerase Chain Reaction), è tecnica che permette di amplificare una specifica
sequenza di DNA milioni di volte in poche ore: si possono eseguire fino a 35 cicli, ad ognuno dei quali la
quantità di DNA raddoppia. In tre ore si compiono 30 cicli. La tecnica è stata inventata da Kary Mullis
nel 1983, e viene applicata a scopo diagnostico, ad esempio per diagnosticare l’infezione da HIV o da un
altro virus. Nel caso dell’HIV si usano come primer delle sequenze di HIV.
In una cellula in divisione, la replicazione del DNA comporta una serie di reazioni mediate da
enzimi, il cui risultato è una copia fedele dell’intero genoma. Gli enzimi prima denaturano la doppia
elica di DNA in eliche singole. Quindi, l’RNA polimerasi sintetizza una breve sequenza di RNA
complementare ad una delle eliche di DNA all’inizio del sito di replicazione. Ad essa si lega la DNA
polimerasi, che produce la sequenza di DNA complementare.
Durante la PCR, l’alta temperatura viene usata per separare il
DNA in eliche singole, e una sequenza sintetica di DNA a singola elica
(20-30 nucleotidi) viene usata come primer. Due differenti sequenze
primer sono usate per delimitare la regione che deve essere amplificata.
Un primer è complementare ad una sequenza di DNA all’inizio della regione bersaglio; l’altro primer è
complementare all’altra sequenza di DNA alla fine della regione bersaglio.
Per eseguire una reazione PCR, una piccola quantità del DNA bersaglio è aggiunta è aggiunta ad
una soluzione contenente:
· DNA polimerasi termostabile, isolata dal batterio Thermus aquaticus, che vive ad alte temperature.
Questo enzima, chiamato Taq DNA polimerasi, rimane attivo nonostante ripetuti riscaldamenti
duranti molti cicli di amplificazione;
· brevi primer di oligonucleotidi;
· i quattro blocchi dei desossinucleotidi di costruzione del DNA;
· il cofattore MgCl2.
La soluzione PCR è fatta passare attraverso
cicli di replicazione che consistono in:
· diversi minuti a 94-96°C, durante i quali il DNA
è denaturato in eliche singole;
· diversi minuti a 50-65°C, durante i quali i
primer ibridano o si fissano (tramite legami
idrogeno) alle loro sequenze complementari su
entrambe le eliche della sequenza bersaglio;
· diversi minuti a 72°C, durante i quali la DNA
polimerasi si lega e produce una sequenza
complementare a partire da ciascun primer.
Man mano che l’amplificazione procede, la
sequenza di DNA tra i primer si duplica ad ogni
ciclo. In trenta di questi cicli, si ha un fattore di amplificazione teorico di un miliardo.
142
PDF created with pdfFactory trial version www.pdffactory.com