
Strategie di mutagenesi
SELEZIONE GENETICA…
seleziono FENOTIPO – determino GENOTIPO
GENETICA INVERSA…
Mutagenesi in vivo
Phage display
creo GENOTIPO – determino FENOTIPO
i.e., traduco una sequenza in una funzione
Mutagenesi in vitro
Mutagenesi In Vitro
•
La mutagenesi in vitro permette di cambiare la sequenza
nucleotidica di un frammento di DNA
•
Le mutazioni possono essere localizzate o generali, casuali o
mirate;
• Analisi di regioni regolative: metodi meno specifici
• Ingegneria proteica: Analisi del contributo di singoli aminoacidi
(o gruppi di aminoacidi), alla struttura e funzione della proteina
target-->metodi specifici
•
Con entrambi i metodi si ottengono mutanti in vitro, senza
selezione fenotipica
Mutagenesi di Sequenze codificanti
Principali metodi di mutagenesi
• MUTAGENESI SITO-DIRETTA
• SCANNING MUTAGENESIS
• MUTAGENESI A SATURAZIONE/CASUALE
• MUTAGENESI PER RICOMBINAZIONE
Mutagenesi Sito-Diretta
•
Ruolo di particulari residui nella struttura, attività catalitica o legame
•
Centinaia di metodi – basati su un principio comune
•
Oligo sintetico con la mutazione appaiato alla regione bersaglio gene wt – primer per
sintesi di DNA in vitro
•
Estensione da parte della DNA polimerasi produce dsDNA mutato
•
Il DNA mutato viene inserito nel gene e si esprime la proteina mutante
Progettazione degli Oligonucleotidi Mutagenici
•
Passaggio critico.
•
•
La sequenza contiene (almeno) 1 mutazione
Può contenere inserzioni, delezioni e sostituzioni con nucleotidi
alterati
•
Lunghezza minima dell’oligo: dipende dai casi
•
•
Mutazioni semplici di una base ~25 nt
Mutazioni complesse possono richiedere oligo >80 nts
Si possono progettare varie caratteristiche per ottimizzare la mutazione:
•Sequenza e composizione in basi
Basi appaiate/non appaiate
Percentuale in GC in varie zone (estremità)
•Temperatura di fusione
•Propensità a formare strutture secondarie
•Specificità di appaiamento
Nel templato
Tra primer (diminuisce la concentrazione critica)
Mutagenesi sito-diretta classica
•Protocollo comprende:
•Progettazione e sintesi degli oligo mutagenici
•Ibridazione degli oligo mutagenici a ssDNA bersaglio (M13) –elimina
competizione tra oligo mutagenico e filamento di DNA complementare
•Estensione dalla DNA polimerasi in presenza di 4 dNTPs
•Formazione di DNA circolare con DNA ligasi
•Trasformazione di batteri
•Screening dei cloni mutati per ibridazione (e.g., oligo) – sequenza
•Recupero del frammento di DNA mutato
•Sostituzione del frammento mutato nel gene wt
1. Clonaggio dell’ inserto nel vettore
2. Denaturazione ed appaiamento
3. Estensione con T4 DNA polimerasi;
Ligazione con T4 DNA ligasi
4. Selezione filamento mutante
Trasformazione
wild-type sequence
Ile
CAA GCG CCC ATT GGC AAA CAA
GTT CGC GGG GAA CCG TTT GTT
mutant oligo
Leu
Mutagenesi sito-diretta classica
Percentuale di cloni mutati 0.1% - 50% a seconda della efficienza
dell’oligo mutagenico
Sviluppo di metodi di selezione negativa del DNA wt;
– KUNKEL: Digestione selettiva del filamento stampo con enzimi di restrizione
o esonucleasi
– USE (Unique Site Elimination) Eliminazione di un sito unico di restrizione
– Associazione con recupero di resistenza o capacità di replicazione
Mutagenesi con Metodo Kunkel
•
Selezione negativa di DNA con uracile in ceppi di E. coli che
esprimono la uracil-DNA glicosidasi
•
Stampo ssDNA ottenuto preparando M13 in dut- ung- F’ E. coli
dut- =difettivi dUTPasi ;
ung- = difettivi
uracil-N-glicosidasi
•La
mutagenesi
genera
un
heteroduplex in cui stampo
con
uracile
ma
filamento
neosintetizzato con timidina
Questo heteroduplex DNA si
trasforma in ceppo ung+ E.
coli – selezione contro il
filamento wt
•Fino ad 80% sono mutanti
Mutagenesi con metodo USE
•
•
Elimina sito di restrizione unico
•
Heteroduplex: lo stampo ha il sito di restrizione , mentre il
filamento nuovo ha la mutazione, ma non il sito di restrizione
•
•
Incubazione con enzima – si linearizza wt ma non heteroduplex
Usa 2 primers – un primer (primer mutagenico) introduce la
mutazione, il secondo elimina il sito unico nel plasmide
Transformazione in ceppo di E. coli che non ripara (BMH71-18
mutS) (DNA lineare transforma 10-1000 volte meno del circolare)
si replica heteroduplex
•
Seconda digestione e transformazione in ceppo di E. coli normale–
(5-50% mutanti)
Mutagenesi a cassetta
•
•
•
•
Sostituzione di un frammento di DNA tra due siti di restrizione
Possibilità di ottenere mutanti multipli
Semplice ed efficiente
Problema: necessità di siti unici (geni sintetici OK)
PCR…
Mutagenesi per PCR
Vantaggi rispetto ai metodi classici;
•
•
•
•
•
Alta percentuale mutanti – selezione non richiesta
Stampi a dsDNA, mutazioni quasi ovunque
Alta temperatura--> meno strutture secondarie
veloce -non richiede clonaggio in vettori particolari
Svantaggi potenziali:
•
Frequenza di errori elevata– si usa polimerasi alta fedeltà (e.g., VENT or
PFU)
•
Ridotta efficienza di applicazione per frammenti > 2-3 kb
Possibilità con la mutagenesi per PCR
• Sostituzione di base
• Overlap extension
• Megaprimer extension
• Quik change
• Delezioni
• Mismatched primers
• PCR-fusion
• Inverted PCR
• Inserzioni
• Overlap extension
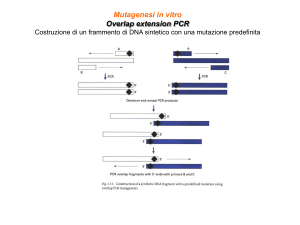
Overlap Extension PCR Mutagenesis
DNA
stampo
PCR con primers mutagenici
Purifica ed unisci
PCR con primers
terminali
DNA mutato
Megaprimer Extension PCR Mutagenesis
DNA
PCR con primer mutagenico
stampo
Purifica megaprimer
PCR con megaprimer+primer terminale
Mutated DNA
QuikChange® (80%)
• PCR
• Proofreading Polymerase
• Amplifica tutto il
plasmide
• DpnI degrada templato
wt metilato
• Non usare con DNA da
ceppi dam• Rapido
Deletion PCR Mutagenesis
Deletion PCR Mutagenesis
Insertion PCR Mutagenesis
Principali metodi di mutagenesi
• MUTAGENESI SITO-DIRETTA
• SCANNING MUTAGENESIS
• MUTAGENESI A SATURAZIONE/CASUALE
• MUTAGENESI PER RICOMBINAZIONE
Scanning Mutagenesis (1)
•
•
•
•
•
Cambia residui di superficie, conservando la struttura 3D
I residui carichi di superficie non sono di solito cruciali per la integrità
strutturale: importanti per legame, oligomeri e catalisi
ALANINE SCANNING MUTAGENESIS
Sostituzione sistematica di residui carichi con alanina:
•
•
•
Elimina la catena laterale dopo il carbonio beta
Elimina interazioni funzionali tra aminoacidi
Non altera la conformazione della proteina
Molto efficace per assegnare funzioni di superficie
Alanine scanning mutagenesis
gggcccttttaaagggaattaattcccccaaatttggggggtttaaatttgggcccccacacatacatatctaccta
DNA
HFKRVVSPWNACSPCYYTQDEED Protein
AFKRVVSPWNACSPCYYTQDEED
HAKRVVSPWNACSPCYYTQDEED
HFARVVSPWNACSPCYYTQDEED
HFKAVVSPWNACSPCYYTQDEED
HFKRAVSPWNACSPCYYTQDEED
HFKRVASPWNACSPCYYTQDEED
HFKRVVAPWNACSPCYYTQDEED
HFKRVVSAWNACSPCYYTQDEED
HFKRVVSPANACSPCYYTQDEED
Scanning Mutagenesis (2)
CYSTEINE SCANNING MUTAGENESIS
•
•
•
•
Residui di cisteina non appaiati sostituiscono specifici aminoacidi
Residui di cisteina: dimensioni medie, non carichi e idrofobici
Reagiscono bene con agenti chimici modificanti tipo N-ethylmaleimide
Si usa per;
•
•
Marcare la proteina per studi di topologia
Misurare la sensibilità a reagenti in acqua o lipidi
Principali metodi di mutagenesi
• MUTAGENESI SITO-DIRETTA
• SCANNING MUTAGENESIS
• MUTAGENESI A SATURAZIONE/CASUALE
• MUTAGENESI PER RICOMBINAZIONE
STRATEGIA GENERALE DI EVOLUZIONE DIRETTA
Complessità
Espressione
Identificazione
Fitness landscape (scenario del
successo evolutivo): possibilità
che si presentano ad una data
proteina in termini di fitness.
Per esplorare queste possibilità
la sequenza della proteina deve
variare all’interno di uno spazio
di sequenza (complessità della
diversità
dei
sistemi
proteici=20n )
Mutagenesi a Saturazione/Casuale
•
GENERA MUTAZIONI SU TUTTA LA SEQUENZA
•
•
Casuale: trascura le informazioni sul ruolo dei singoli aminoacidi
Fornisce informazioni su tutto lo “sequence space” – i.e., relazioni
tra sequenza aa, struttura 3D e funzione
•
Se utilizzata su piccoli frammenti della proteina
•
Si può costruire un catalogo di aminocidi permessi o deleteri
•MUTAGENESI CHIMICA/RADIAZIONI
•MUTAGENESI A CASSETTA
•MUTAGENESI PER INCORPORAZIONE ERRATA
•MUTAGENESI CON SET DI OLIGONUCLEOTIDI PARTICOLARI
MUTAGENESI CASUALE
CON OLIGO DEGENERATI
OLIGO CON ANALOGHI
DI BASI
5-BROMOURIDINA
2-AMINOPURINA
Error-prone PCR con Taq-polimerasi
• Nella PCR aumento
– concentrazione MgCl2
– # cicli
- concentrazione polimerasi
• Oppure uso:
- MnCl2
-dNTP sbilanciati
-dITP
Mutagenesi per ricombinazione
UTILIZZA LA VARIABILITA’ GIA’ PRESENTE IN REGIONI SEPARATE
DI UNA SEQUENZA O PIU’ SEQUENZE
• DNA shuffling:
frammentazione + PCR
• StEP (Staggered Extension Process):
estensione corta + PCR
• Random-priming:
primers casuali + PCR
DNA SHUFFLING SU GENI DA FAMIGLIE PROTEICHE
Incroci di molecole
DNA SHUFFLING.
DNAsi e PCR senza primers
DNAse I
5’
*
*
5’
5’
*
*
5’
Denature & anneal
DNAse I
5’
**
*
5’
fill in & amplify
5’
3’
*
*
*
*
3’
5’
DNA shuffling
(= Molecular Breeding)
Hetero-duplex formation
Homo-duplex formation
5’
3’
3’
5’
5’
PCR
5’
3’
5’
PCR
3’
5’
Resembles synthesis of
synthetic gene
(Directed Mutagenesis)
3’
3’
5’
3’
3’
5’
Resembles synthesis of
gene fusions
(Directed Mutagenesis)
StEP recombination
Staggered Extension Process
NB I geni devono presentare un grado
di omologia di sequenza elevato
StEP recombination. Only one
primer and single strands from two
parent genes (templates) are
shown. (A) Denatured template
genes are primed with one defined
primer. (B) Short fragments are
produced by brief Polymerasecatalyzed primer extension. (C)
Through another cycle of StEP,
fragments randomly prime the
templates (template switching)
and extend further. (D) This
process is repeated until full-length
genes are produced resulting in a
genepool of recombined parent
genes (E).
Staggered Extension Process (StEP)
Denaturation
Annealing, Extension
Denaturation
Annealing, Extension
SCOPE (Structure based Protein engineering)
Beta-lattamasi
SCHEMA (algoritmo per predire quali frammenti possono
essere ricombinati senza disturbare la struttura)
www.che.caltech.edu/groups/fha/code.html
ALCUNI KIT DI MUTAGENESI COMMERCIALI
Amersham-Pharmacia www.apbiotech.com
Sculptor IVM mutagenesis kit Biotech
USE mutagenesis kit
Bio-Rad
www.bio-rad.com
Muta-gene in vitro mutagenesis kit
Clontech
www.clontech.com
Transformer site-directed mutagenesis kit
Life Technologies
www.lifetech.com
CFLP Powerscan mutation detection system
PanVera
www.panvera.com
Mutan-Express Km kit
Promega
www.promega.com
Altered Sites II in vitro mutagenesis systems
Gene Editor System
Interchange in vitro mutagenesis system
Stratagene
www.stratagene.com Quik-change site-directed mutagenesis kit
ExSite PCR-based mutagenesis kit
Chameleon ds site-directed mutagenesis kit
NEB
www.neb.com
Code20 kit










![mutazioni genetiche [al DNA] effetti evolutivi [fetali] effetti tardivi](http://s1.studylibit.com/store/data/004205334_1-d8ada56ee9f5184276979f04a9a248a9-300x300.png)
